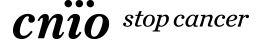- Todos
- A
- B
- C
- D
- E
- F
- G
- H
- I
- J
- K
- L
- M
- N
- Ñ
- O
- P
- Q
- R
- S
- T
- U
- V
- W
- X
- Y
- Z
A
Análisis de ligamiento - Linkage analysis (sinónimo: análisis indirecto del ADN)
Estudio de los polimorfismos de las secuencias de ADN (variantes normales) que están próximos a/o dentro de un gen de interés (ligamiento) para identificar, en una misma familia, la segregación de una mutación patológica en un gen determinado.
EXPLICACIÓN:
Las tres fases del análisis de ligamiento son las siguientes:
1. Establecer los haplotipos de cada individuo: Se analizan los múltiples marcadores de ADN dispuestos a cada lado (marcadores flanqueantes) o dentro (marcadores intragénicos) de la región del gen de interés, con objeto de determinar el conjunto de marcadores (haplotipos) de cada miembro de la familia.
2. Establecer la fase, comparando los haplotipos entre los miembros de la familia: Mediante la comparación de los haplotipos de los miembros de la familia con un estado genético conocido (es decir, afectados, no afectados), se puede identificar el haplotipo asociado con el alelo patológico.
3. Determinar el estado genético: Una vez establecido el haplotipo asociado con la enfermedad, es posible determinar el estado genético de los miembros de una familia de riesgo.
Ejemplo: Análisis de ligamiento para el diagnóstico prenatal de una enfermedad ligada al cromosoma X.
Algunas implicaciones clínicas
El análisis de ligamiento se utiliza generalmente cuando el análisis de ADN directo no es viable, porque se desconoce el gen de interés o no se puede detectar una mutación en ese gen en una familia específica.
En la mayoría de los casos, el haplotipo en sí carece de significación; sólo tiene sentido en el contexto de un estudio familiar. Sólo en el caso raro de que el desequilibrio del ligamiento sea un haplotipo específico, habrá una probabilidad muy elevada de que esté asociado con una mutación patológica específica.
La exactitud del análisis de ligamiento depende de:
La exactitud del diagnóstico clínico en un miembro o miembros de la familia que estén afectados.
La distancia entre la mutación patológica y los marcadores. El análisis de ligamiento puede proporcionar resultados falso positivo o falso negativo si, durante la formación de los gametos (meiosis), se produce recombinación que implique a los marcadores y suponga un intercambio entre el material genético de los cromosomas heredados de la madre o del padre. El riesgo de recombinación es proporcional a la distancia entre la mutación patológica y los marcadores. El riesgo de recombinación es más bajo si se utilizan marcadores intragénicos.
La informatividad de los marcadores genéticos en la familia del paciente. Si la secuencia de ADN de una variante determinada difiere entre los cromosomas heredados de la madre y del padre, este marcador es informativo. Por el contrario, si la secuencia de ADN de una variante determinada no difiere entre los dos cromosomas, el marcador no es informativo.
Análisis de microsatélites - Microsatellite analysis
Uso de secuencias repetitivas muy variables, que se encuentran en las regiones microsatélites adyacentes a los genes o intragénicas o en otras áreas de interés, como marcadores para el análisis de ligamiento, huellas moleculares de adn u otras aplicaciones diagnósticas.
Análisis directo del ADN - Direct DNA analysis
La utilización de técnicas de secuenciación, análisis mutacional, rastreo de mutaciones o cualquier otro medio de estudio molecular genético, para detectar variantes en un gen que pueden estar asociadas a trastornos genéticos específicos. El análisis directo del adn es posible sólo cuando se conoce el gen o genes o la región genómica, asociados con la enfermedad.
Las técnicas utilizadas para el análisis de ADN directo pueden ser las siguientes:
Análisis de secuencias
Rastreo de mutaciones (p. ej. CSGE, DHPLC, SSCP)
Análisis mutacional (p. ej. PCR, Southern blot, análisis de repeticiones de trinucleótidos, FISH, análisis de truncación de proteínas (PTT), estudios de heterocigosidad, análisis RFLP, ASO)
El análisis de ADN directo contrasta con el análisis de ligamiento (que anteriormente se denominaba análisis de ADN indirecto)
Algunas implicaciones clínicas
El análisis de ADN directo sólo es posible cuando se conoce el gen (genes) o la región genómica asociados con una enfermedad
El análisis de ligamiento se puede realizar cuando no se conoce el gen o los genes asociados con una enfermedad
B
C
Clonación posicional - Positional cloning (sinónimo: Genética inversa)
Clonación o identificación de un gen causante de una enfermedad específica, basándose en su localización en el genoma, determinada por diversos métodos, incluyendo análisis de ligamiento, mapeo genómico (físico) y bioinformática, cuando no se conoce la información sobre la base bioquímica de la enfermedad. Se distingue de la estrategia más común de clonación génica en que en ésta se parte de un producto protéico conocido, se determina su secuencia de aminoácidos y se utiliza esa información para aislar el gen.
Cosegregación - Cosegregation
Transmisión conjunta de dos o más genes ligados en un mismo cromosoma.
Ejemplo de cosegregación y recombinación: Pareja de cromosomas en meiosis que contienen los marcadores A, B y C en uno de los dos cromosomas homólogos y a, b y c en el otro. Recombinación entre los loci A y B (y a y b) que están muy separados; cosegregación de los loci B y C (y b y c) que están estrechamente ligados.
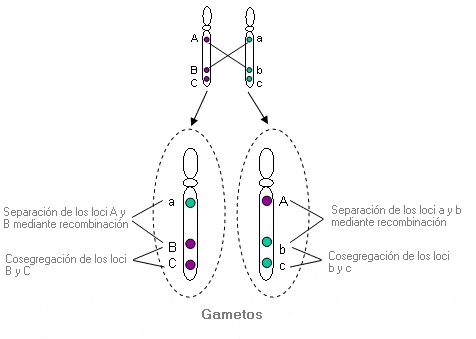
Debido a la gran distancia entre A y B (y entre a y b), la probabilidad de recombinación entre los dos loci es alta. Puesto que B y C (y b y c) están más juntos, la probabilidad de recombinación entre estos loci es baja; por tanto, B y C (y b y c) tienden a ser heredados juntos en el mismo cromosoma (es decir, cosegregan en la meiosis) con más frecuencia que A y B (y a y b).