- Todos
- A
- B
- C
- D
- E
- F
- G
- H
- I
- J
- K
- L
- M
- N
- Ñ
- O
- P
- Q
- R
- S
- T
- U
- V
- W
- X
- Y
- Z
A
Alelo - Allele
Un alelo es cada una de las dos o más versiones de un gen. Un individuo hereda dos alelos para cada gen, uno del padre y el otro de la madre. Los alelos se encuentran en la misma posición dentro de los cromosomas homólogos. Si los dos alelos son idénticos, el individuo es homocigoto para este gen.
Tipos de alelos denominados según su función: En la figura siguiente se muestran las proporciones aproximadas de los alelos en un locus genético hipotético, en una población determinada. Nota: Las mutaciones patológicas, los polimorfismos y las variantes alélicas de significación desconocida se consideran alelos “mutantes” en todos los casos.
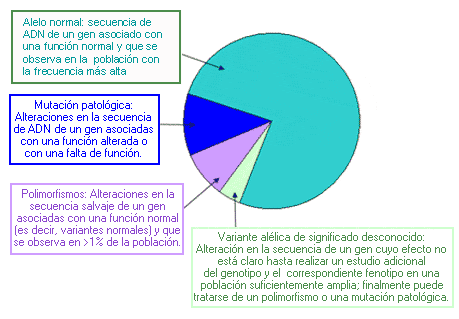
- Alelo materno: Heredado de la madre
- Alelo paterno: Heredado del padre
Los individuos pueden ser:
- Homocigóticos: cuando portan los alelos del gen iguales, cada uno procedente de un progenitor.
- Heterocigóticos: cuando los dos alelos del gen son diferentes.
Alteración de la secuencia - Sequence alteration: (sinónimo: mutación)
Cualquier cambio producido en el adn[o mas concretamente en un gen con respecto a su estado natural. Si no tiene repercusión clínica se denomina polimorfismo; cuando se trata de un cambio que tiene o puede tener repercusión clínica se suele denominar mutación. Cuando no se conoce si el cambio tiene o no repercusión clínica se habla de cambio o variante de significado desconocido.
Análisis de fragmentos de restricción de longitud polimórfica - Restriction fragment length polymorphism analysis (sinónimo: análisis RFLP, estudios RFLP)
Estudio de los fragmentos de ADN de tamaño previsible originados por la digestión (corte) de una cadena de ADN por una enzima de restricción específica. Las alteraciones (mutaciones o polimorfismos) producidas en la secuencia de ADN que destruyen o crean nuevos sitios de corte en el ADN, modifican el tamaño (y el número) de los fragmentos de ADN resultantes de la digestión por una enzima de restricción específica.

Análisis de ligamiento - Linkage analysis (sinónimo: análisis indirecto del ADN)
Estudio de los polimorfismos de las secuencias de ADN (variantes normales) que están próximos a/o dentro de un gen de interés (ligamiento) para identificar, en una misma familia, la segregación de una mutación patológica en un gen determinado.
EXPLICACIÓN:
Las tres fases del análisis de ligamiento son las siguientes:
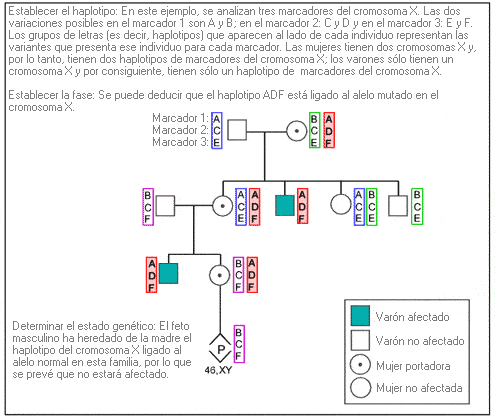
1. Establecer los haplotipos de cada individuo: Se analizan los múltiples marcadores de ADN dispuestos a cada lado (marcadores flanqueantes) o dentro (marcadores intragénicos) de la región del gen de interés, con objeto de determinar el conjunto de marcadores (haplotipos) de cada miembro de la familia.
2. Establecer la fase, comparando los haplotipos entre los miembros de la familia: Mediante la comparación de los haplotipos de los miembros de la familia con un estado genético conocido (es decir, afectados, no afectados), se puede identificar el haplotipo asociado con el alelo patológico.
3. Determinar el estado genético: Una vez establecido el haplotipo asociado con la enfermedad, es posible determinar el estado genético de los miembros de una familia de riesgo.
Ejemplo: Análisis de ligamiento para el diagnóstico prenatal de una enfermedad ligada al cromosoma X.
Algunas implicaciones clínicas
El análisis de ligamiento se utiliza generalmente cuando el análisis de ADN directo no es viable, porque se desconoce el gen de interés o no se puede detectar una mutación en ese gen en una familia específica.
En la mayoría de los casos, el haplotipo en sí carece de significación; sólo tiene sentido en el contexto de un estudio familiar. Sólo en el caso raro de que el desequilibrio del ligamiento sea un haplotipo específico, habrá una probabilidad muy elevada de que esté asociado con una mutación patológica específica.
La exactitud del análisis de ligamiento depende de:
La exactitud del diagnóstico clínico en un miembro o miembros de la familia que estén afectados.
La distancia entre la mutación patológica y los marcadores. El análisis de ligamiento puede proporcionar resultados falso positivo o falso negativo si, durante la formación de los gametos (meiosis), se produce recombinación que implique a los marcadores y suponga un intercambio entre el material genético de los cromosomas heredados de la madre o del padre. El riesgo de recombinación es proporcional a la distancia entre la mutación patológica y los marcadores. El riesgo de recombinación es más bajo si se utilizan marcadores intragénicos.
La informatividad de los marcadores genéticos en la familia del paciente. Si la secuencia de ADN de una variante determinada difiere entre los cromosomas heredados de la madre y del padre, este marcador es informativo. Por el contrario, si la secuencia de ADN de una variante determinada no difiere entre los dos cromosomas, el marcador no es informativo.
B
Bloque haplotípico - Haplotype Block
Región del genoma donde los diferentes polimorfismos genéticos como los SNPs son heredados en bloque.
C
D
E
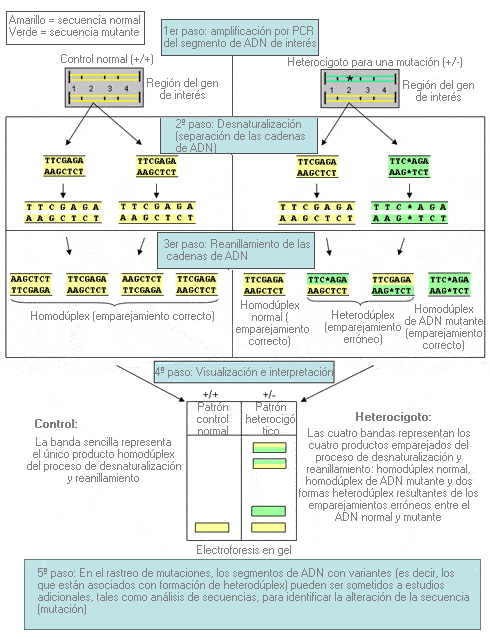
Electroforesis en gel sensible a conformación - Conformation-sensitive gel electrophoresis (sinónimo: CSGE)
Técnica electroforética que consiste en el análisis de un segmento de ADN para detectar errores de emparejamiento entre pares de bases normales y mutadas (ver explicación esquemática). Se emplea para el rastreo de mutaciones.
F
Frecuencia de portadores - Carrier rate
Proporción de individuos en una población que tienen una sola copia de una variante genética (mutación o polimorfismo).
Los individuos portadores de una sola mutación de un gen recesivo no se pueden identificar normalmente por su fenotipo (es decir, no están afectados); la frecuencia de portadores se puede calcular utilizando la ley de Hardy-Weinberg cuando se conoce la incidencia de la enfermedad (es decir, la frecuencia de homocigotos).

Cálculo de la tasa de portadores (2pq) para una enfermedad autosómica recesiva utilizando la ley de Hardy-Weinberg cuando se conoce la frecuencia de la enfermedad (q2):
Frecuencia de la enfermedad (q2)=1/2000; Puesto que p+q = 1; p =1-0,022 = 0,978.
Por lo tanto, 2pq = 2(0,978)(0,022) = 0,043 o aproximadamente 4%.
La tasa de portadores de esta enfermedad recesiva en particular es de aproximadamente un 4% o de 1 entre 25 individuos, dada una frecuencia o incidencia de la patología en esta población de 1 entre 2000.
Las tasas de portadores se pueden determinar de forma similar en enfermedades recesivas ligadas al cromosoma X.
G
H
I
J
K
L
M
Mutación - Mutation (sinónimos: alteración de la secuencia, mutación del splicing)
Cualquier alteración producida en un gen con respecto a su estado natural; puede ser patológica o una variante no patológica.
1. Mutaciones patológicas: aquellas que dan lugar a una función anormal del gen y a un fenotipo de enfermedad. Los tipos de mutaciones patológicas son los siguientes:


Adaptado de Thompson & Thompson Genetics in Medicine, 6th Edition; RL Nussbaum, RR McInnes, HF Willard, Genetic Variation in Individuals: Mutation and Polymorphism,pág 81, Copyright 2001, con permiso de Elsevier
2. Polimorfismos (variantes normales): Mutaciones que no tienen efectos adversos sobre la función del gen y no dan lugar a un fenotipo alterado (enfermedad).
N
Ñ
O
P
Polimorfismo - Polymorphism
Polimorfismo de Conformación de Cadena Simple o SSCP -
Polimorfismo de longitud de fragmentos de restricción - Restriction fragment length polymorphism (sinónimo: RFPL)
Variación natural (polimórfica) de una secuencia de ADN entre los individuos. Se visualiza porque se suprimen o se crean nuevos sitios de restricción (corte) en el ADN, que cuando es digerido por una endonucleasa da lugar a fragmentos de ADN de diferente longitud.
Polimorfismo de nucleótido único - Single Nucleotide Polymorphism o SNP
Variación en la secuencia de ADN que implica una sola base: adenina (A), timina (T), citosina (C) o guanina (G). Son los polimorfismos genéticos más frecuentes y se considera que determinan gran parte de la variabilidad genética entre individuos. Se conocen varios millones de SNP repartidos por todos los cromosomas humanos, aproximadamente 1 cada 300-100 pares de bases. La frecuencia de un SNP en población general se sitúa por encima del 1%.
Diversas técnicas de análisis masivo del genoma se fundamentan en el estudio de miles o millones de SNP que cubren la práctica totalidad del genoma (Genome Wide Association Studies, o GWAS), para buscar la asociación de algunos de ellos con enfermedades concretas, utilizando para ello grandes números tanto de individuos enfermos, como de individuos sanos.
Polimorfismos de conformación de cadenas sencillas - Single-stranded conformational polymorphism (SSCP)
Técnica para el rastreo de mutaciones que identifica segmentos de ADN de cadena sencilla que muestran un patrón de migración anormal en la electroforesis en gel.
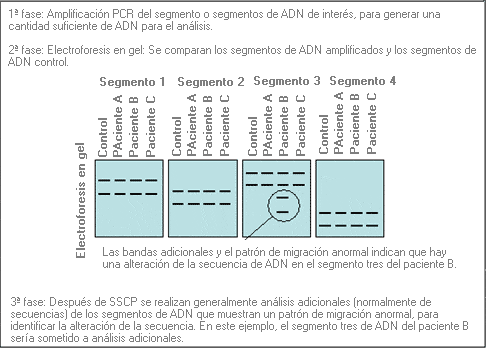
Algunas implicaciones clínicas
La técnica SSCP se puede utilizar cuando las mutaciones están distribuidas por todo el gen.
El SSCP puede comprender el análisis de todo el gen o sólo de regiones específicas.
Q
R
S
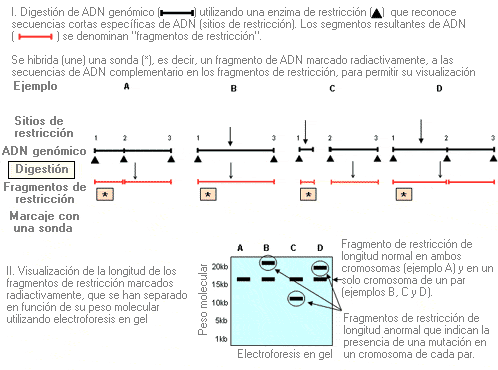
Southern blot - Southern blot: (sinónimos: Southern blotting, análisis de Southern blotting)
Técnica de análisis de genética molecular que se utiliza para detectar diferencias en la longitud de los fragmentos de ADN tras ser digeridos por enzimas de restricción (lo que se conoce normalmente como RFLP: polimorfismos de longitud de fragmentos de restricción). La longitud de los fragmentos de restricción es variable en tamaño, desde pocas bases a kilobases.
T
TagSNP - TagSNP
Es un SNP (Single Nucleotide Polymorphim) o polimorfismo de una base en una región del genoma en la que existe un alto desequilibrio de ligamiento, y que representa al conjunto de SNPs dentro ese bloque haplotípico. Con el análisis de los tagSNPs es posible inferir la totalidad de la variación genética e identificar asociaciones fenotípicas sin genotipar el resto de los SNPs en esta región cromosómica.