- Todos
- A
- B
- C
- D
- E
- F
- G
- H
- I
- J
- K
- L
- M
- N
- Ñ
- O
- P
- Q
- R
- S
- T
- U
- V
- W
- X
- Y
- Z
A
Alelo - Allele
Un alelo es cada una de las dos o más versiones de un gen. Un individuo hereda dos alelos para cada gen, uno del padre y el otro de la madre. Los alelos se encuentran en la misma posición dentro de los cromosomas homólogos. Si los dos alelos son idénticos, el individuo es homocigoto para este gen.
Tipos de alelos denominados según su función: En la figura siguiente se muestran las proporciones aproximadas de los alelos en un locus genético hipotético, en una población determinada. Nota: Las mutaciones patológicas, los polimorfismos y las variantes alélicas de significación desconocida se consideran alelos “mutantes” en todos los casos.
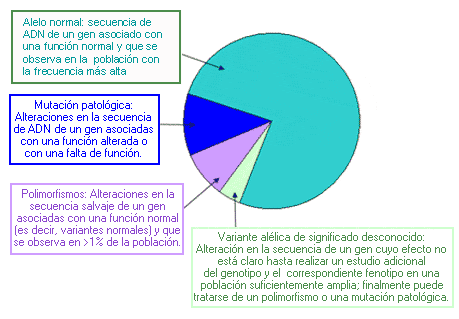
- Alelo materno: Heredado de la madre
- Alelo paterno: Heredado del padre
Los individuos pueden ser:
- Homocigóticos: cuando portan los alelos del gen iguales, cada uno procedente de un progenitor.
- Heterocigóticos: cuando los dos alelos del gen son diferentes.
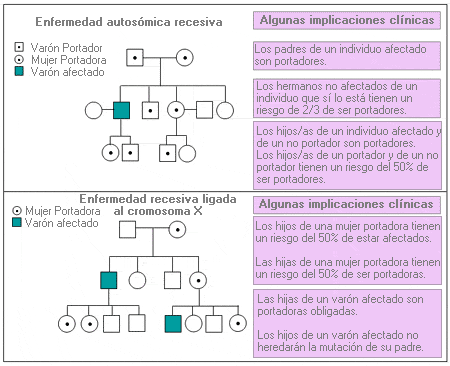
Autosómico dominante - Autosomal dominant
Término que se utiliza para describir un rasgo o patología asociados a un determinado alelo, que están presentes en todos los individuos que han heredado una sola copia de dicho alelo (heterocigotos). Se refiere específicamente a un gen de uno de los 22 pares de autosomas.
Arbol genealógico que representa el patrón de herencia autosómica dominante.
Probabilidad de que una mutación de un gen autosómico dominante sea transmitida a los descendientes.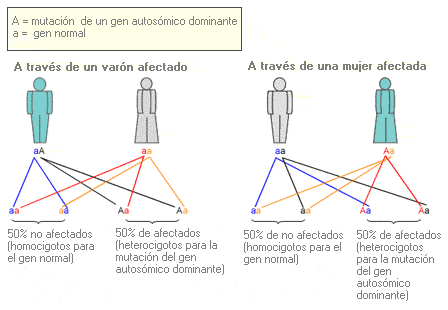
Autosómico recesivo - Autosomal recessive
Término que se utiliza para describir un rasgo o patología que requiere la presencia de las dos copias de un determinado alelo para que se exprese el fenotipo. Se refiere específicamente a los genes de uno de los 22 pares de autosomas.
Árbol genealógico que representa el patrón de herencia autosómica recesiva.
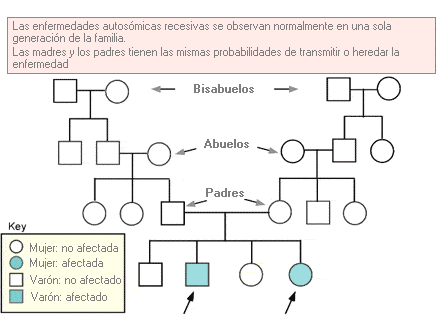
Probabilidad de que una mutación de un gen autosómico recesivo sea transmitida a los descendientes.
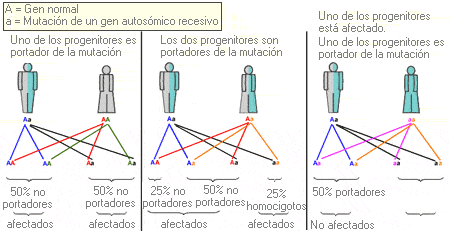
B
C
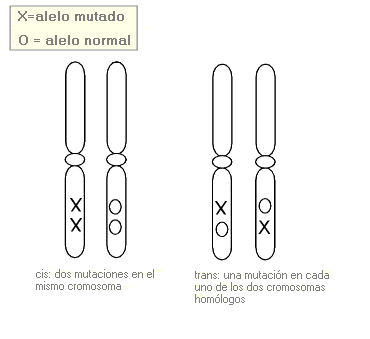
Configuración en cis - Cis configuration (sinónimos: cis, acoplamiento)
Término que indica que un individuo que es heterocigoto en dos loci adyacentes presenta esas dos mutaciones en el mismo cromosoma, en vez de una en cada cromosoma homólogo (configuración en trans).
D
E
Estudios de portadores - Carrier testing (sinónimos: detección de portadores, estudios de heterocigotos)
Pruebas que se realizan para identificar a los individuos asintomáticos que presentan una mutación en un gen causante de una enfermedad autosómica recesiva o recesiva ligada al cromosoma X.
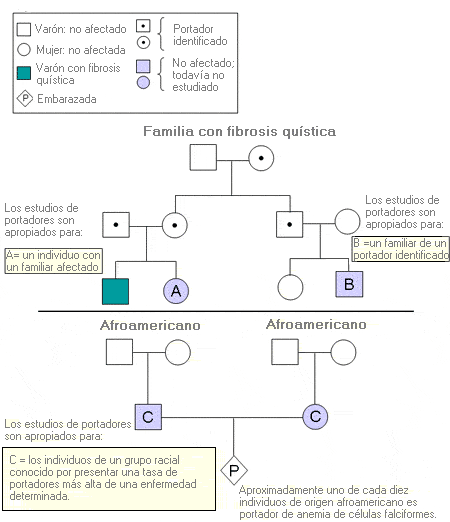 Los estudios de portadores se proponen a los individuos (A) con familiares afectados por una enfermedad genética; (B) con familiares que son portadores identificados y (C) que pertenecen a grupos étnicos o raciales conocidos por presentar una tasa de portadores más alta para una patología particular.
Los estudios de portadores se proponen a los individuos (A) con familiares afectados por una enfermedad genética; (B) con familiares que son portadores identificados y (C) que pertenecen a grupos étnicos o raciales conocidos por presentar una tasa de portadores más alta para una patología particular.
F
Frecuencia de portadores - Carrier rate
Proporción de individuos en una población que tienen una sola copia de una variante genética (mutación o polimorfismo).
Los individuos portadores de una sola mutación de un gen recesivo no se pueden identificar normalmente por su fenotipo (es decir, no están afectados); la frecuencia de portadores se puede calcular utilizando la ley de Hardy-Weinberg cuando se conoce la incidencia de la enfermedad (es decir, la frecuencia de homocigotos).

Cálculo de la tasa de portadores (2pq) para una enfermedad autosómica recesiva utilizando la ley de Hardy-Weinberg cuando se conoce la frecuencia de la enfermedad (q2):
Frecuencia de la enfermedad (q2)=1/2000; Puesto que p+q = 1; p =1-0,022 = 0,978.
Por lo tanto, 2pq = 2(0,978)(0,022) = 0,043 o aproximadamente 4%.
La tasa de portadores de esta enfermedad recesiva en particular es de aproximadamente un 4% o de 1 entre 25 individuos, dada una frecuencia o incidencia de la patología en esta población de 1 entre 2000.
Las tasas de portadores se pueden determinar de forma similar en enfermedades recesivas ligadas al cromosoma X.
G
H
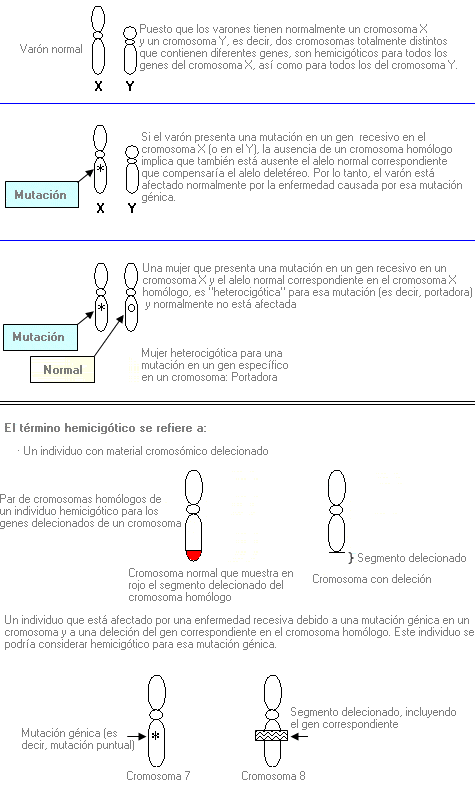
Hemicigoto - Hemizygous
Heterocigoto - Heterozygote
Individuo que tiene dos alelos diferentes en un locus, uno en cada cromosoma. En el caso de una mutación patológica, un alelo es normal y otro anormal.
Heterocigoto compuesto: Individuo que tiene dos alelos anormales o mutados diferentes en un mismo locus, uno en cada cromosoma; se refiere normalmente a los individuos afectados por una enfermedad autosómica recesiva.
Heterocigoto doble: Individuo que presenta dos mutaciones génicas diferentes (es decir, es heterocigótico) en dos loci genéticos distintos.
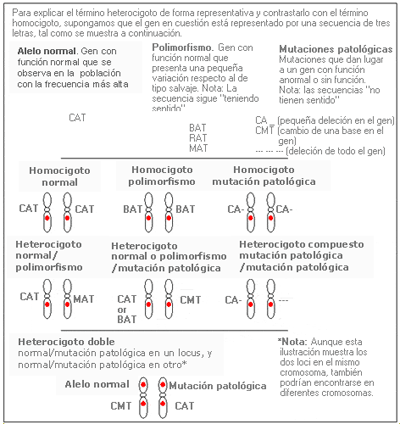
Heterocigoto compuesto - Compound heterozygote
Individuo que tiene dos alelos anormales o mutados diferentes en un mismo locus, uno en cada cromosoma; normalmente se refiere a los individuos afectados por una enfermedad autosómica recesiva.
Los hijos/as afectados de padres portadores de una mutación diferente en el mismo locus del gen son heterocigotos compuestos.
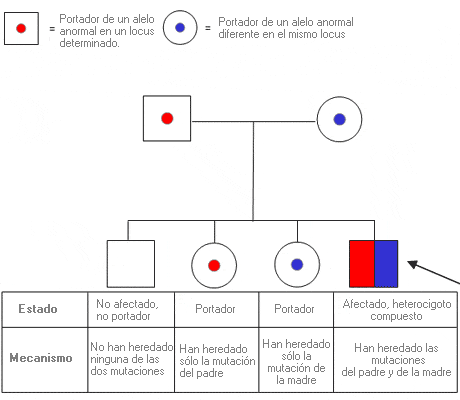
Heterocigoto doble - Double heterozygote
Individuo que es heterocigoto para mutación en dos loci genéticos diferentes. Es decir, es portador de dos mutaciones en genes distintos.
Cuando el padre y la madre presentan una mutación en dos loci genéticos diferentes, sus hijos pueden ser heterocigotos dobles. Los loci pueden estar en el mismo cromosoma o en diferentes cromosomas, según se muestra a continuación.
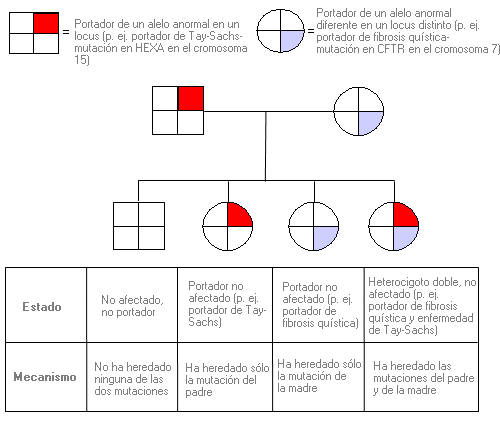
Nota: Puesto que en el ejemplo anterior se incluyen dos loci diferentes (dos alelos para cada uno), en cada hijo/a pueden darse 16 combinaciones posibles de los cuatro alelos de los padres.
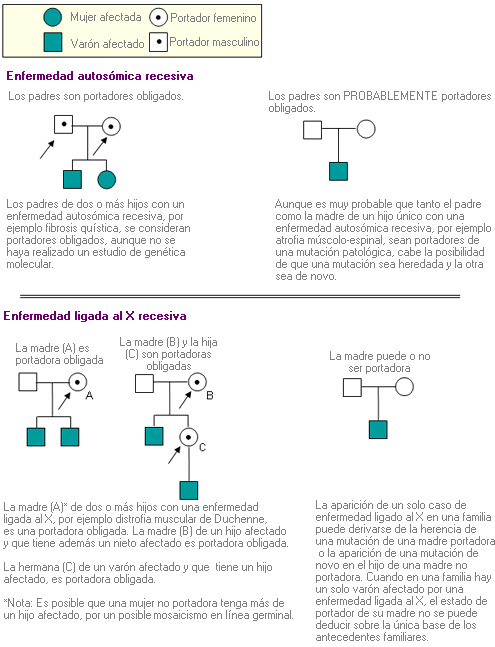
Heterocigoto obligado - Obligate heterozygote (sinónimo: portador obligado)
Individuo que de acuerdo al análisis de los antecedentes familiares debe ser portador de una mutación genética, aunque clínicamente pueda no estar afectado.
Heterosis - Heterosis
Aquella situación en la que el heterocigoto presenta superioridad respecto a los homocigotos para un carácter determinado.
I
J
K
L
M
N
Ñ
O
P
PCR cuantitativa - Quantitative PCR (sinónimos: PCR cuantitativa en tiempo real)
Uso de PCR para determinar la cantidad de ADN o ARN en una muestra; se utiliza frecuentemente para detectar mutaciones por deleción y mutaciones por duplicación.
Anteriormente, la PCR cuantitativa determinaba la cantidad de producto (ADN o ARN) tras completar un número específico de ciclos; sin embargo, este procedimiento no es del todo fiable o preciso y se ha sustituido en gran parte por la PCR cuantitativa “en tiempo real” (denominada también “cinética”), en la que se determina la cantidad del producto (ADN o ARN) tras completar cada ciclo de PCR.
PCR cuantitativa en tiempo real
I. Se amplifica la región de ADN o ARN de interés en presencia de una sonda fluorescente; la intensidad de la señal fluorescente generada aumenta en proporción a la cantidad de producto resultante en PCR.
II. La intensidad de la señal fluorescente, que se mide al completar cada ciclo de PCR, se representa en una “gráfica de amplificación” para determinar el CT (del inglés cycle threshold), que se define como el punto en el que se detecta por primera vez la señal fluorescente, es decir, el producto de la PCR.
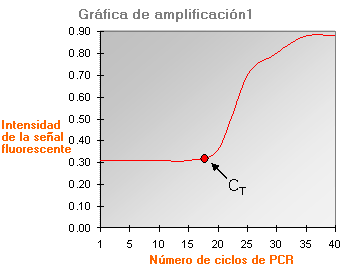
III. Se compara el valor CT frente a una curva estándar (basándose en los valores CT de muestras control y sus respectivas cantidades iniciales de ADN o ARN conocidas) para determinar la cantidad de ADN o ARN en la muestra del paciente. La presencia de una mutación por deleción o duplicación se puede deducir comparando la cantidad de ADN o ARN en la muestra del paciente con la cantidad en una muestra control. Por ejemplo, si la cantidad de ADN o ARN en la muestra del paciente fuese ~50% respecto a la del control normal, esto indicaría una mutación por deleción; si la cantidad de ADN o ARN en la muestra fuese ~150% respecto a la del control normal, indicaría una mutación por duplicación.
1Adaptado de PennState College of Medicine, Quantitative Real-Time PCR
Algunas implicaciones clínicas
Al igual que la PCR, la PCR cuantitativa se ha convertido en una técnica estándar en todos los laboratorios clínicos de genética molecular.
Los análisis para detectar mutaciones por deleción hemicigótica (p. ej. deleciones en DMD en varones afectados por distrofia muscular de Duchenne) se pueden realizar con técnicas de PCR estándar. Sin embargo, se debe realizar PCR cuantitativa, en lugar de estándar, para detectar mutaciones por deleción heterocigótica (p. ej. deleciones heterocigóticas en (1) individuos afectados por un trastorno causado por una deleción en un gen contiguo autosómico, como, por ejemplo, el síndrome de Williams, (2) portadores de un trastorno autosómico recesivo, como la atrofia muscular espinal y (3) mujeres portadoras de un trastorno ligado al cromosoma X, por ejemplo la distrofia muscular de Duchenne).
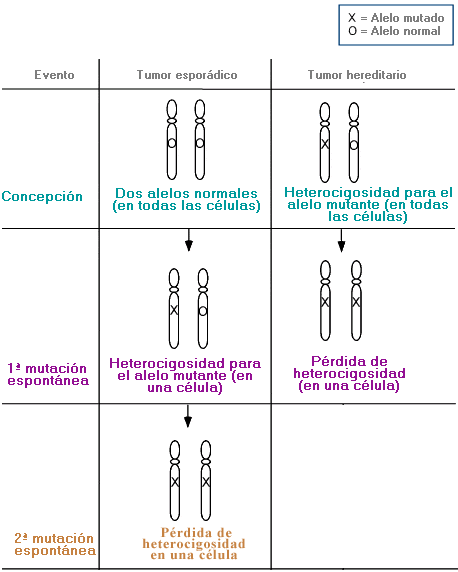
Pérdida de heterocigosidad - Loss of heterozygosity (LOH, de sus siglas en inglés)
Cuando en un locus determinado, que es heterocigoto para un alelo mutante deletéreo y un alelo normal, se produce una deleción, u otro evento mutacional, en el alelo normal que convierte a la célula bien en hemicigótica (es decir, con un único alelo deletéreo), bien en homocigótica para el alelo deletéreo.
La LOH es el mecanismo en el que se basa el desarrollo de algunos tipos de cáncer en los que una sola mutación en uno de los homólogos de un gen supresor tumoral no es suficiente para iniciar el crecimiento del tumor. Sin embargo, la deleción o inactivación del segundo alelo en el cromosoma homólogo, da lugar a un crecimiento celular incontrolado.
La LOH puede ser el fundamento del desarrollo del cáncer en tumores esporádicos y hereditarios. En los tumores esporádicos, los dos alelos son normales en el momento de la concepción, mientras que en los tumores hereditarios hay un alelo mutante en el momento de la concepción.
Portador - Carrier
Portador obligado - Obligate carrier (sinónimo: heterocigoto obligado)
Portador sintomático - Manifesting carrier
Individuo que en un locus determinado presenta un alelo recesivo patológico en un cromosoma y un alelo normal en el otro cromosoma y que manifiesta algunos síntomas de la enfermedad. Este término se usa con frecuencia para portadoras de una mutación recesiva ligada al cromosoma X que manifiestan signos de la enfermedad, aunque su fenotipo suele ser menos severo que el de los varones con la misma mutación.
Aunque con algunas excepciones, los portadores de una mutación patológica recesiva por regla general no manifiestan síntomas clínicos de la enfermedad. Sin embargo, los portadores de mutaciones recesivas ligadas al cromosoma X presentan ocasionalmente algunas manifestaciones clínicas de la enfermedad, por lo que se les denomina portadores sintomáticos. Se supone que el mecanismo subyacente en los portadores sintomáticos es una inactivación sesgada o no aleatoria del cromosoma X, de tal manera que en la mayoría de las células el cromosoma X activo es el que tiene la mutación.
Ejemplos de enfermedades ligadas al cromosoma X en las que son frecuentes las portadoras sintomáticas:
Hemofilia A: Tiempo de coagulación prolongado, hemorragia excesiva durante intervenciones quirúrgicas menores y menstruaciones abundantes.
Distrofia muscular de Duchenne: Debilidad muscular, hipertrofia de pantorrillas leve y/o miocardiopatía dilatada.
Síndrome X frágil (mutación con penetrancia completa): Retraso mental.