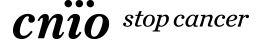- Todos
- A
- B
- C
- D
- E
- F
- G
- H
- I
- J
- K
- L
- M
- N
- Ñ
- O
- P
- Q
- R
- S
- T
- U
- V
- W
- X
- Y
- Z
A
Amplicón - Amplicon
Fragmento de ADN amplificado por la reacción en cadena de la polimerasa (PCR) o cualquier otro proceso que dé lugar a la producción de diferentes copias de ese fragmento.
Análisis de secuencias - Sequence analysis (sinónimo: secuenciación génica, secuenciación)
Proceso mediante el cual se determina la secuencia de nucleótidos en un segmento de ADN.
Se puede secuenciar un gen completo, pero la mayoría de las veces se secuencian sólo regiones específicas del gen que muy probablemente contendrán mutaciones (p. ej. los exones y los límites entre los intrones y los exones).
Al secuenciar un segmento de ADN, se identifican la mayoría de las variaciones producidas con respecto a la secuencia normal (por el contrario, el análisis mutacional identifica sólo mutaciones específicas en un segmento determinado de ADN).
Tipos de alteraciones de las secuencias que se pueden detectar:
Alteraciones de secuencia descritas en la literatura como patógenas
Alteraciones de secuencias previsiblemente patógenas, pero no descritas en la literatura
Alteraciones de secuencias de significación clínica imprevisible
Alteraciones de secuencias previsiblemente benignas, pero no descritas en la literatura
Alteraciones de secuencias benignas descritas en la literatura
Posibilidades en caso de que no se detecten alteraciones de las secuencias:
El paciente no presenta una mutación en el gen analizado (p. ej. hay una alteración de la secuencia en otro gen, en un locus diferente)
El paciente presenta una alteración de la secuencia que no se puede detectar mediante el análisis de secuencias (p. ej. deleción de tamaño grande)
El paciente presenta una alteración de la secuencia en una región del gen (p. ej. una región intrónica o reguladora) que no se ha detectado en el análisis de laboratorio
1Adaptado de ACGM Recommendations for Standards for Interpretation of Sequence Variations (2000)
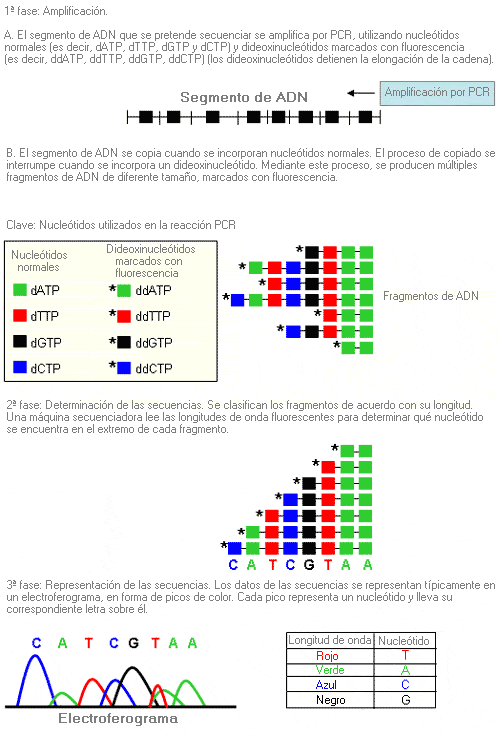
Análisis de truncación de proteínas - Protein truncation testing (sinónimo: PTT)
Método utilizado para identificar las proteínas que se acortan (truncan) a consecuencia de mutaciones que causan específicamente la terminación prematura del proceso de traducción del ARNm.
B
C
Cebador - Primer
Una secuencia corta de oligonucleótidos que se utiliza en la reacción en cadena de la polimerasa (PCR) para la creación de moléculas más largas de ADN.
D
E
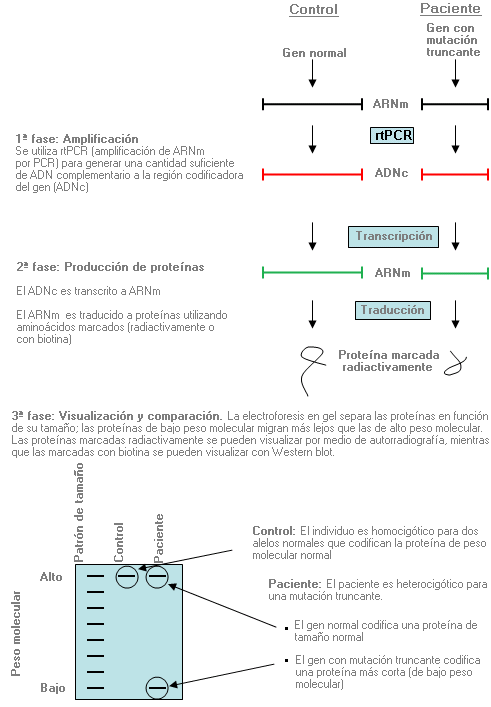
Estudios de repeticiones de trinucleótidos - Trinucleotide repeat testing
Análisis genético cuyo objetivo es la cuantificación del número de repeticiones de grupos de tres nucleótidos en un segmento determinado de ADN.
Para cuantificar las repeticiones de trinucleótidos, se pueden utilizar dos tipos de análisis mutacional que se representan en el diagrama siguiente:
Algunas implicaciones clínicas:
El análisis Southern blot no se realiza de forma rutinaria en todos los laboratorios para clarificar la interpretación de los resultados de las bandas no concluyentes por PCR. Cuando se reciba un informe de un número de repeticiones “homocigóticas” normales, particularmente en un niño sintomático de corta edad que podría presentar una expansión grande, es recomendable que el médico confirme que se ha realizado el análisis Southern blot para evaluar la posibilidad de que haya una repetición de trinucleótidos muy expandida.
Si no está disponible el análisis Southern blot, se puede realizar un análisis mutacional basado en PCR/electroforesis en gel de los padres de un paciente con un número de repeticiones “homocigóticas” normales, para determinar si el paciente podría haber heredado de sus padres dos alelos con el mismo número de repeticiones.
F
G
H
I
J
K
L
M
N
Ñ
O
P
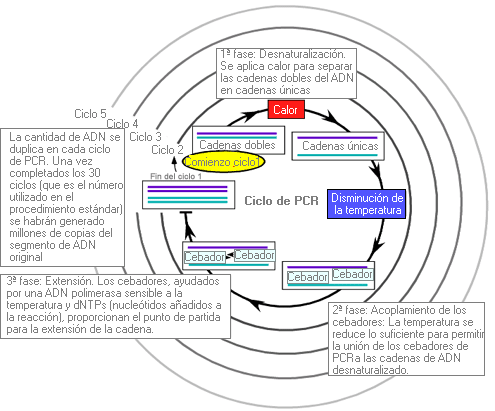
PCR - PCR (Polymerase chain reaction) (sinónimo: reacción en cadena de la polimerasa)
Procedimiento que genera millones de copias de un segmento corto de ADN mediante ciclos repetidos de: 1) desnaturalización del ADN, 2) acoplamiento de los oligonucleótidos y 3) extensión mediante la acción de la ADN polimerasa. La PCR es un procedimiento muy común en los estudios de genética molecular y se puede utilizar para: 1) generar una cantidad suficiente de ADN para realizar determinadas pruebas (p. ej. análisis de la secuencia, rastreo de mutaciones); o 2) puede ser una prueba diagnóstica en sí misma (por ejemplo amplificación específica del alelo, cuantificación del número de repeticiones de trinucleótidos).
PCR cuantitativa - Quantitative PCR (sinónimos: PCR cuantitativa en tiempo real)
Uso de PCR para determinar la cantidad de ADN o ARN en una muestra; se utiliza frecuentemente para detectar mutaciones por deleción y mutaciones por duplicación.
Anteriormente, la PCR cuantitativa determinaba la cantidad de producto (ADN o ARN) tras completar un número específico de ciclos; sin embargo, este procedimiento no es del todo fiable o preciso y se ha sustituido en gran parte por la PCR cuantitativa “en tiempo real” (denominada también “cinética”), en la que se determina la cantidad del producto (ADN o ARN) tras completar cada ciclo de PCR.
PCR cuantitativa en tiempo real
I. Se amplifica la región de ADN o ARN de interés en presencia de una sonda fluorescente; la intensidad de la señal fluorescente generada aumenta en proporción a la cantidad de producto resultante en PCR.
II. La intensidad de la señal fluorescente, que se mide al completar cada ciclo de PCR, se representa en una “gráfica de amplificación” para determinar el CT (del inglés cycle threshold), que se define como el punto en el que se detecta por primera vez la señal fluorescente, es decir, el producto de la PCR.
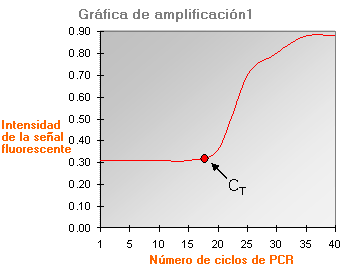
III. Se compara el valor CT frente a una curva estándar (basándose en los valores CT de muestras control y sus respectivas cantidades iniciales de ADN o ARN conocidas) para determinar la cantidad de ADN o ARN en la muestra del paciente. La presencia de una mutación por deleción o duplicación se puede deducir comparando la cantidad de ADN o ARN en la muestra del paciente con la cantidad en una muestra control. Por ejemplo, si la cantidad de ADN o ARN en la muestra del paciente fuese ~50% respecto a la del control normal, esto indicaría una mutación por deleción; si la cantidad de ADN o ARN en la muestra fuese ~150% respecto a la del control normal, indicaría una mutación por duplicación.
1Adaptado de PennState College of Medicine, Quantitative Real-Time PCR
Algunas implicaciones clínicas
Al igual que la PCR, la PCR cuantitativa se ha convertido en una técnica estándar en todos los laboratorios clínicos de genética molecular.
Los análisis para detectar mutaciones por deleción hemicigótica (p. ej. deleciones en DMD en varones afectados por distrofia muscular de Duchenne) se pueden realizar con técnicas de PCR estándar. Sin embargo, se debe realizar PCR cuantitativa, en lugar de estándar, para detectar mutaciones por deleción heterocigótica (p. ej. deleciones heterocigóticas en (1) individuos afectados por un trastorno causado por una deleción en un gen contiguo autosómico, como, por ejemplo, el síndrome de Williams, (2) portadores de un trastorno autosómico recesivo, como la atrofia muscular espinal y (3) mujeres portadoras de un trastorno ligado al cromosoma X, por ejemplo la distrofia muscular de Duchenne).
Polimorfismos de conformación de cadenas sencillas - Single-stranded conformational polymorphism (SSCP)
Técnica para el rastreo de mutaciones que identifica segmentos de ADN de cadena sencilla que muestran un patrón de migración anormal en la electroforesis en gel.
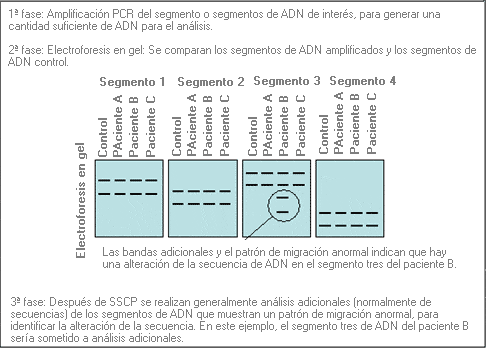
Algunas implicaciones clínicas
La técnica SSCP se puede utilizar cuando las mutaciones están distribuidas por todo el gen.
El SSCP puede comprender el análisis de todo el gen o sólo de regiones específicas.
Q
R
Reacción en cadena de la polimerasa - Polymerase chain reaction (PCR)
Véase PCR.
S
T
Termociclador - Thermocycler
También conocido como máquina de PCR es un instrumento usado en biología molecular que permite realizar de forma automatizada y secuencial los ciclos de temperaturas necesarios para llevar a cabo la reacción en cadena de la polimerasa (PCR) de amplificación de ADN.