- Todos
- A
- B
- C
- D
- E
- F
- G
- H
- I
- J
- K
- L
- M
- N
- Ñ
- O
- P
- Q
- R
- S
- T
- U
- V
- W
- X
- Y
- Z
A
Alu, Secuencias repetidas Alu - Alu, Alu element
Secuencias de ADN repetidas distribuidas a lo largo del genoma. Son las secuencias repetidas más frecuentes, representando el 5% del genoma humano. Su nombre se debe a que son reconocidas y cortadas por la enzima de restricción Alu.
Análisis de matrices de ADN - DNA microarray analysis
ARN (ácido ribonucleico) - RNA (ribonucleic acid)
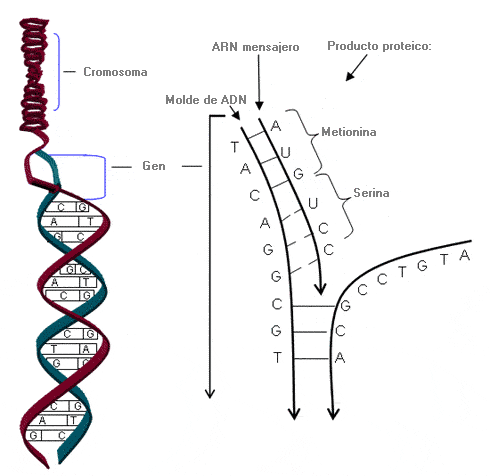
El ácido ribonucleico (ARN o RNA) es un ácido nucleico formado por una cadena de ribonucleótidos. Está formado por una cadena simple de ribonucleótidos, cada uno de ellos formado por ribosa, un fosfato y una de las cuatro bases nitrogenadas (adenina, guanina, citosina y uracilo). El ARN celular es lineal y monocatenario (de una sola cadena), pero en el genoma de algunos virus es de doble hebra.
Atlas del Genoma del Cáncer - The Cancer Genome Atlas TCGA
Proyecto internacional iniciado en el año 2005 con el objetivo de catalogar los cambios moleculares, a nivel de ADN, ARN, proteico y epigenético, existentes en los diferentes tipos de tumores mediante plataformas de análisis masivo y potentes análisis bioinformáticos. Con este proyecto se pretende mejorar el diagnóstico y el tratamiento de los pacientes oncológicos además de proporcionar información para prevenir y entender cuáles son los mecanismos moleculares asociados al desarrollo de esta enfermedad (https://cancergenome.nih.gov/)
B
BAM (Binary Aligned Mapped) -
Formato binario para el almacenamiento de datos de secuenciación. La extensión de archivo (.bam) contiene información sobre lecturas de secuencias después de haber sido estas alineadas contra un genoma de referencia. Muy utilizado para almacenar datos de secuenciación masiva.
Bloque haplotípico - Haplotype Block
Región del genoma donde los diferentes polimorfismos genéticos como los SNPs son heredados en bloque.
C
Célula - Cell
La unidad básica de un organismo vivo. Todas las eucariotas (con núcleo diferenciado), excepto las germinales, contienen una copia completa del genoma del organismo al que pertenecen.
Clonación posicional - Positional cloning (sinónimo: Genética inversa)
Clonación o identificación de un gen causante de una enfermedad específica, basándose en su localización en el genoma, determinada por diversos métodos, incluyendo análisis de ligamiento, mapeo genómico (físico) y bioinformática, cuando no se conoce la información sobre la base bioquímica de la enfermedad. Se distingue de la estrategia más común de clonación génica en que en ésta se parte de un producto protéico conocido, se determina su secuencia de aminoácidos y se utiliza esa información para aislar el gen.
Cobertura - Coverage
Número de veces que una porción del genoma es secuenciada en una reacción de secuenciación. Frecuentemente expresado como profundidad de lectura o “depth coverage” y numéricamente como 1X, 2X, 50X, 100X, etc.
D
E
Expresión génica - Gene expression
Proceso mediante el cual el ARN y las proteínas se elaboran a partir de las instrucciones codificadas en los genes. La expresión génica está controlada por proteínas y moléculas de ARN que se unen al genoma o al ARN y regulan sus niveles de producción y los de sus productos. Las alteraciones en la expresión génica cambian las funciones de las células, tejidos, órganos u organismos completos.
F
G
Gen - Gene
Genoma - Genome
Conjunto de los genes de un individuo o una especie contenida en un juego haploide de cromosomas.
Genómica - Genomics
La genómica se encarga del estudio de los genomas. Se considera a un genoma como el conjunto de información genética (ADN) de un organismo. A diferencia de la genética clásica, en la que a partir de un fenotipo se busca el o los genes responsables de dicho fenotipo, la genómica tiene como objetivo predecir la función de los genes a partir de su secuencia. Para conseguir este tipo de objetivos emplea herramientas basadas en análisis genéticos masivos (del orden de miles de genes) frecuentemente de forma simultánea (en un reducido número de experimentos).
Guía RNA - Guide RNA
Segmentos cortos de ARN utilizados para dirigir la enzima de corte de ADN a la ubicación de destino en el genoma. Los segmentos de gARN contienen la región de homología con la secuencia diana (normalmente 20 bases) y una secuencia que interacciona con la nucleasa (por ejemplo, Cas9). Los gARN utilizados en la edición del genoma son sintéticos y no ocurren en la naturaleza.
H
Haplotipo - Haplotype
Conjunto de alelos contenidos en un locus (o en varios loci) de una misma dotación haploide. El haplotipo podemos referirlo a un solo locus o a un genoma completo pero siempre se refiere a uno de los dos alelos de cada gen.
Herencia mitocondrial - Mitochondrial inheritance
Las mitocondrias son organelas citoplasmáticas que producen ATP (la energía que origina la mayoría de las reacciones químicas en el organismo) y contienen su propio genoma. Las mutaciones producidas en los genes mitocondriales son responsables de diversos síndromes reconocidos y se heredan siempre de la madre, puesto que los óvulos contienen mitocondrias, mientras que los espermatozoides no.
I
J
K
L
L1 -
Familia de elementos nucleares largos dispersos (LINE), presente en el genoma humano en número moderado de copias (alrededor de 80.000). Formada por unidades de unas 6 kb que pueden comportarse como retrotransposones.
M
Microsatélite - Microsatellite (sinónimos: ADN satélite, repeticiones cortas en tándem)
Segmentos repetitivos de ADN que comprenden de dos a cinco nucleótidos (repeticiones de dinucleótidos/trinucleótidos/ tetranucleótidos/ pentanucleótidos) dispersos por todo el genoma en las regiones no codificadoras que hay entre o dentro de los genes (intrones), se utilizan frecuentemente como marcadores en el análisis de ligamiento debido a la alta variabilidad natural existente en el número de repeticiones entre los individuos. Estas regiones son propiamente inestables y susceptibles a mutaciones.
N
Ñ
O
P
Pirrolisina - Pyrrolysine
La pirrolisina (abreviaturas Pyl o O) es un aminoácido natural, derivado de la lisina, codificado en el genoma de algunas arqueas metanógenas y una especie de bacteria (Desulfitobacterium hafniense). Forma parte de un código genético inusual, propio de estos organismos, y se considera el aminoácido proteinogénico número 22. Se encuentra codificada en el ARNm por el codón UAG.
Polimorfismo de nucleótido único - Single Nucleotide Polymorphism o SNP
Variación en la secuencia de ADN que implica una sola base: adenina (A), timina (T), citosina (C) o guanina (G). Son los polimorfismos genéticos más frecuentes y se considera que determinan gran parte de la variabilidad genética entre individuos. Se conocen varios millones de SNP repartidos por todos los cromosomas humanos, aproximadamente 1 cada 300-100 pares de bases. La frecuencia de un SNP en población general se sitúa por encima del 1%.
Diversas técnicas de análisis masivo del genoma se fundamentan en el estudio de miles o millones de SNP que cubren la práctica totalidad del genoma (Genome Wide Association Studies, o GWAS), para buscar la asociación de algunos de ellos con enfermedades concretas, utilizando para ello grandes números tanto de individuos enfermos, como de individuos sanos.
Proyecto Genoma Humano - Human Genome Project
El Proyecto Genoma Humano fue una investigación internacional que descifró la totalidad de la secuencia del genoma humano, mapeando y secuenciando todos sus genes. Oficialmente se inició en 1990 como un programa a quince años vista, con el que se pretendía registrar los 80.000-100.000 genes humanos (finalmente se comprobó que eran alrededor de 20.000-25.000). Los rápidos avances tecnológicos aceleraron los tiempos de modo que los resultados iniciales se publicaron en febrero de 2001, y la secuenciación se completó en el 2003, aunque no se conocen en detalle las funciones de todos los elementos del genoma.
Los objetivos concretos del Proyecto fueron:
• Identificar los aproximadamente 100.000 genes humanos en el ADN.
• Determinar la secuencia de 3 billones de bases químicas que conforman el ADN.
• Guardar la información en bases de datos.
• Desarrollar de modo rápido y eficiente tecnologías de secuenciación.
• Desarrollar herramientas para análisis de datos.
• Dirimir las cuestiones éticas, legales y sociales que se derivan del proyecto.
Q
R
Replicación del ADN - DNA replication
La replicación del ADN es el proceso mediante el cual se duplica una molécula (sintetiza una copia idéntica) de ADN. Cuando una célula se divide, en primer lugar, debe duplicar su genoma para que cada célula hija contenga un juego completo de cromosomas. De esta manera se garantiza la transmisión de la información genética.
S
Secuenciación del ADN - DNA sequencing
Es el conjunto de métodos que tienen por objetivo determinar el orden exacto de los nucleótidos (A, C, G y T) en la molécula de ADN. La secuencia de bases de ADN lleva la información que una célula necesita para originar moléculas de ARN y proteínas. Los avances en las técnicas de secuenciación de ADN han sido muy notables en los años recientes gracias al Proyecto Genoma Humano. Es posible aplicar estas técnicas para conocer la secuencia de un único gen, de varios genes (paneles), al conjunto de genes de un individuo (exoma) o, finalmente a todo el genoma de un individuo.
El método de secuenciación del ADN desarrollado por Frederick Sanger y colaboradores en 1977 ha sido el de uso más extendido en los últimos 40 años. En los años más recientes ese método está siendo sustituido por la secuenciación de nueva generación (“next-generation sequencing”, NGS, o los términos sinónimos “massively parallel” o “deep sequencing”) que ha revolucionado el mundo de la investigación biomédica al ser capaz de analizar simultáneamente millones de pequeños fragmentos de ADN. La tecnología de NGS permite disponer en horas de la secuencia del exoma o del genoma humano completo.
SNP - Single Nucleotide Polimorphism
Variación en la secuencia del adn (con respecto a una secuencia consenso) que afecta a una única base (A,T,G,C) en posiciones concretas del genoma (1 cada 1000 bases en promedio), y que se observa en la población con una frecuencia de al menos 1%. En la especie humana se estima que existen en torno a de 12 millones de SNPs, de los que a finales de 2007 se han identificado algo más de un tercio.
T
TagSNP - TagSNP
Es un SNP (Single Nucleotide Polymorphim) o polimorfismo de una base en una región del genoma en la que existe un alto desequilibrio de ligamiento, y que representa al conjunto de SNPs dentro ese bloque haplotípico. Con el análisis de los tagSNPs es posible inferir la totalidad de la variación genética e identificar asociaciones fenotípicas sin genotipar el resto de los SNPs en esta región cromosómica.
TCGA -
Véase el Atlas del Genoma del Cáncer.
Transferencia genética - Gene transfer
Transferencia de material genético, que puede comprender desde un pequeño segmento de adn hasta un genoma entero, de una célula humana a otro tipo de célula en cultivo para estudiar la frecuencia con que los marcadores genéticos conocidos son transferidos conjuntamente al genoma receptor; se utiliza para determinar la proximidad física de los marcadores genéticos en el genoma humano; se utiliza asimismo para estudiar la expresión y regulación génica.
Transposon (Tn) -
Son regiones de ADN (200-2000 pares de bases) capaces de moverse de manera autosuficiente de una parte a otra parte del genoma, mediante un mecanismo de corta-pega y cuya consecuencia es la producción de mutaciones.
U
V
Valor C - C Value
La cantidad de ADN por genoma haploide (un solo juego cromosómico), en el caso de los organismos diploides como el ser humano, el valor C es la cantidad correspondiente a un juego de 23 cromosomas.
VNTR - Variable Number of Tandem Repeats
Acrónimo inglés de "Variable Number of Tandem Repeats". Es una región en el genoma hipervariable donde una secuencia de entre 6-25 nucleótidos aparece repetida en tándem. Estas regiones son polimórficas, por lo que cada individuo presenta diferente número de repeticiones. También se les conoce como minisatélites, y son utilizados como marcadores genéticos en estudios de ligamiento y en genética forense.