- Todos
- A
- B
- C
- D
- E
- F
- G
- H
- I
- J
- K
- L
- M
- N
- Ñ
- O
- P
- Q
- R
- S
- T
- U
- V
- W
- X
- Y
- Z
A
Adaptador (relativo a secuenciación masiva) - Adapter
Cebador que se une a los extremos 5´y 3´ de cada fragmento de ADN en una librería de secuenciación. Actúa en la amplificación y secuenciación del ácido nucleico adyacente.
Adenina - Adenine
ADN (Ácido desoxirribonucleico) - DNA ( Deoxyribonucleic acid)
Es la molécula portadora la información genética, que posibilita su transmisión de una generación a la siguiente.
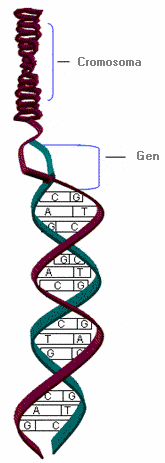
El ADN está constituido por dos cadenas de nucleótidos complementarias unidas entre sí por puentes de hidrógeno, enrollada la una sobre la otra formando una doble hélice. El elemento esencial del ADN es el nucleótido formado por un azúcar, la desoxirribosa, una base nitrogenada y un grupo fosfato. Las bases nitrogenadas son purinas: Adenina (A) y Guanina (G), y pirimidinas: Timina (T) y Citosina (C). Estas bases se unen al carbono 1 de la desoxirribosa para formar los nucleósidos, que a la vez se unirán con los grupos fosfato para formar los nucleótidos. Las bases nitrogenadas se unen de manera específica, guanina con citosina y adenina con timina, mediante puentes de hidrógeno.
El orden o secuencia de nucleótidos es la estructura primaria del ADN. La información genética reside en la secuencia de bases. Los genes son segmentos del ADN que codifican para proteínas. El orden de las bases en el gen determina la secuencia de aminoácidos que compondrán la proteína.
En las células eucariotas, el ADN se encuentra en el núcleo celular formando los cromosomas.
ADNc - cADN
ADN complementario, generado a partir del molde de ARN, mediante la retrotranscripción del ARNm maduro.
Alineamiento de secuencias - Sequence alingment
Alteración de la secuencia - Sequence alteration: (sinónimo: mutación)
Cualquier cambio producido en el adn[o mas concretamente en un gen con respecto a su estado natural. Si no tiene repercusión clínica se denomina polimorfismo; cuando se trata de un cambio que tiene o puede tener repercusión clínica se suele denominar mutación. Cuando no se conoce si el cambio tiene o no repercusión clínica se habla de cambio o variante de significado desconocido.
Alu, Secuencias repetidas Alu - Alu, Alu element
Secuencias de ADN repetidas distribuidas a lo largo del genoma. Son las secuencias repetidas más frecuentes, representando el 5% del genoma humano. Su nombre se debe a que son reconocidas y cortadas por la enzima de restricción Alu.
Amplicón - Amplicon
Fragmento de ADN amplificado por la reacción en cadena de la polimerasa (PCR) o cualquier otro proceso que dé lugar a la producción de diferentes copias de ese fragmento.
Amplificación - Amplification
Cualquier proceso que causa la replicación de una secuencia concreta de ADN en cantidad desproporcionadamente elevada.
Amplificación puente (relativo a secuenciación masiva) - Bridge amplification
Generación de copias in situ de una molécula específica de ADN en un soporte sólido cubierto de cebadores.
Análisis basados en ADN - DNA-based analysis
Pruebas que implican el estudio de ADN mediante un análisis de ligamiento, secuenciación o cualquiera de los diferentes métodos utilizados para la detección de mutaciones.
Análisis de dosis - Dosage analysis
Método para determinar la cantidad de diversos componentes, incluyendo ADN, ARN y proteínas, por comparación con un estándar conocido; se puede utilizar para determinar el número de copias de una secuencia de ADN (es decir, para investigar posibles mutaciones por duplicación y deleción), mediante la comparación visual de la intensidad de las bandas o la cuantificación numérica con densitometría. Si un gen presenta copias adicionales, la intensidad es de más del 100% en el gel o película; mientras que si se ha perdido una copia del gen, la intensidad es de aproximadamente un 50%.
Análisis de fragmentos de restricción de longitud polimórfica - Restriction fragment length polymorphism analysis (sinónimo: análisis RFLP, estudios RFLP)
Estudio de los fragmentos de ADN de tamaño previsible originados por la digestión (corte) de una cadena de ADN por una enzima de restricción específica. Las alteraciones (mutaciones o polimorfismos) producidas en la secuencia de ADN que destruyen o crean nuevos sitios de corte en el ADN, modifican el tamaño (y el número) de los fragmentos de ADN resultantes de la digestión por una enzima de restricción específica.

Análisis de ligamiento - Linkage analysis (sinónimo: análisis indirecto del ADN)
Estudio de los polimorfismos de las secuencias de ADN (variantes normales) que están próximos a/o dentro de un gen de interés (ligamiento) para identificar, en una misma familia, la segregación de una mutación patológica en un gen determinado.
EXPLICACIÓN:
Las tres fases del análisis de ligamiento son las siguientes:
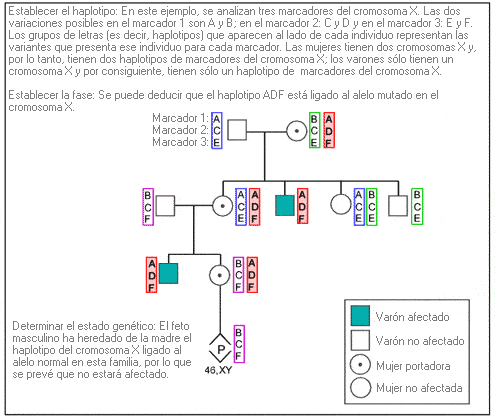
1. Establecer los haplotipos de cada individuo: Se analizan los múltiples marcadores de ADN dispuestos a cada lado (marcadores flanqueantes) o dentro (marcadores intragénicos) de la región del gen de interés, con objeto de determinar el conjunto de marcadores (haplotipos) de cada miembro de la familia.
2. Establecer la fase, comparando los haplotipos entre los miembros de la familia: Mediante la comparación de los haplotipos de los miembros de la familia con un estado genético conocido (es decir, afectados, no afectados), se puede identificar el haplotipo asociado con el alelo patológico.
3. Determinar el estado genético: Una vez establecido el haplotipo asociado con la enfermedad, es posible determinar el estado genético de los miembros de una familia de riesgo.
Ejemplo: Análisis de ligamiento para el diagnóstico prenatal de una enfermedad ligada al cromosoma X.
Algunas implicaciones clínicas
El análisis de ligamiento se utiliza generalmente cuando el análisis de ADN directo no es viable, porque se desconoce el gen de interés o no se puede detectar una mutación en ese gen en una familia específica.
En la mayoría de los casos, el haplotipo en sí carece de significación; sólo tiene sentido en el contexto de un estudio familiar. Sólo en el caso raro de que el desequilibrio del ligamiento sea un haplotipo específico, habrá una probabilidad muy elevada de que esté asociado con una mutación patológica específica.
La exactitud del análisis de ligamiento depende de:
La exactitud del diagnóstico clínico en un miembro o miembros de la familia que estén afectados.
La distancia entre la mutación patológica y los marcadores. El análisis de ligamiento puede proporcionar resultados falso positivo o falso negativo si, durante la formación de los gametos (meiosis), se produce recombinación que implique a los marcadores y suponga un intercambio entre el material genético de los cromosomas heredados de la madre o del padre. El riesgo de recombinación es proporcional a la distancia entre la mutación patológica y los marcadores. El riesgo de recombinación es más bajo si se utilizan marcadores intragénicos.
La informatividad de los marcadores genéticos en la familia del paciente. Si la secuencia de ADN de una variante determinada difiere entre los cromosomas heredados de la madre y del padre, este marcador es informativo. Por el contrario, si la secuencia de ADN de una variante determinada no difiere entre los dos cromosomas, el marcador no es informativo.
Análisis de matrices de ADN - DNA microarray analysis
Análisis de metilación - Methylation analysis
Métodos utilizados para evaluar el estado de metilación de un gen (unión de los grupos metilo a las citosinas del ADN). Los genes metilados no se expresan. La metilación desempeña un papel importante en la inactivación del cromosoma X y en la impronta genética.
1. Análisis con enzimas de restricción sensibles a metilación: El ADN es digerido (cortado) por una enzima de restricción que sólo puede digerir ADN no metilado. Se realizan análisis adicionales (por ejemplo, Southern blot y/o PCR) para determinar si el ADN se ha cortado. La presencia de ADN no cortado indica que el ADN está metilado, mientras que la presencia de ADN cortado indica que éste no está metilado.
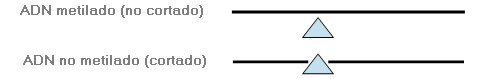
2. Análisis de secuencia tratada con bisulfito: El ADN es tratado para sustituir todos los nucleótidos citosina no metilados por nucleótidos timina. Se secuencia el ADN tratado. El ADN metilado conserva su secuencia original (normal), mientras que el ADN no metilado presenta una secuencia anormal en la que todos los nucleótidos citosina (C) se han sustituido por nucleótidos timina (T).

Análisis de microsatélites - Microsatellite analysis
Uso de secuencias repetitivas muy variables, que se encuentran en las regiones microsatélites adyacentes a los genes o intragénicas o en otras áreas de interés, como marcadores para el análisis de ligamiento, huellas moleculares de adn u otras aplicaciones diagnósticas.
Análisis de secuencias - Sequence analysis (sinónimo: secuenciación génica, secuenciación)
Proceso mediante el cual se determina la secuencia de nucleótidos en un segmento de ADN.
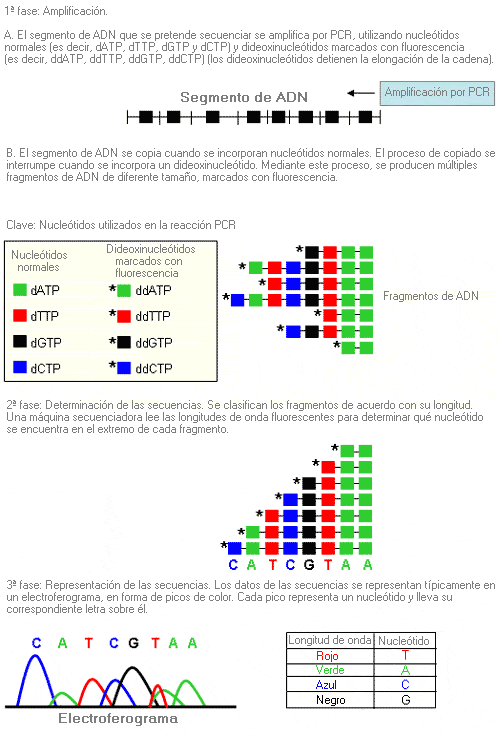
Se puede secuenciar un gen completo, pero la mayoría de las veces se secuencian sólo regiones específicas del gen que muy probablemente contendrán mutaciones (p. ej. los exones y los límites entre los intrones y los exones).
Al secuenciar un segmento de ADN, se identifican la mayoría de las variaciones producidas con respecto a la secuencia normal (por el contrario, el análisis mutacional identifica sólo mutaciones específicas en un segmento determinado de ADN).
Tipos de alteraciones de las secuencias que se pueden detectar:
Alteraciones de secuencia descritas en la literatura como patógenas
Alteraciones de secuencias previsiblemente patógenas, pero no descritas en la literatura
Alteraciones de secuencias de significación clínica imprevisible
Alteraciones de secuencias previsiblemente benignas, pero no descritas en la literatura
Alteraciones de secuencias benignas descritas en la literatura
Posibilidades en caso de que no se detecten alteraciones de las secuencias:
El paciente no presenta una mutación en el gen analizado (p. ej. hay una alteración de la secuencia en otro gen, en un locus diferente)
El paciente presenta una alteración de la secuencia que no se puede detectar mediante el análisis de secuencias (p. ej. deleción de tamaño grande)
El paciente presenta una alteración de la secuencia en una región del gen (p. ej. una región intrónica o reguladora) que no se ha detectado en el análisis de laboratorio
1Adaptado de ACGM Recommendations for Standards for Interpretation of Sequence Variations (2000)
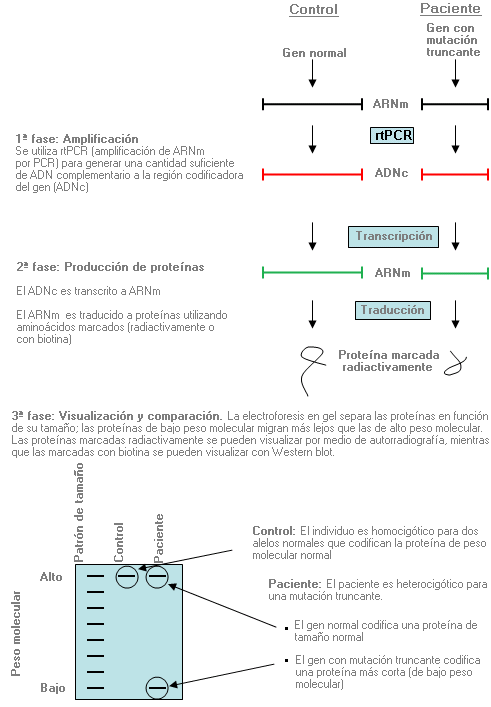
Análisis de truncación de proteínas - Protein truncation testing (sinónimo: PTT)
Método utilizado para identificar las proteínas que se acortan (truncan) a consecuencia de mutaciones que causan específicamente la terminación prematura del proceso de traducción del ARNm.
Análisis del haplotipo - Haplotype analysis
Estudio de genética molecular para identificar un conjunto de segmentos de adn que están estrechamente relacionados en su modo de herencia (ligados). Se utiliza en el análisis de ligamiento o cuando un rasgo o característica determinados presentan desequilibrio de ligamiento con un marcador genético o un grupo de [GLOSARIO]marcador[FIN_GLOSARIO]es.
Análisis del origen parental - Parental origen analysis
Análisis directo del ADN - Direct DNA analysis
La utilización de técnicas de secuenciación, análisis mutacional, rastreo de mutaciones o cualquier otro medio de estudio molecular genético, para detectar variantes en un gen que pueden estar asociadas a trastornos genéticos específicos. El análisis directo del adn es posible sólo cuando se conoce el gen o genes o la región genómica, asociados con la enfermedad.
Las técnicas utilizadas para el análisis de ADN directo pueden ser las siguientes:
Análisis de secuencias
Rastreo de mutaciones (p. ej. CSGE, DHPLC, SSCP)
Análisis mutacional (p. ej. PCR, Southern blot, análisis de repeticiones de trinucleótidos, FISH, análisis de truncación de proteínas (PTT), estudios de heterocigosidad, análisis RFLP, ASO)
El análisis de ADN directo contrasta con el análisis de ligamiento (que anteriormente se denominaba análisis de ADN indirecto)
Algunas implicaciones clínicas
El análisis de ADN directo sólo es posible cuando se conoce el gen (genes) o la región genómica asociados con una enfermedad
El análisis de ligamiento se puede realizar cuando no se conoce el gen o los genes asociados con una enfermedad
Arginina - Arginine
La arginina (arg o R) es uno de los 20 aminoácidos que se encuentran formando parte de las proteínas. Es un aminoácido condicionalmente esencial (se necesita en la dieta solo bajo ciertas condiciones), y puede estimular la función inmunológica al aumentar el número de leucocitos. La arginina está involucrada en la síntesis de creatina, poliaminas y en el ADN. En el ARNm está codificado por los codones CGU, CGC, CGA, CGG, AGA, y AGG.
ARN mensajero (ARNm) - Messenger RNA (mRNA)
Es el ácido ribonucleico que transfiere el código genético procedente del núcleo celular a un ribosoma en el citoplasma. Se forma durante el proceso de transcripción del ADN al ARN, durante el cual la ARN polimerasa debe unirse al ADN con ayuda de cofactores de transcripción. El proceso de transcripción se lleva a cabo en el núcleo en eucariotas y en el citoplasma en procariotas.
Atlas del Genoma del Cáncer - The Cancer Genome Atlas TCGA
Proyecto internacional iniciado en el año 2005 con el objetivo de catalogar los cambios moleculares, a nivel de ADN, ARN, proteico y epigenético, existentes en los diferentes tipos de tumores mediante plataformas de análisis masivo y potentes análisis bioinformáticos. Con este proyecto se pretende mejorar el diagnóstico y el tratamiento de los pacientes oncológicos además de proporcionar información para prevenir y entender cuáles son los mecanismos moleculares asociados al desarrollo de esta enfermedad (https://cancergenome.nih.gov/)
B
BAC, Cromosoma Artificial Bacteriano - Bacterial Artificial Chromosome
Segmento grande de ADN, generalmente constituido por 100.000-200.000 bases, de cualquier especie que es clonado e introducido en una bacteria. Una vez dentro de la bacteria, pueden obtenerse múltiples copias del mismo.
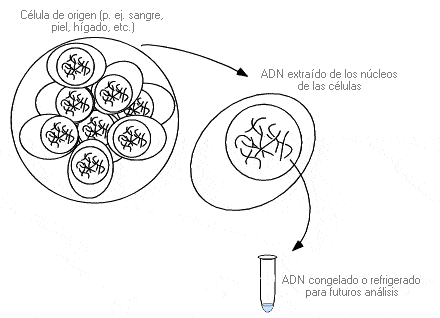
Banco de ADN - DNA banking
Proceso de almacenaje durante un periodo indefinido del adn extraído de cualquiera de las posibles fuentes celulares, conservado bajo congelación o refrigeración para futuros análisis.
C
cADN, Librería de - cDNA library
Colección de secuencias de adn generadas a partir de la secuencia del ARN mensajero (mARN). Estas librerías contienen sólo adn codificante de proteínas (genes), quedando excluídas las secuencias de adn que no codifican.
Cebador - Primer
Una secuencia corta de oligonucleótidos que se utiliza en la reacción en cadena de la polimerasa (PCR) para la creación de moléculas más largas de ADN.
ChIP (chromatin immunoprecipitation) -
Citosina - Cytosine
Clon - Clone
Copia idéntica de una secuencia de ADN, o de todo un gen, o de un ser vivo. Una o más células derivadas de un solo antecesor e idénticas a éste.
Clonación - Cloning
Aislar un gen o una secuencia específica de ADN.
Codón - Codon
En el adn o ARN, secuencia de tres nucleótidos que codifica determinado aminoácido o indica el comienzo o la terminación del proceso de traducción (codón de inicio, parada o terminación).
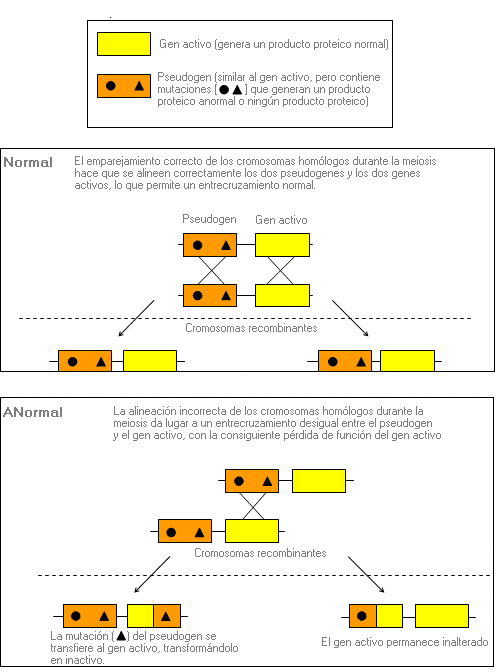
Conversión génica - Gene conversion
Transferencia de secuencias de ADN entre dos genes muy similares, que se produce la mayoría de las veces por un entrecruzamiento desigual durante la meiosis. Puede ser un mecanismo que da lugar a una mutación si la transferencia de material altera la secuencia codificadora del gen o si el mismo material transferido contiene una o más mutaciones.
Puesto que los pseudogenes guardan una gran similitud con el gen activo, puede producirse un emparejamiento erróneo. La recombinación entre el pseudogen y el gen activo podría transferir una mutación desde el pseudogen al gen activo.
Cromátidas - Chromatids
Cromosoma - Chromosome
Estructura física, también llamada cromatina, que consiste en una molécula de adn compactado organizada en genes y mantenida por proteínas llamadas histonas.
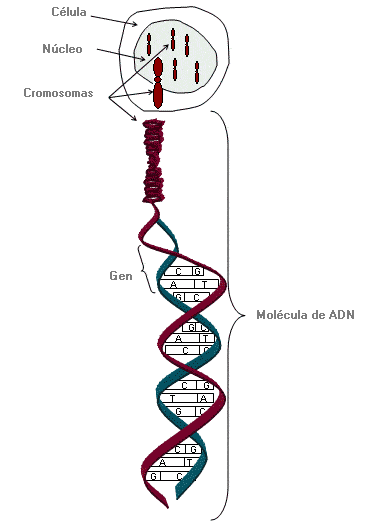 Los cromosomas están localizados en el núcleo de una célula. Presentan una estructura en forma de hélice compacta, excepto durante el proceso de división celular en el que se alargan.
Los cromosomas están localizados en el núcleo de una célula. Presentan una estructura en forma de hélice compacta, excepto durante el proceso de división celular en el que se alargan.
Las células humanas contienen normalmente 46 cromosomas dispuestos en 23 pares, 22 de los cuales son autosomas (cromosomas no sexuales) y un par son los cromosomas sexuales.
Diagrama adaptado de “Understanding Genetic Testing”, National Institutes of Health
D
Deleción - Deletion
Alteración genética consistente en la pérdida de un segmento de ADN. Su magnitud es variable, pudiendo ser tan pequeñas como un solo par de bases o tan grandes que afecten a uno o más genes.
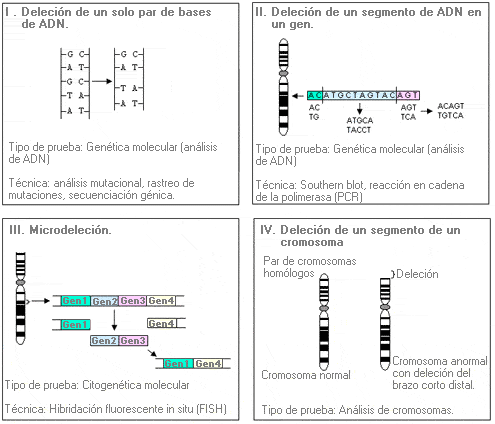
Densitometría - Densitometry
Método para identificar la dosis o la expresión génica mediante la medición de la absorción de la luz en una autorradiografía (película) de una banda (o mancha) que representa el ADN, el ARN o una muestra de proteínas. Es una técnica útil para detectar mutaciones por duplicación y mutaciones por deleción heterocigótica.
Desequilibrio de ligamiento - Linkage disequilibrium
En una población, la herencia simultánea y transmitida de un conjunto de marcadores de adn específicos a lo largo de generaciones sucesivas sin que se produzca recombinación entre ellos.
DGGE - (Denaturing gradient gel electrophoresis) (sinónimo: Electroforesis en gel con gradiente desnaturalizante)
Técnica de electroforesis para muestras de adn de cadena doble que utiliza un gel en el que se crea un gradiente desnaturalizante mediante una sustancia química, por ejemplo, urea. Se emplea para la identificación de mutaciones que afectan al patrón de migración del adn en esas condiciones de electroforesis. La presencia de la mutación se detecta mediante comparación del patrón electroforético (punto de desnaturalización) de la muestra problema con la secuencia salvaje u original.
DHPLC (Denaturing high-performance liquid chromatography) -
Cromatografía líquida desnaturalizante de alto rendimiento. En genética es un método utilizado para la detección de mutaciones de uno o pocos nucleótidos. La base de la técnica es la diferencia de migración que presentan fragmentos homodúplex (secuencia doble de ADN que presenta un 100% de apareamiento entre las bases) frente a los heterodúplex (apareamiento no total) durante su migración por un gradiente en una columna.
Disomía uniparental - Uniparental disomy (sinónimo: UPD)
Situación en la que un par de cromosomas homólogos o algún segmento de un par de cromosomas homólogos son heredados de sólo uno de los progenitores. La disomía uniparental puede dar lugar a un fenotipo anormal en algunos casos. También se ha descrito disomía uniparental somática que afecta sólo a las células que componen un determinado tejido. En estos casos el origen de la disomía no es la herencia directa sino un proceso anómalo de replicación del ADN.
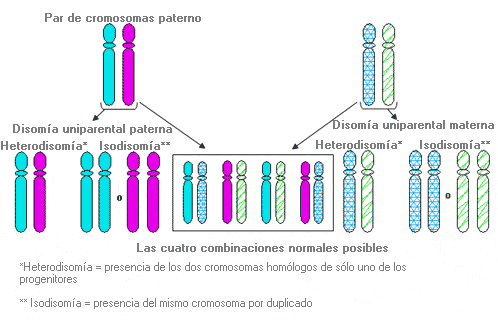
La disomía uniparental da lugar a un fenotipo anormal cuando los cromosomas implicados llevan impronta, por lo que los genes de estos cromosomas sólo tienen un alelo activo (es decir, sólo el alelo materno o paterno del par es activo).
Síndromes asociados con disomía uniparental (UPD)
Síndrome de Prader-Willi (UPD15 materno)
Síndrome de Angelman (UPD15 paterno)
Diabetes mellitus neonatal transitoria (UPD6 paterno)
Síndrome de Russell-Silver (UPD7 materno)
Síndrome de Beckwith-Wiedeman (UPD11 materno)
Síndrome de MUPD14 (UPD14 materno)
Dominio - Domain
Región específica o secuencia de aminoácidos de una proteína, que está asociada a una función determinada o al segmento de adn correspondiente.
Duplicación - Duplication
Modificación de la secuencia genética (patológica o no) consistente en la presencia de un segmento adicional de adn que da lugar a copias repetidas de una parte de un gen, un gen entero o una serie de genes. Su origen más probable es un entrecruzamiento desigual durante la replicación del segmento duplicado durante la formación de los gametos en la meiosis.
E
Edición génica o genómica - Gene/genome editing
Técnica que permite a los investigadores alterar el ADN de las células u organismos para insertar, eliminar o modificar un gen o secuencia de genes para silenciar, mejorar o cambiar las características del gen.
La edición del genoma o la ingeniería del genoma es un tipo de ingeniería genética en la que el ADN se altera en el genoma de una célula u organismo vivo usando nucleasas modificadas genéticamente o "tijeras moleculares". Estas nucleasas crean roturas de doble cadena específicas de sitio (double strand breaks, DSB) en ubicaciones deseadas en el genoma. Las roturas inducidas de doble cadena se reparan a través de los sistemas de reparación de “non homology en joining” (NHEJ) o la recombinación homóloga (HR), dando como resultado mutaciones dirigidas.
Electroforesis - Electrophoresis
Electroforesis en gel con gradiente desnaturalizante - Denaturing gradient gel electrophoresis (sinónimo: DGGE)
Técnica de electroforesis para muestras de ADN de cadena doble que utiliza un gel en el que se crea un gradiente desnaturalizante mediante una sustancia química, por ejemplo, urea. Se emplea para la identificación de mutaciones que afectan al patrón de migración del ADN en esas condiciones de electroforesis. La presencia de la mutación se detecta mediante comparación del patrón electroforético (punto de desnaturalización) de la muestra problema con la secuencia salvaje u original.
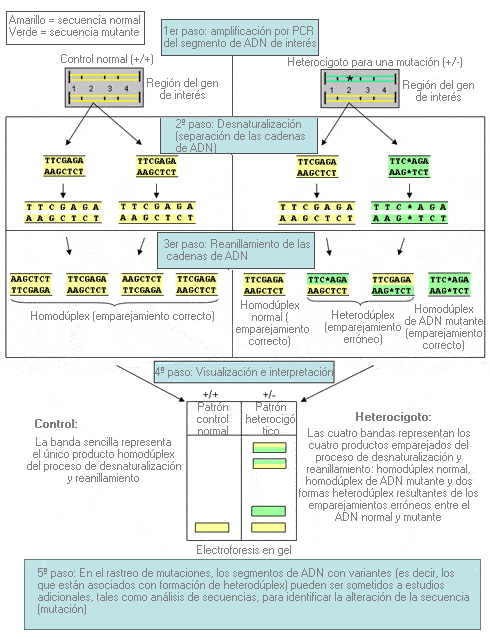
Electroforesis en gel sensible a conformación - Conformation-sensitive gel electrophoresis (sinónimo: CSGE)
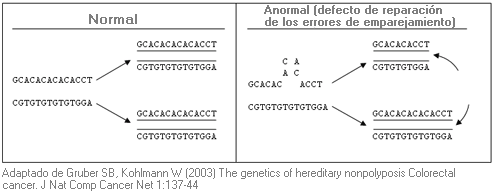
Técnica electroforética que consiste en el análisis de un segmento de ADN para detectar errores de emparejamiento entre pares de bases normales y mutadas (ver explicación esquemática). Se emplea para el rastreo de mutaciones.
Endonucleasa de restricción - Restriction endonuclease
Una enzima de restricción o endonucleasa de restricción es una enzima que escinde ADN en fragmentos en o cerca de sitios de reconocimiento específicos dentro de la molécula conocida como sitios de restricción. Las enzimas de restricción se clasifican comúnmente en cuatro tipos. Para cortar el ADN, todas las enzimas de restricción hacen dos incisiones, una a través de cada cadena principal de azúcar-fosfato (es decir, cada cadena) de la doble hélice de ADN.
Enfoque isoeléctrico - Isoelectric focusing
Método de detección de mutaciones basado en la separación de proteínas en función del valor de pH en el que su carga neta es cero (punto isoeléctrico). Se suele utilizar conjuntamente con la técnica western blot para permitir la identificación de productos proteicos de tipo salvaje frente a mutante. La alteración de una secuencia de adn que dé lugar a una sustitución de aminoácidos puede variar el punto isoeléctrico de una proteína.
Entrecruzamiento - Crossing over (sinónimo: recombinación)
Intercambio de un segmento de adn entre los dos cromosomas homólogos durante la meiosis. Tiene como resultado una combinación nueva de material genético en el gameto.
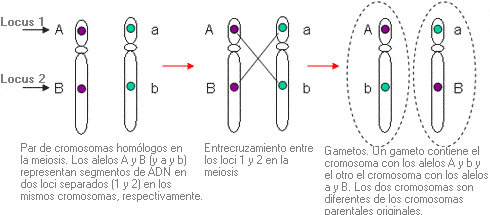 Dos genes localizados en el mismo cromosoma pueden segregarse juntos o no. El entrecruzamiento entre los cromosomas homólogos durante la meiosis da lugar a combinaciones nuevas de genes en el gameto, y por lo tanto, en los descendientes.
Dos genes localizados en el mismo cromosoma pueden segregarse juntos o no. El entrecruzamiento entre los cromosomas homólogos durante la meiosis da lugar a combinaciones nuevas de genes en el gameto, y por lo tanto, en los descendientes.
Gracias al proceso de entrecruzamiento, cada cromosoma transmitido de un progenitor a su descendiente contiene algunos segmentos heredados de las dos ramas ascendientes de ese progenitor. Por ello, el entrecruzamiento desempeña un papel importante asegurando la variabilidad genética entre individuos, incluso de la misma familia.
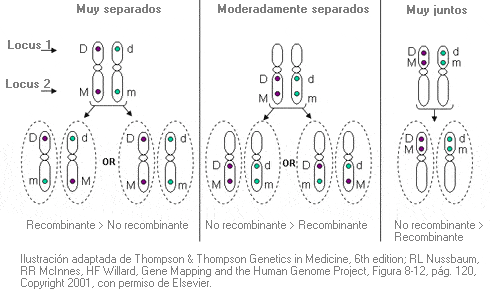
El comportamiento durante la meiosis de los alelos en dos loci (1 y 2) del mismo cromosoma depende de su proximidad.
Epidemiología genética - Genetic epidemiology
La epidemiología genética estudia la interacción entre los factores genéticos y ambientales que dan origen a las enfermedades del ser humano. Utiliza marcadores de ADN que son estudiados mediante análisis molecular. Dado el volumen de datos (que hacen referencia a poblaciones numerosas) lo habitual es que sean almacenados en bases de datos y tratados mediante herramientas bioinformáticas. Las estrategias de investigación incluyen, entre otras, análisis de segregación familiar, asociaciones alélicas y estudios de interacción gen y medio ambiente.
Epigenética - Epigenetics
Cambios reversibles de ADN (modificaciones químicas) que alteran la expresión de los genes que son objeto de los mismos. Los tipos de cambios epigenéticos más frecuentes incluyen la metilación de la citosina del ADN así como la metilación, acetilación y fosforilación de las proteínas histonas que conforman la cromatina. La metilación más estudiada es una modificación del ADN, en la que un grupo metilo es trasferido desde S-adenosilmetionina a una posición C-5 de citosina por una ADN-5 metiltransferasa. La metilación del ADN ocurre, casi exclusivamente, en dinucleótidos CpG, teniendo un importante papel en la regulación de la expresión del gen. Estos factores genéticos son determinados por el ambiente celular en lugar de por la herencia.
Escisión reparación - Excision repair
Estudios de cigosidad - Zygosity testing
Proceso mediante el cual se comparan las secuencias de adn para determinar si los individuos nacidos en una gestación múltiple (mellizos, trillizos, etc.) son monocigóticos (idénticos) o dicigóticos (fraternales). Estos estudios se utilizan frecuentemente para identificar un donante apropiado para trasplante de órganos o para calcular el riesgo de susceptibilidad a desarrollar una enfermedad.
Estudios de genética molecular - Molecular genetic testing (sinónimos: análisis de ADN, análisis basados en ADN, estudios moleculares)
Pruebas que implican el estudio del ADN, mediante análisis de ligamiento, secuenciación o detección de mutaciones, utilizando uno o varios de los diferentes métodos disponibles.
Estudios de inestabilidad de microsatélites - Microsatellite instability testing (sinónimo: estudios MSI)
Análisis genéticos realizados en muestras tumorales que detectan la deficiencia en el sistema de reparación de errores de emparejamiento de bases del ADN. La deficiencia se manifiesta al comparar el número de repeticiones de nucleótidos en un grupo de marcadores tipo microsatélite en una muestra problema (tumor) frente al número observado en una muestra normal del mismo paciente. Se considera que hay estabilidad de microsatélites (MSS) cuando el número de repeticiones en cada marcador, tanto en el tejido tumoral como en el normal, es el mismo. Existe inestabilidad de microsatélites (MSI) cuando el número de repeticiones en el tejido tumoral y el normal es diferente.
Replicación de microsatélites
Procedimiento
Se extrae ADN de tejido tumoral y normal del mismo individuo.
Se evalúa un grupo de marcadores microsatélites. Generalmente, este grupo incluye los marcadores BAT25, BAT26, D2S123, D5S346 y D17S250; la mayoría de los laboratorios clínicos utilizan un grupo de 10 marcadores.
Interpretación1
Variación del número de repeticiones de nucleótidos entre el ADN extraído de tejido tumoral y normal:
MSI de frecuencia alta (MSI-H): 30% de los marcadores analizados muestran diferencias entre el ADN tumoral y el normal.
MSI de frecuencia baja (MSI-L): Mayor que cero, pero 30% de los marcadores analizados muestran diferencias.
Estabilidad de los microsatélites (MSS): Ninguno de los marcadores analizados muestra diferencias.
Seguimiento.
El hallazgo de inestabilidad de microsatélites (MSI) en el tumor de un individuo con cáncer de colon o endometrio diagnosticado a una edad temprana, con unos antecedentes familiares de cáncer relacionado con HNPCC o la presencia de tumores primarios múltiples en el mismo individuo, sugiere una mutación en línea germinal en un gen de reparación de errores de emparejamiento (p. ej. MLH1, MSH2, MSH6, PMS2, PMS1). En estos casos están indicados los estudios de genética molecular para la búsqueda de mutaciones en línea germinal.
Puesto que aproximadamente el 20% de los cánceres de colon esporádicos son MSI-H debido a mutaciones somáticas o a la metilación del gen MLH1, la edad a la que se desarrolla el tumor y los antecedentes familiares son factores a tener en cuenta a la hora de distinguir entre los cánceres de colon esporádicos que son MSI-H y los que están relacionados con HNPCC.
1The National Cancer Institute Workshop on Microsatellite Instability and RER Phenotypes in Cancer Detection and Familial Predisposition MSI testing recommendations include [Boland et al 1998]
Estudios de repeticiones de trinucleótidos - Trinucleotide repeat testing
Análisis genético cuyo objetivo es la cuantificación del número de repeticiones de grupos de tres nucleótidos en un segmento determinado de ADN.
Para cuantificar las repeticiones de trinucleótidos, se pueden utilizar dos tipos de análisis mutacional que se representan en el diagrama siguiente:
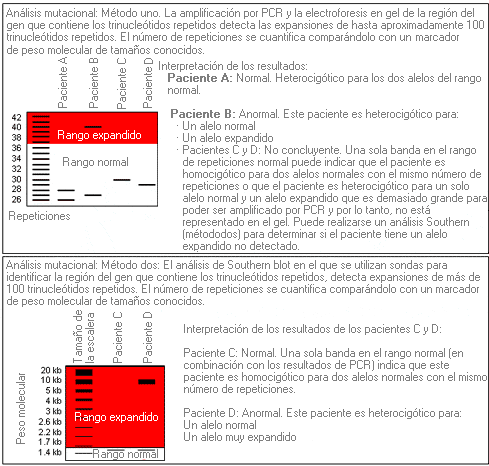
Algunas implicaciones clínicas:
El análisis Southern blot no se realiza de forma rutinaria en todos los laboratorios para clarificar la interpretación de los resultados de las bandas no concluyentes por PCR. Cuando se reciba un informe de un número de repeticiones “homocigóticas” normales, particularmente en un niño sintomático de corta edad que podría presentar una expansión grande, es recomendable que el médico confirme que se ha realizado el análisis Southern blot para evaluar la posibilidad de que haya una repetición de trinucleótidos muy expandida.
Si no está disponible el análisis Southern blot, se puede realizar un análisis mutacional basado en PCR/electroforesis en gel de los padres de un paciente con un número de repeticiones “homocigóticas” normales, para determinar si el paciente podría haber heredado de sus padres dos alelos con el mismo número de repeticiones.
Estudios diagnósticos - Diagnostic testing
Pruebas realizadas con objeto de confirmar o descartar una enfermedad genética conocida o que se sospecha en un individuo sintomático o, prenatalmente, en un feto con riesgo de desarrollar una patología genética específica.
Algunos ejemplos clínicos de estudios diagnósticos.
Distrofia muscular de Duchenne (DMD): Dado que se detectan deleciones o duplicaciones en el gen DMD en el 65% de los casos, el análisis de ADN de este gen en muestras de sangre es un procedimiento diagnóstico primario menos costoso e invasivo que el análisis de distrofina en biopsia muscular.
Hemocromatosis hereditaria (HHC): Dado que las manifestaciones clínicas y/o la saturación de transferrina sérica y la concentración de hierro sérico son compatibles con HHC, los estudios de genética molecular resultan menos costosos e invasivos que la biopsia hepática para confirmar el diagnóstico. Las mutaciones patológicas en HFE conocidas se detectan en aproximadamente el 87% de los casos de HHC.
Fibrosis quística. Mediante el estudio de las mutaciones en CFTR más comunes, se podrán detectar mutaciones en aproximadamente el 90% de los pacientes caucasianos descendientes de judíos no askenazi; sin embargo, en los individuos de cualquier etnia, los análisis de cloruro en muestras de sudor son una prueba diagnóstica más exacta.
Etiqueta de secuencia expresada - Expressed sequence tag, EST
Una etiqueta de secuencia expresada (o "expressed sequence tag", EST) es una subsecuencia corta de secuencia conocida dentro de una secuencia de cADN.
Exón - Exon
Secuencia codificante de ADN. El exón es la región de un gen que no se separa durante el proceso de splicing (corte y empalme) manteniéndose en el ARN mensajero.
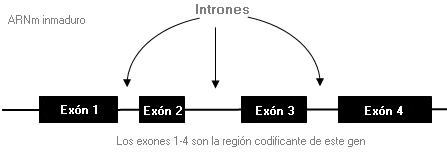
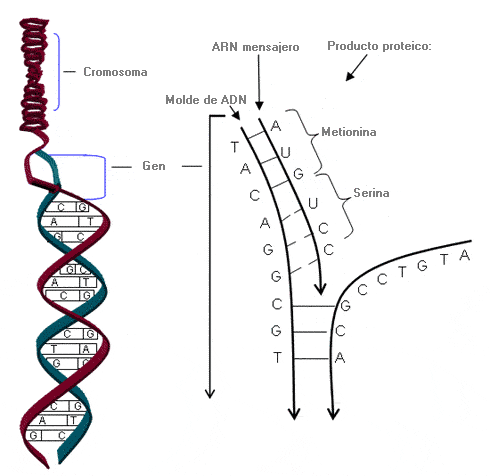
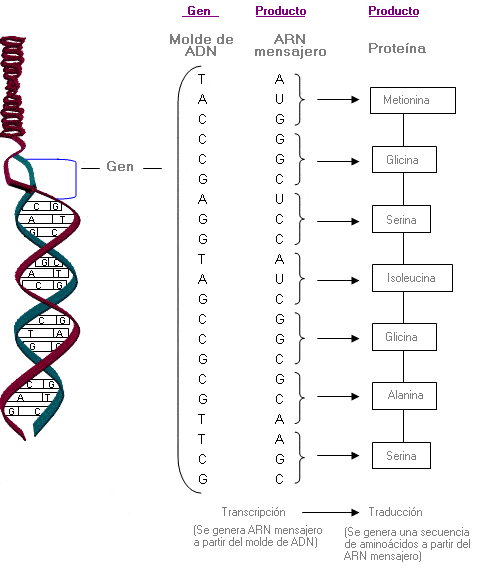
Figura 1. El ADN transcrito inicialmente a ARN mensajero inmaduro (ARNm) consta de secuencias codificantes (exones) y no codificantes (intrones).
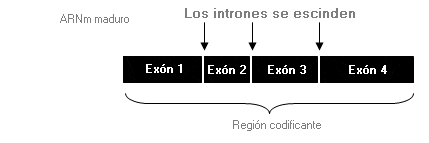
Figura 2. Los intrones se han escindido del ARNm inmaduro para formar ARNm maduro, con lo cual quedan sólo los exones que codificarán finalmente el producto aminoacídico.
Ilustraciones adaptadas de Thompson & Thompson Genetics in Medicine, 6th edition; RL Nussbaum, RR McInnes, HF Willard, The Human Genome: Structure and Function of Genes and Chromosomes, Figura 3-6, pág. 20, Copyright 2001, con permiso de Elsevier.
F
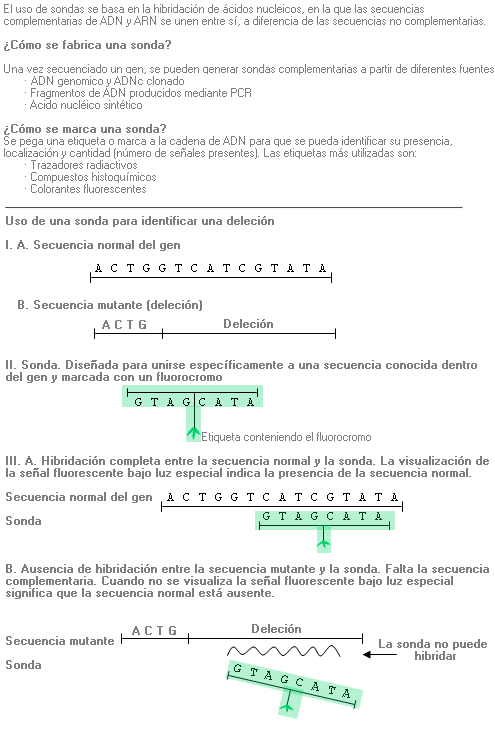
FISH - Fluorescent in situ hybridization (sinónimo: hibridación in situ con fluorescencia)
Técnica que se utiliza para identificar, en una muestra problema, la presencia de cromosomas determinados o regiones específicas de cromosomas cuya secuencia es conocida, (sondas de ADN), y que marcamos con fluorescencia. La detección se realiza tras la hibridación (reconocimiento de la secuencia y emparejamiento base a base) de la Sonda de ADN marcada sobre el ADN cromosómico desnaturalizado de la muestra problema.
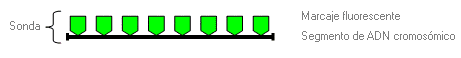
1ª fase. Preparación de la sonda. Una sonda es un segmento de ADN marcado con fluorescencia que es complementario a la región cromosómica de interés.
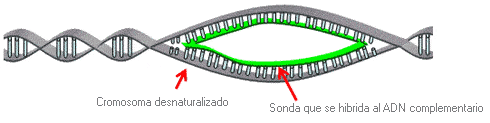
2ª fase. Hibridación. Los cromosomas desnaturalizados, fijados en el portaobjetos del microscopio, son expuestos a la sonda marcada con fluorescencia. La hibridación (fusión) tiene lugar entre la sonda y el ADN cromosómico complementario (es decir, se acopla).
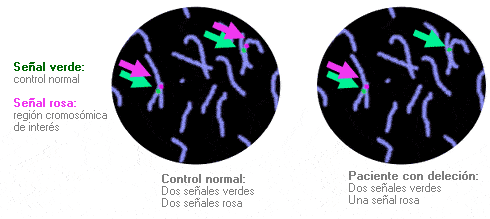
3ª fase. Visualización. Tras la hibridación, se examina el portaobjetos al microscopio con luz fluorescente. Las señales fluorescentes indican la presencia de ADN cromosómico complementario, la ausencia de tales señales implica que no hay ADN cromosómico complementario.
FISH en interfase - Interphasic FISH
Técnica que se utiliza para identificar, en una muestra problema, la presencia de cromosomas determinados o regiones específicas de cromosomas cuya secuencia es conocida, (sondas de ADN), y que marcamos con fluorescencia. La detección se realiza tras la hibridación (reconocimiento de la secuencia y emparejamiento base a base) de la sonda de ADN marcada sobre el ADN cromosómico desnaturalizado de la muestra problema. En la FISH en interfase, las sondas se introducen en el núcleo interfásico. La FISH en interfase se utiliza, por ejemplo para detectar tipos específicos de aneuploidía en células fetales, así como determinadas deleciones, duplicaciones y otras anomalías en células tumorales. Al contrario que la FISH en metafase, la FISH en interfase no permite visualizar los cromosomas individualizados; por lo tanto, no se podrán detectar determinados reordenamientos estructurales.
FISH en metafase - Metaphase FISH
Técnica que se utiliza para identificar, en una muestra problema, la presencia de cromosomas determinados o regiones específicas de cromosomas cuya secuencia es conocida, (sondas de ADN, y que marcamos con fluorescencia). La detección se realiza tras la hibridación (reconocimiento de la secuencia y emparejamiento base a base) de la sonda de ADN marcada sobre el ADN cromosómico desnaturalizado de la muestra problema. En la FISH en metafase, se utiliza como muestra cromosomas obtenidos a partir de células en mitosis. La división celular tiene varias fases y en la fase denominada metafase, los cromosomas se condensan y se pueden distinguir de forma individual. Al contrario que la FISH en interfase, la FISH en metafase permite visualizar la hibridación de las sondas de ADN marcado sobre cromosomas individualizados, lo que facilita la localización de la anomalía en el cromosoma específico en el que se produce.
FISH subtelomérica - Subtelomeric FISH
Técnica en la que se utilizan sondas de ADN específicas para las áreas subteloméricas de los brazos largos y cortos de cada cromosoma, que permite la detección de deleciones y translocaciones subteloméricas crípticas y submicroscópicas, que pueden ser causa de retraso mental.
Footprinting - Footprinting
Técnica empleada para la identificación de secuencias de ADN que ligan proteínas específicas. Al unirse la proteína ésta protege algunos enlaces fosfodiesteres del ADN y por lo tanto también protege de la acción de ADNasas.
G
Gen - Gene
Gen de fusión - Fusion gene
Unión accidental del ADN de dos genes. Los genes de fusión pueden ocurrir como resultado de una translocación, deleción o inversión cromosómica. Las fusiones génicas pueden dar lugar a proteínas híbridas o a la regulación incorrecta de la unidad de transcripción de un gen por los elementos reguladores en cis (potenciadores) de otro.
Gen mapeado - Mapped gene
Gen para el que se ha establecido su posición relativa en un segmento de ADN o en un cromosoma.
Gen reportero - Reporter gene
Gen cuyo producto es fácilmente detectable o medible y, por lo tanto, puede usarse como un indicador de si una construcción de ADN se ha transferido con éxito. Por ejemplo: cloranfenicol transacetilasa.
Genómica - Genomics
La genómica se encarga del estudio de los genomas. Se considera a un genoma como el conjunto de información genética (ADN) de un organismo. A diferencia de la genética clásica, en la que a partir de un fenotipo se busca el o los genes responsables de dicho fenotipo, la genómica tiene como objetivo predecir la función de los genes a partir de su secuencia. Para conseguir este tipo de objetivos emplea herramientas basadas en análisis genéticos masivos (del orden de miles de genes) frecuentemente de forma simultánea (en un reducido número de experimentos).
Guanina - Guanine
Una de las cuatro bases que componen el ADN. Las otras tres son adenina, citosina y timina. La guanina siempre se empareja con la citosina.
Guía RNA - Guide RNA
Segmentos cortos de ARN utilizados para dirigir la enzima de corte de ADN a la ubicación de destino en el genoma. Los segmentos de gARN contienen la región de homología con la secuencia diana (normalmente 20 bases) y una secuencia que interacciona con la nucleasa (por ejemplo, Cas9). Los gARN utilizados en la edición del genoma son sintéticos y no ocurren en la naturaleza.
H
Helicasa - Helicase
Enzima que participa en el proceso de replicación de la molécula de ADN desenrrollando la doble hélice cerca del punto de bifurcación de la horquilla replicadora.
Heterocromatina - Heterochromatin
Forma compacta de ADN, que se presenta en múltiples variedades. Estas variedades se encuentran en un continuo entre los dos extremos de heterocromatina constitutiva y facultativa.
Histonas - Histones
Proteínas altamente alcalinas que se encuentran en los núcleos de células eucarióticas y cuya función es el empaquetamiento y ordenamiento del ADN en unidades estructurales llamadas nucleosomas. Son los principales componentes proteicos de la cromatina, que actúan como carretes alrededor de los cuales se enrolla el ADN y desempeñan un papel en la regulación genética.
I
Inestabilidad de microsatélites - Microsatellite instability (sinónimos : MSI, fenotipo de error de replicación, RER)
Discrepancia entre el tamaño de los microsatélites en el adn de tejido tumoral comparado con el no tumoral de la misma persona, a consecuencia de las mutaciones producidas en un gen en la vía de reparación de los errores de emparejamiento de bases del adn (MMR). Estos gen es si no estuviesen mutados corregirían normalmente estos errores.
Inserción - Insertion
Anomalía cromosómica en la que el material de un cromosoma se inserta en otro cromosoma; mutación en la que se inserta un segmento de ADN en un gen o en otro segmento de ADN, alterando potencialmente la secuencia codificante.
Integración - Integration
Proceso de recombinación que inserta una molécula de ADN pequeña en un locus más grande.
Intrón - Intron
Secuencia no codificante de ADN que se transcribe a ARN mensajero (ARNm) en su estado inmaduro, pero es escindida del mismo al transformarse en ARNm maduro antes de la traducción.

Figura 1. El ADN transcrito inicialmente a ARN mensajero inmaduro (ARNm) consta de secuencias codificadoras (exones) y no codificadoras (intrones).
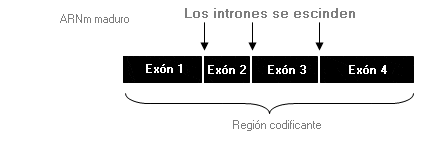
Figura 2. Los intrones se han escindido del ARNm inmaduro para formar ARNm maduro, con lo cual quedan sólo los exones que codificarán finalmente el producto del aminoácido.
Ilustraciones adaptadas de Thompson & Thompson Genetics in Medicine, 6th edition; RL Nussbaum, RR McInnes, HF Willard, The Human Genome: Structure and Function of Genes and Chromosomes, Figura 3-6, pág. 20, Copyright 2001, con permiso de Elsevier.
J
K
L
Librería - Librery
Colección desordenada de clones de ADN de un organismo particular cuya relación entre sí puede establecerse mediante un mapeo físico.
Lugar de restricción - Restriction Site
Ubicaciones en una molécula de ADN que contiene secuencias de nucleótidos específicas (4-8 pares de bases de longitud), que son reconocidas por enzimas de restricción.
M
Mapeo génico - Gene mapping
Proceso gracias al cual se determina las posiciones relativas de genes en una molécula de ADN, de un cromosoma o de un plásmido, y de la distancia, dada en unidades de ligamiento o física, que hay entre ellos.
Marcador - Marker
Segmento identificable de ADN (p. ej. RFLP, VNTR, microsatélite) que al presentar una variación amplia entre los individuos, permite analizar su herencia y segregación con los alelos de un gen determinado. Se utiliza en el análisis de ligamiento.
Marcador flanqueante - Flanking marker
Región polimórfica identificable de ADN (es decir, un marcador) localizada en un gen, a diferencia del marcador intragénico, que está localizado dentro de un mismo gen. Los marcadores flanqueantes se utilizan en el análisis de ligamiento para analizar la herencia del gen en cuestión.
Marcador intragénico - Intragenic marker
Región polimórfica identificable de ADN (es decir, un marcador) localizado dentro de la extensión de la secuencia de un gen (es decir, intragénico), a diferencia del marcador flanqueante, que está localizado en uno de los dos lados de un gen. Los marcadores intragénicos se utilizan en el análisis de ligamiento para investigar la herencia del gen en cuestión.
Mecanismo de reparación de bases mal emparejadas - Mismatch repair mechanism (sinónimo: reparación de emparejamientos erróneos)
Sistema de control de la integridad del ADN, en el que intervienen determinados genes, que identifica, escinde y corrige los errores de emparejamiento de las bases durante la replicación del ADN. Las mutaciones producidas en los genes responsables de este mecanismo pueden dar lugar a determinadas enfermedades genéticas y algunos tipos de cáncer.
Megabase, Mb - Megabase
Unidad de longitud para fragmentos de ADN que equivale a 1 millón de nucleótidos (aproximadamente 1 centimorgan, cM).
Metilación - Methylation
Unión de los grupos metilo a las citosinas del ADN. Se relaciona con una transcripción reducida del gen y se considera el mecanismo principal en la inactivación del cromosoma X y en la impronta genómica.
Microsatélite - Microsatellite (sinónimos: ADN satélite, repeticiones cortas en tándem)
Segmentos repetitivos de ADN que comprenden de dos a cinco nucleótidos (repeticiones de dinucleótidos/trinucleótidos/ tetranucleótidos/ pentanucleótidos) dispersos por todo el genoma en las regiones no codificadoras que hay entre o dentro de los genes (intrones), se utilizan frecuentemente como marcadores en el análisis de ligamiento debido a la alta variabilidad natural existente en el número de repeticiones entre los individuos. Estas regiones son propiamente inestables y susceptibles a mutaciones.
Mutación puntual - Point mutation
Alteración en la secuencia del ADN causada por el cambio, inserción o deleción de un único nucleótido.
Mutación sin cambio de pauta de lectura - In-frame mutation
Mutación que no produce un cambio en el marco de lectura del triplete cuando se produce la transcripción de ADN a ARN mensajero. Estas mutaciones, sin embargo, pueden dar lugar a la síntesis de un producto proteico anormal si, al traducirse el ARN a proteína, el cambio de triplete supone un cambio de aminoácido.
N
Nucleótido - Nucleotide
Molécula compuesta por una base nitrogenada (adenina, guanina, timina o citosina en ADN; adenina, guanina, uracilo o citosina en ARN), un grupo fosfato y un azúcar (desoxirribosa en ADN; ribosa en ARN). El ADN y el ARN son polímeros de muchos nucleótidos.
Ñ
Nucleótido - Nucleotide
Molécula compuesta por una base nitrogenada (adenina, guanina, timina o citosina en ADN; adenina, guanina, uracilo o citosina en ARN), un grupo fosfato y un azúcar (desoxirribosa en ADN; ribosa en ARN). El ADN y el ARN son polímeros de muchos nucleótidos.
O
Oligonucleótidos específicos del alelo - Allele-specific oligonucleotide testing (ASO)
Prueba de genética molecular empleada para la detección de una mutación específica por medio de un segmento sintético de ADN de una longitud aproximada de 20 pares de bases (oligonucleótido). El investigador diseña la secuencia específica que se une a la secuencia complementaria en una muestra de ADN problema, lo que permite su identificación.
P
Panel de genes - Multigene panel
Prueba genética en la que se analiza simultáneamente el estado de un grupo de genes asociados con una determinada patología. Son muy usados los que analizan el conjunto de genes asociados con la susceptibilidad a desarrollar cáncer colorrectal, de mama, etc., o los genes relacionados con la discapacidad intelectual. Los avances en las técnicas de secuenciación permiten analizar en un único test un número prácticamente ilimitado de genes. Frente al habitual análisis de un único gen, el uso de estos paneles se ha extendido en los años más recientes tanto en el terreno de la investigación, como en la práctica clínica ya que permiten una valoración potencialmente más completa de los riesgos derivados de la alteración de genes de susceptibilidad.
Par de bases - Base pair: (sinónimo: bp)
Dos bases nitrogenadas unidas mediante enlaces débiles en el ADN de cadena doble; el emparejamiento específico de estas bases (adenina con timina y guanina con citosina) facilita la replicación exacta del ADN; la cuantificación de los pares de bases (p. ej. 8 bp) se refiere a la longitud física de una secuencia de nucleótidos.
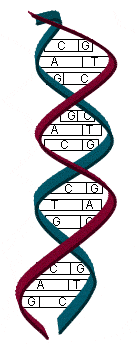
ADN de cadena doble que muestra el emparejamiento específico de los pares de bases G y C, A y T .
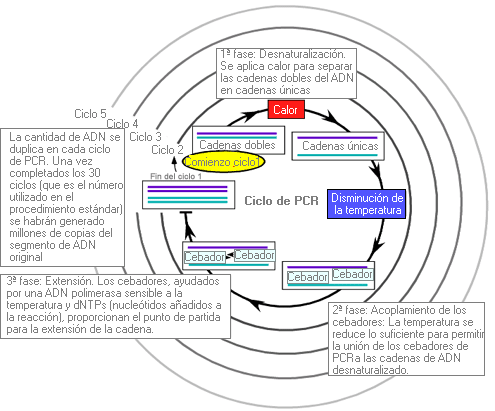
PCR - PCR (Polymerase chain reaction) (sinónimo: reacción en cadena de la polimerasa)
Procedimiento que genera millones de copias de un segmento corto de ADN mediante ciclos repetidos de: 1) desnaturalización del ADN, 2) acoplamiento de los oligonucleótidos y 3) extensión mediante la acción de la ADN polimerasa. La PCR es un procedimiento muy común en los estudios de genética molecular y se puede utilizar para: 1) generar una cantidad suficiente de ADN para realizar determinadas pruebas (p. ej. análisis de la secuencia, rastreo de mutaciones); o 2) puede ser una prueba diagnóstica en sí misma (por ejemplo amplificación específica del alelo, cuantificación del número de repeticiones de trinucleótidos).
PCR cuantitativa - Quantitative PCR (sinónimos: PCR cuantitativa en tiempo real)
Uso de PCR para determinar la cantidad de ADN o ARN en una muestra; se utiliza frecuentemente para detectar mutaciones por deleción y mutaciones por duplicación.
Anteriormente, la PCR cuantitativa determinaba la cantidad de producto (ADN o ARN) tras completar un número específico de ciclos; sin embargo, este procedimiento no es del todo fiable o preciso y se ha sustituido en gran parte por la PCR cuantitativa “en tiempo real” (denominada también “cinética”), en la que se determina la cantidad del producto (ADN o ARN) tras completar cada ciclo de PCR.
PCR cuantitativa en tiempo real
I. Se amplifica la región de ADN o ARN de interés en presencia de una sonda fluorescente; la intensidad de la señal fluorescente generada aumenta en proporción a la cantidad de producto resultante en PCR.
II. La intensidad de la señal fluorescente, que se mide al completar cada ciclo de PCR, se representa en una “gráfica de amplificación” para determinar el CT (del inglés cycle threshold), que se define como el punto en el que se detecta por primera vez la señal fluorescente, es decir, el producto de la PCR.
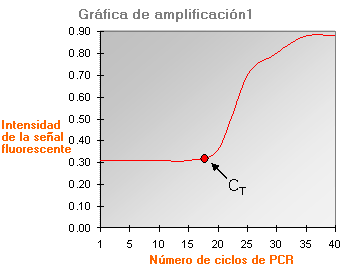
III. Se compara el valor CT frente a una curva estándar (basándose en los valores CT de muestras control y sus respectivas cantidades iniciales de ADN o ARN conocidas) para determinar la cantidad de ADN o ARN en la muestra del paciente. La presencia de una mutación por deleción o duplicación se puede deducir comparando la cantidad de ADN o ARN en la muestra del paciente con la cantidad en una muestra control. Por ejemplo, si la cantidad de ADN o ARN en la muestra del paciente fuese ~50% respecto a la del control normal, esto indicaría una mutación por deleción; si la cantidad de ADN o ARN en la muestra fuese ~150% respecto a la del control normal, indicaría una mutación por duplicación.
1Adaptado de PennState College of Medicine, Quantitative Real-Time PCR
Algunas implicaciones clínicas
Al igual que la PCR, la PCR cuantitativa se ha convertido en una técnica estándar en todos los laboratorios clínicos de genética molecular.
Los análisis para detectar mutaciones por deleción hemicigótica (p. ej. deleciones en DMD en varones afectados por distrofia muscular de Duchenne) se pueden realizar con técnicas de PCR estándar. Sin embargo, se debe realizar PCR cuantitativa, en lugar de estándar, para detectar mutaciones por deleción heterocigótica (p. ej. deleciones heterocigóticas en (1) individuos afectados por un trastorno causado por una deleción en un gen contiguo autosómico, como, por ejemplo, el síndrome de Williams, (2) portadores de un trastorno autosómico recesivo, como la atrofia muscular espinal y (3) mujeres portadoras de un trastorno ligado al cromosoma X, por ejemplo la distrofia muscular de Duchenne).
Pirimidina - Pyrimidine
Es un compuesto orgánico, similar al benceno, y a la piridina pero con dos átomos de nitrógeno que sustituyen al carbono en las posiciones 1 y 3. La pirimidina tiene tres derivados que forman parte de los ácidos nucleicos: la timina, la citosina y el uracilo; las tres tienen un grupo carbonilo (C=O) en el carbono 2; las dos primeras forman parte del ADN donde se aparean con sus purinas complementarias, mientras que la última está presente solo en el ARN. En el ADN, adenina (A) y guanina (G) se emparejan con timina (T) y citosina (C), respectivamente, y en el ARN adenina se une a uracilo (U) y la guanina a citosina.
Plásmido - Plasmid
Molécula pequeña, a menudo circular, de ADN extracromosómico, cuya replicación es independiente de la del ADN cromosómico. Son comunes en bacterias y levaduras. Por lo general contienen un reducido número de genes que suele conferir ciertas ventajas al hospedador, por ejemplo, resistencia a un determinado antibiótico.
Los plásmidos se usan en ingeniería genética aprovechando su capacidad para reproducirse de manera independiente del ADN cromosómico.
Polimorfismo - Polymorphism
Polimorfismo de longitud de fragmentos de restricción - Restriction fragment length polymorphism (sinónimo: RFPL)
Variación natural (polimórfica) de una secuencia de ADN entre los individuos. Se visualiza porque se suprimen o se crean nuevos sitios de restricción (corte) en el ADN, que cuando es digerido por una endonucleasa da lugar a fragmentos de ADN de diferente longitud.
Polimorfismo de nucleótido único - Single Nucleotide Polymorphism o SNP
Variación en la secuencia de ADN que implica una sola base: adenina (A), timina (T), citosina (C) o guanina (G). Son los polimorfismos genéticos más frecuentes y se considera que determinan gran parte de la variabilidad genética entre individuos. Se conocen varios millones de SNP repartidos por todos los cromosomas humanos, aproximadamente 1 cada 300-100 pares de bases. La frecuencia de un SNP en población general se sitúa por encima del 1%.
Diversas técnicas de análisis masivo del genoma se fundamentan en el estudio de miles o millones de SNP que cubren la práctica totalidad del genoma (Genome Wide Association Studies, o GWAS), para buscar la asociación de algunos de ellos con enfermedades concretas, utilizando para ello grandes números tanto de individuos enfermos, como de individuos sanos.
Polimorfismos de conformación de cadenas sencillas - Single-stranded conformational polymorphism (SSCP)
Técnica para el rastreo de mutaciones que identifica segmentos de ADN de cadena sencilla que muestran un patrón de migración anormal en la electroforesis en gel.
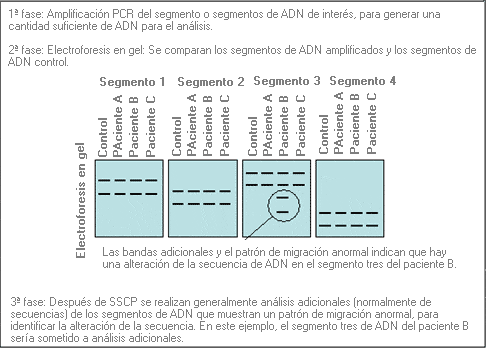
Algunas implicaciones clínicas
La técnica SSCP se puede utilizar cuando las mutaciones están distribuidas por todo el gen.
El SSCP puede comprender el análisis de todo el gen o sólo de regiones específicas.
Potenciador - Enhancer
Pequeña región del ADN eucariota que puede aumentar los niveles de transcripción de un gen o un grupo de genes mediante la unión a proteínas (factores de transcripción). Las regiones enhancer no tienen por qué estar localizadas cerca de los genes sobre los que actúa, ni siquiera en el mismo cromosoma.
Primer - Primer
Producto génico - Gene product
Profase - Prophase
Es la primera fase de la mitosis y de la meiosis. En ella se produce la condensación de todo el material genético (ADN)-que normalmente existe en forma de cromatina condensada dentro de una estructura altamente ordenada llamada cromosoma- y el desarrollo bipolar del huso acromático. Los dos pares de centríolos migran hacia extremos opuestos de la célula, la membrana nuclear se disuelve al final de la profase y los cromosomas se anclan en los microtúbulos del huso gracias al cinetocoro que se encuentra en el centrómero.
Promotor - Promoter
Secuencia de ADN necesaria para activar un gen. La transcripción del gen se inicia en el promotor. Generalmente se encuentran cerca del comienzo de un gen, el promotor tiene un sitio de unión para la enzima ARN polimerasa encargada de la síntesis del ARNm.
Prueba de parentesco - Parentage testing (sinónimos: prueba de maternidad, prueba de paternidad)
Proceso mediante el cual se comparan secuencias de ADN de un niño y un adulto determinados para calcular la probabilidad de que los dos individuos estén emparentados. Mediante los análisis de ADN se puede descartar con seguridad, pero no confirmar absolutamente que un individuo es el padre o la madre biológicos.
Pseudogen - Pseudogene
Copia de un gen que carece normalmente de intrones y de otras secuencias de ADN esenciales para su función. Los pseudogenes, aunque son genéticamente similares al gen funcional original, no se expresan y frecuentemente contienen numerosas mutaciones.
Pseudogenes:
Consisten en secuencias de ADN muy similares a los genes conocidos, pero no son funcionales.
Se encuentran dispersos en el genoma.
Se cree que son subproductos de la evolución, que representan genes rudimentarios que una vez fueron funcionales.
Se supone que han perdido su función original por la introducción de mutaciones en las secuencias codificadoras o reguladoras.
 Supuestos mecanismos por los que se forman los pseudogenes:
Supuestos mecanismos por los que se forman los pseudogenes:
Duplicación de un gen funcional seguida de la introducción de numerosas mutaciones que finalmente dan lugar a su inactivación.
Retrotransposición – proceso mediante el cual se generan una o más copias de ADN a partir del transcripto de ARNm del gen y se vuelven a insertar en el genoma en cualquier localización, no necesariamente cerca del sitio de su gen progenitor. Puesto que se generan a partir de ARNm procesado, estos pseudogenes carecen de intrones.
Puntos calientes de mutación - Hotspot mutation region
Secuencias de adn muy susceptibles a mutaciones debido a una inestabilidad inherente, tendencia hacia un entrecruzamiento desigual o predisposición química a sustituciones de nucleótidos simples; región en la que se observan mutaciones con más frecuencia de la habitual.
Purina - Purine
Es una base nitrogenada, un compuesto orgánico heterocíclico aromático. La estructura de la purina está compuesta por dos anillos fusionados, uno de seis átomos y el otro de cinco. Dos de las bases de los ácidos nucleicos, adenina y guanina, son derivados de una purina. En el ADN, estas bases se unen con sus pirimidinas complementarias, la timina y la citosina, a través de enlaces de hidrógeno.
Q
R
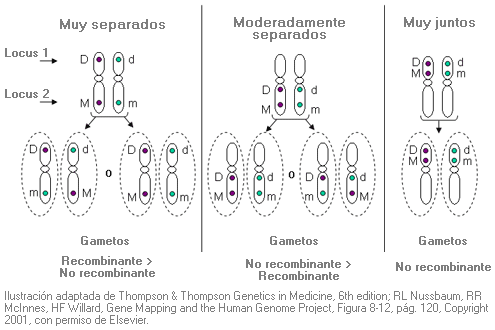
Recombinación - Recombination (sinónimo: sobrecruzamiento)
Intercambio de un segmento de ADN entre dos cromosomas homólogos durante la meiosis (aunque en algunas ocasiones se puede dar en mitosis), cuyo resultado es una combinación nueva de material genético en el gameto.
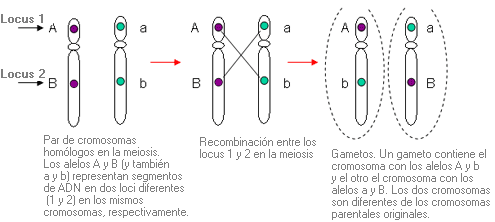 Dos genes que están localizados en el mismo cromosoma pueden segregarse juntos o no. La recombinación entre los cromosomas homólogos durante la meiosis da lugar a combinaciones nuevas de genes en el gameto, y por lo tanto, a descendientes.
Dos genes que están localizados en el mismo cromosoma pueden segregarse juntos o no. La recombinación entre los cromosomas homólogos durante la meiosis da lugar a combinaciones nuevas de genes en el gameto, y por lo tanto, a descendientes.
A través del proceso de recombinación, cada cromosoma transmitido de un padre a su hijo contiene algunos segmentos heredados del abuelo y otros de la abuela de ese niño.
Fuente: Elsevier.
La recombinación desempeña un papel importante para asegurar la variabilidad genética entre los individuos.
El comportamiento durante la meiosis de los alelos en dos loci (1 y 2) del mismo cromosoma depende de su proximidad.
Región de puntos calientes de mutación - Hotspot mutation region
Secuencias de ADN muy susceptibles de ser mutadas debido a una inestabilidad inherente, tendencia hacia un entrecruzamiento desigual o predisposición química a sustituciones de nucleótidos simples; región en la que se observan mutaciones con más frecuencia de la habitual.
Región subtelomérica - Subtelomeric region
Región cromosómica más cercana al telómero (cada uno de los extremos del romosoma) que está compuesta por secuencias repetitivas de ADN muy polimórficas, situadas típicamente en las proximidades de las áreas ricas en genes. Las microdeleciones y las reordenamientos sutiles que alteran los genes en las regiones subteloméricas pueden causar retraso mental; para detectar estas anomalías normalmente es necesario utilizar hibridación fluorescente in situ (FISH) o arrays de CGH, que permiten una evaluación más detallada de las regiones subteloméricas.
Repeticiones en tándem de número variable - Variable number tandem repeats (sinónimo: VNTR)
Ordenación lineal de múltiples copias de secuencias cortas repetidas de ADN, de longitud variable pero mayores de tres bases, y muy polimórficas, por lo que resultan útiles como marcadores en el análisis de ligamiento.
Replicación del ADN - DNA replication
La replicación del ADN es el proceso mediante el cual se duplica una molécula (sintetiza una copia idéntica) de ADN. Cuando una célula se divide, en primer lugar, debe duplicar su genoma para que cada célula hija contenga un juego completo de cromosomas. De esta manera se garantiza la transmisión de la información genética.
S
Secuenciación del ADN - DNA sequencing
Es el conjunto de métodos que tienen por objetivo determinar el orden exacto de los nucleótidos (A, C, G y T) en la molécula de ADN. La secuencia de bases de ADN lleva la información que una célula necesita para originar moléculas de ARN y proteínas. Los avances en las técnicas de secuenciación de ADN han sido muy notables en los años recientes gracias al Proyecto Genoma Humano. Es posible aplicar estas técnicas para conocer la secuencia de un único gen, de varios genes (paneles), al conjunto de genes de un individuo (exoma) o, finalmente a todo el genoma de un individuo.
El método de secuenciación del ADN desarrollado por Frederick Sanger y colaboradores en 1977 ha sido el de uso más extendido en los últimos 40 años. En los años más recientes ese método está siendo sustituido por la secuenciación de nueva generación (“next-generation sequencing”, NGS, o los términos sinónimos “massively parallel” o “deep sequencing”) que ha revolucionado el mundo de la investigación biomédica al ser capaz de analizar simultáneamente millones de pequeños fragmentos de ADN. La tecnología de NGS permite disponer en horas de la secuencia del exoma o del genoma humano completo.
Síndrome de microdeleción - Microdeletion syndrome (sinónimo: síndrome de deleción de genes contiguos)
Síndrome causado por una deleción cromosómica que comprende varios genes y es demasiado pequeña para poder ser detectada al microscopio utilizando métodos citogenéticos convencionales. Dependiendo del tamaño de la deleción, se pueden utilizar ocasionalmente otras técnicas, tales como FISH u otros métodos de análisis de ADN, para su identificación.
Sitio de restricción - Restriction site
Secuencia de ADN que es reconocida por una endonucleasa (una proteína que corta el ADN) como el lugar en el que se cortará el ADN, o también llamado "diana de corte".
SNP - Single Nucleotide Polimorphism
Variación en la secuencia del adn (con respecto a una secuencia consenso) que afecta a una única base (A,T,G,C) en posiciones concretas del genoma (1 cada 1000 bases en promedio), y que se observa en la población con una frecuencia de al menos 1%. En la especie humana se estima que existen en torno a de 12 millones de SNPs, de los que a finales de 2007 se han identificado algo más de un tercio.
Sobrecruzamiento desigual - Unequal crossing over
Emparejamiento incorrecto e intercambio de ADN entre regiones de cromosomas homólogos o no homólogos, pero genéticamente similares, que da lugar a la duplicación o deleción del ADN en las células hijas.
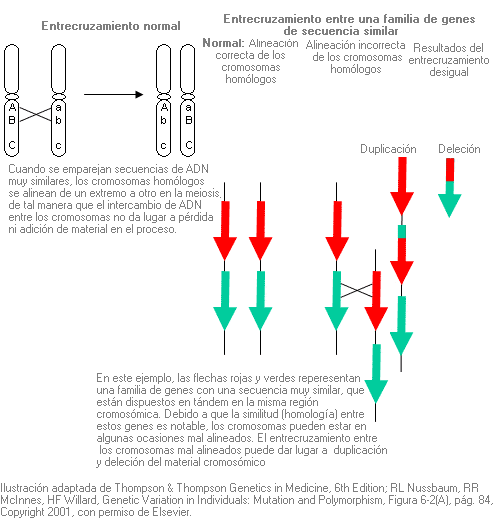 Situaciones más susceptibles a un entrecruzamiento desigual:
Situaciones más susceptibles a un entrecruzamiento desigual:
Miembros de “familias” multigénicas cuyos genes comparten una homología significativa entre sí (p. ej. grupo de genes de alfaglobina; grupo de genes de pigmentos visuales rojos y verdes).
Cuando un gen y un pseudogen están dispuestos en tándem en un cromosoma (p. ej. CYP21 y su pseudogen CYP21P; SMN1y SMN2).
Cuando hay secuencias similares a lo largo de la misma cadena de ADN que predisponen a inversiones o deleciones (p. ej. el gen F8; síndromes de genes contiguos, tales como deleción en 22q11.2).
Cuando hay secuencias no codificadoras homólogas (p. ej. familia Alu de secuencias repetidas de ADN dispersas en todo el genoma).
Sonda - Probe
Southern blot - Southern blot: (sinónimos: Southern blotting, análisis de Southern blotting)
Técnica de análisis de genética molecular que se utiliza para detectar diferencias en la longitud de los fragmentos de ADN tras ser digeridos por enzimas de restricción (lo que se conoce normalmente como RFLP: polimorfismos de longitud de fragmentos de restricción). La longitud de los fragmentos de restricción es variable en tamaño, desde pocas bases a kilobases.
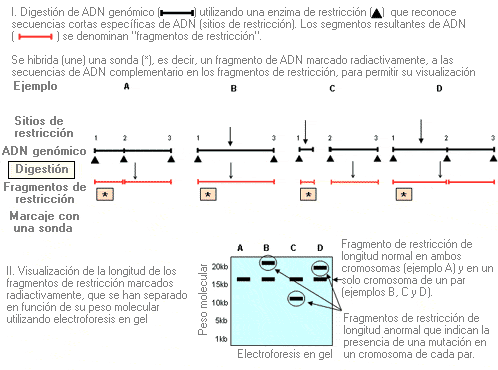
Sustitución - Substitution
Es el cambio o sustitución de una base por otra en la secuencia de ADN. Se conoce como “transición” cuando la base cambia por otra del mismo tipo: purina (A y G) por purina o pirimidina (C y T) por pirimidina. En la “transversión” el cambio es de una purina por una pirimidina o de una pirimidina por una purina.
T
Tasa de mutación - Mutation rate
La tasa de mutación de un gen o una secuencia de ADN es la frecuencia en la que se producen nuevas mutaciones en ese gen o la secuencia en cada generación.
Telofase - Telophase
Es la cuarta y última fase de la mitosis celular. Es la reversión de los procesos que tuvieron lugar durante la profase y prometafase. Los cromosomas ya están separados en dos grupos de cromátidas en los dos extremos de la célula. Las cromátidas se desenrollan y el ADN vuelve a tomar forma de hilos de cromatina difusa. Se crea alrededor de cada grupo una membrana nuclear. El huso mitótico desaparece.
Telómero - Telomere
Los telómeros son los extremos de los cromosomas. Son regiones de ADN no codificante, altamente repetitivas (5'-TTAGGG-3') cuya función principal es la estabilidad estructural de los cromosomas en las células eucariotas, la división celular y el tiempo de vida de las estirpes celulares.
Temperatura de melting (Tm) - Melting Temperature
También llamada temperatura de fusión del ADN es la temperatura en la cual el 50% del ADN tiene sus hebras separadas (desnaturalización del ADN).
Templado (hebra) - Template (strand)
Hebra del ADN dúplex que actúa como molde para la transcripción.
Termociclador - Thermocycler
También conocido como máquina de PCR es un instrumento usado en biología molecular que permite realizar de forma automatizada y secuencial los ciclos de temperaturas necesarios para llevar a cabo la reacción en cadena de la polimerasa (PCR) de amplificación de ADN.
Timina - Thymine
Es una de las cinco bases nitrogenadas constituyentes de los ácidos nucleicos (las otras cuatro son la adenina, la guanina, la citosina, y el uracilo, este último sólo presente en el ARN). Es un compuesto heterocíclico derivado de la pirimidina que parte del ADN y en el código genético se representa con la letra T.
Transcripción - Transcription
Es la síntesis enzimática de ARN utilizando un templado de ADN. Es el primer proceso de la expresión génica donde se transfiere la información contenida en la secuencia del ADN hacia la secuencia de la proteína utilizando diversos ARN como intermediarios. Esta secuencia de ADN es copiada a ARN con una enzima llamada ARN polimerasa que sintetiza ARN mensajero que contiene la información de la secuencia del ADN. Este proceso también podría llamarse síntesis del ARN mensajero.
Transcriptasa Inversa - Reverse transcriptase
También llamada retrotranscriptasa es una enzima de tipo ADN-polimerasa, codificada por retrovirus, cuya función es sintetizar ADN de doble cadena utilizando como templado una molécula de ARN monocatenario, es decir, catalizar la retrotranscripción o transcripción inversa.
Transfección - Transfection
Término general utilizado para referirse a la introducción de ADN exógeno al interior de una célula eucariota.
Transferencia genética - Gene transfer
Transferencia de material genético, que puede comprender desde un pequeño segmento de adn hasta un genoma entero, de una célula humana a otro tipo de célula en cultivo para estudiar la frecuencia con que los marcadores genéticos conocidos son transferidos conjuntamente al genoma receptor; se utiliza para determinar la proximidad física de los marcadores genéticos en el genoma humano; se utiliza asimismo para estudiar la expresión y regulación génica.
Transgénico - Transgenic
Célula u organismo que contiene en su línea germinal un ADN exógeno introducido experimentalmente.
Transposon (Tn) -
Son regiones de ADN (200-2000 pares de bases) capaces de moverse de manera autosuficiente de una parte a otra parte del genoma, mediante un mecanismo de corta-pega y cuya consecuencia es la producción de mutaciones.
U
Unidad transcripcional - Transcriptional unit
Es un concepto muy amplio que engloba a cualquier segmento de ADN transcrito a un único ARN, independiente del número de genes de los que contenga información.
Uracilo - Uracil
Es una pirimidina, una de las cuatro bases nitrogenadas que forman parte del ARN y en el código genético se representa con la letra U. El uracilo reemplaza en el ARN a la timina que es una de las cuatro bases nitrogenadas que forman el ADN. Al igual que la timina, el uracilo siempre se empareja con la adenina mediante dos puentes de hidrógeno, pero le falta el grupo metilo.
V
Valina - Valine
La valina (abreviada Val o V) es uno de los veinte aminoácidos codificados por el ADN. En el ARN mensajero, está codificada por GUA, GUG, GUU o GUC. Nutricionalmente, en humanos, es uno de los aminoácidos esenciales.
Valor C - C Value
La cantidad de ADN por genoma haploide (un solo juego cromosómico), en el caso de los organismos diploides como el ser humano, el valor C es la cantidad correspondiente a un juego de 23 cromosomas.
Virus - Virus
Agente infeccioso que posee material genético en forma de ADN o ARN con una envuelta proteica y que es incapaz de llevar a cabo actividades metabólicas o reproducirse por sí solo, teniendo que invadir una célula huésped (procarionte, eucarionte vegetal, eucarionte animal) para obtener la energía para sobrevivir.
W
X
Y
YAC (Yeast Artificial Chromosome) -
Vector de clonamiento en la forma de un cromosoma artificial de levadura. Es un vector que pretende imitar las características de un cromosoma normal de una levadura (eucariota), portando un centrómero y telómeros terminales. Esto permite clonar en levaduras, largas secuencias de ADN eucarionte (200 kb - 3.000 kb).
Z
Zipper de Leucina - Leucine zipper
También llamado cremallera de leucina es un motivo supersecundario de proteínas que consta de dos segmentos peptídicos en hélice alfa. En un extremo, estas hélices interaccionan entre sí gracias a la interacción entre cadenas laterales de leucina y en el otro lo hacen con secuencias específicas de bases en el surco mayor del ADN. Es un dominio de dimerización de expresión, en particular en proteínas de la clase factores de transcripción.