- Todos
- A
- B
- C
- D
- E
- F
- G
- H
- I
- J
- K
- L
- M
- N
- Ñ
- O
- P
- Q
- R
- S
- T
- U
- V
- W
- X
- Y
- Z
A
Alelo - Allele
Un alelo es cada una de las dos o más versiones de un gen. Un individuo hereda dos alelos para cada gen, uno del padre y el otro de la madre. Los alelos se encuentran en la misma posición dentro de los cromosomas homólogos. Si los dos alelos son idénticos, el individuo es homocigoto para este gen.
Tipos de alelos denominados según su función: En la figura siguiente se muestran las proporciones aproximadas de los alelos en un locus genético hipotético, en una población determinada. Nota: Las mutaciones patológicas, los polimorfismos y las variantes alélicas de significación desconocida se consideran alelos “mutantes” en todos los casos.
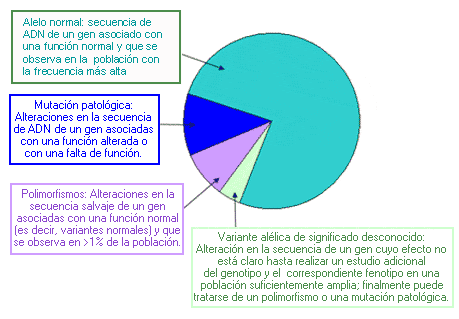
- Alelo materno: Heredado de la madre
- Alelo paterno: Heredado del padre
Los individuos pueden ser:
- Homocigóticos: cuando portan los alelos del gen iguales, cada uno procedente de un progenitor.
- Heterocigóticos: cuando los dos alelos del gen son diferentes.
B
C
Coeficiente de endogamia (F) - Coefficient of imbreeding
Probabilidad de que un individuo homocigoto en un locus determinado haya recibido ambos alelos del mismo ancestro (alelos idénticos por descendencia-identical by descent).
D
E
Expresividad variable - Variable expressivity
Variación de las manifestaciones clínicas (tipo y severidad) de un trastorno genético entre individuos afectados, incluso de la misma familia.
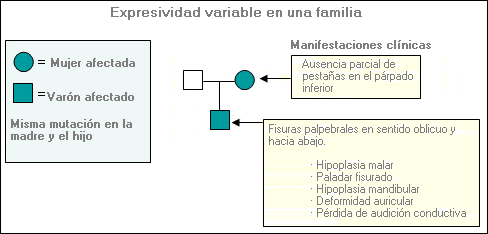
Síndrome de Treacher Collins como ejemplo de expresividad variable
Patrón de herencia: Autosómica dominante Gen: TCOF1 Locus: 5q32-q33.1
Manifestaciones clínicas principales:
Fisuras palpebrales en sentido oblicuo y hacia abajo
Coloboma palpebral
Ausencia parcial o total de pestañas en el párpado inferior
Hipoplasia malar
Paladar fisurado
Hipoplasia mandibular
Deformidades auriculares
Pérdida de audición
Posibles explicaciones para la expresividad variable:
Diferentes mutaciones en el mismo gen (heterogeneidad alélica)
Diferentes mutaciones en varios genes en diferentes locus (heterogeneidad genética)
Existencia de genes modificadores (otros genes que afectan a la expresión del gen de interés)
Factores medioambientales
Factores desconocidos
¿Cuál es la diferencia entre expresividad variable y penetrancia?
La expresividad variable se refiere a la diversidad de manifestaciones clínicas observadas en individuos que presentan un trastorno determinado. El término expresividad variable se aplica a los trastornos que siguen todos los patrones de herencia.
La penetrancia es la proporción de individuos con una mutación patológica que presentan manifestaciones clínicas del trastorno. El término penetrancia se aplica normalmente a los trastornos autosómicos dominantes.
¿De qué forma influye la expresividad variable en la penetrancia?
La penetrancia depende de las manifestaciones de la enfermedad que se utilizan para determinar si un individuo está afectado. Por ejemplo, se puede suponer que los individuos con manifestaciones leves de un trastorno autosómico dominante no están afectados, por lo que la penetrancia reducida del trastorno es aparente. Consideremos lo siguiente:
Se sabe que una paciente presenta una mutación en FBN1 causante del síndrome de Marfan porque tanto su padre como su hijo están afectados. Los resultados de la exploración física de esta paciente son normales y se cree que no presenta manifestaciones del síndrome de Marfan. Sin embargo, el ecocardiograma revela dilatación de la raíz aórtica, que es una manifestación clínica característica del síndrome de Marfan. Los estudios basados en los resultados de la exploración física y el ecocardiograma revelarán una penetrancia más alta del síndrome de Marfan que los estudios basados sólo en la exploración física.
F
G
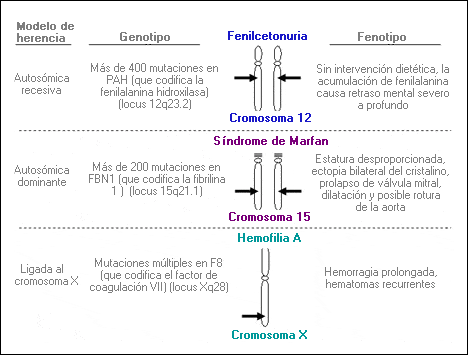
Genotipo - Genotype
Constitución genética de un organismo o célula; se refiere también al grupo específico de alelos heredados en un locus.
H
Haploinsuficiencia - Haploinsufficiency
Situación en la que un individuo está clínicamente afectado porque una sola copia del gen normal es incapaz de producir proteína en cantidad o calidad suficiente para asegurar la función normal. Puede deberse a que el individuo es heterocigótico para determinada mutación génica o es hemicigótico en un locus particular, debido normalmente a una deleción del alelo correspondiente.
Haplotipo - Haplotype
Conjunto de alelos contenidos en un locus (o en varios loci) de una misma dotación haploide. El haplotipo podemos referirlo a un solo locus o a un genoma completo pero siempre se refiere a uno de los dos alelos de cada gen.
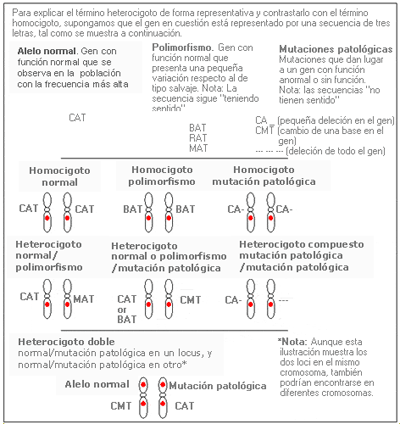
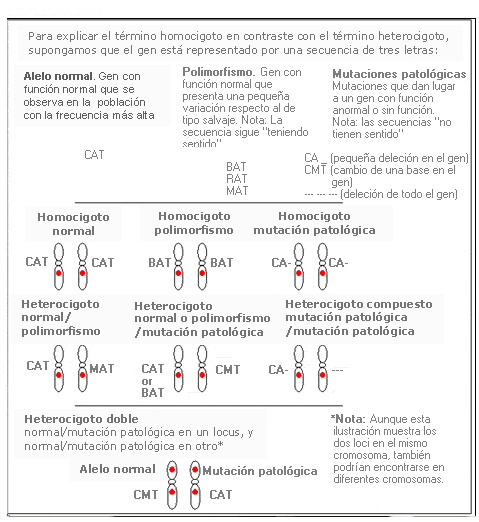
Heterocigoto - Heterozygote
Individuo que tiene dos alelos diferentes en un locus, uno en cada cromosoma. En el caso de una mutación patológica, un alelo es normal y otro anormal.
Heterocigoto compuesto: Individuo que tiene dos alelos anormales o mutados diferentes en un mismo locus, uno en cada cromosoma; se refiere normalmente a los individuos afectados por una enfermedad autosómica recesiva.
Heterocigoto doble: Individuo que presenta dos mutaciones génicas diferentes (es decir, es heterocigótico) en dos loci genéticos distintos.
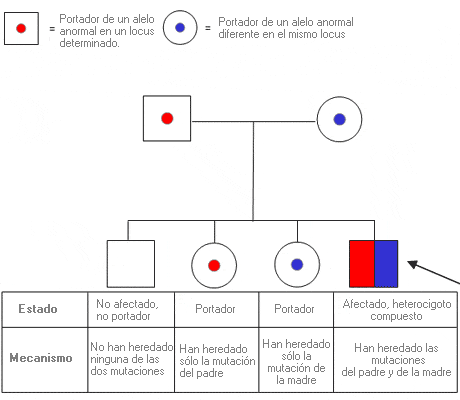
Heterocigoto compuesto - Compound heterozygote
Individuo que tiene dos alelos anormales o mutados diferentes en un mismo locus, uno en cada cromosoma; normalmente se refiere a los individuos afectados por una enfermedad autosómica recesiva.
Los hijos/as afectados de padres portadores de una mutación diferente en el mismo locus del gen son heterocigotos compuestos.
Heterocigoto doble - Double heterozygote
Individuo que es heterocigoto para mutación en dos loci genéticos diferentes. Es decir, es portador de dos mutaciones en genes distintos.
Cuando el padre y la madre presentan una mutación en dos loci genéticos diferentes, sus hijos pueden ser heterocigotos dobles. Los loci pueden estar en el mismo cromosoma o en diferentes cromosomas, según se muestra a continuación.
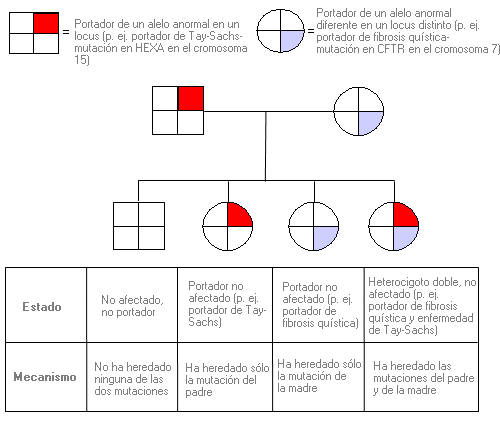
Nota: Puesto que en el ejemplo anterior se incluyen dos loci diferentes (dos alelos para cada uno), en cada hijo/a pueden darse 16 combinaciones posibles de los cuatro alelos de los padres.
Heterogeneidad alélica - Allelic heterogeneity (sinónimo: heterogeneidad molecular)
Heterogeneidad de locus - Locus heterogeneity
Situación en que las mutaciones producidas en Genes que se encuentran en diferentes loci cromosómicos dan lugar al mismo Fenotipo.
Ejemplo de heterogeneidad de locus: Retinitis pigmentosa (RP)
Fenotipo: Las anomalías de los fotorreceptores (bastoncillos y conos) de la retina dan lugar a una adaptación inadecuada a la oscuridad, causan pérdida de visión periférica y finalmente pérdida progresiva de visión.
Loci: Las mutaciones producidas en los genes en más de 35 loci diferentes dan lugar a retinitis pigmentosa no sindrómica que es clínicamnete indistinguible, por lo que no es posible deducir el gen alterado basándose sólo en el exámen ocular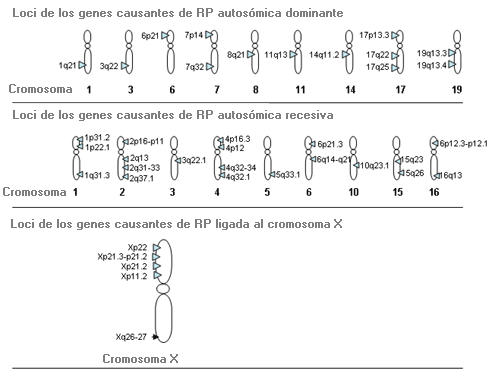
Comparar y contrastar el concepto de heterogeneidad de locus con lo siguiente:
Heterogeneidad alélica: Un solo fenotipo originado por diferentes mutaciones en el mismo gen y locus cromosómico (Nota: algunos trastornos muestran heterogeneidad de locus, otros heterogeneidad alélica y otros ambas características)
Expresividad: Variación de las manifestaciones clínicas (tipo y severidad) de una enfermedad genética entre individuos que presentan la misma alteración genética, incluso dentro de la misma familia.
(Nota: La heterogeneidad de locus implica que los individuos con diferentes genotipos tienen el mismo fenotipo; mientras que la expresividad variable implica que los individuos con el mismo genotipo presenten un fenotipo que muestra diferencias tanto en la severidad como en la edad de comienzo de sus manifestaciones).
Heterogeneidad genética - Locus heterogeneity
Situación en que las mutaciones producidas en genes distintos dan lugar al mismo fenotipo.
Fenotipo: Las anomalías de los fotorreceptores (bastoncillos y conos) de la retina dan lugar a una adaptación inadecuada a la oscuridad, causan pérdida de visión periférica y finalmente pérdida progresiva de visión.
Loci implicados: mutaciones producidas en más de 35 genes diferentes dan lugar a retinitis pigmentosa no sindrómica, que es clínicamente indistinguible. A esto se debe que no sea posible deducir qué gen es el alterado basándose sólo en el examen ocular.
Heterogeneidad alélica: Un solo fenotipo originado por diferentes mutaciones en el mismo gen y locus cromosómico. (Nota: algunos trastornos muestran heterogeneidad genética, otros heterogeneidad alélica y otros ambas características).
Expresividad variable: variación de las manifestaciones clínicas (tipo y severidad) de una enfermedad genética entre individuos que presentan la misma alteración genética, incluso dentro de la misma familia.
(Nota: La heterogeneidad genética implica que los individuos con diferentes genotipos tienen el mismo fenotipo, mientras que la expresividad variable implica que los individuos con el mismo genotipo presenten un fenotipo que muestra diferencias tanto en la severidad como en la edad de comienzo de sus manifestaciones).
Homocigoto - Homozygote
Individuo que tiene dos alelos idénticos en un mismo locus determinado, uno en cada cromosoma.
I
Informatividad - Informativeness
En relación a un marcador genético, un descendiente de una pareja será informativo cuando la probabilidad de que se pueda deducir el origen parental de cada uno de los alelos de un locus en concreto es alta.
Integración - Integration
Proceso de recombinación que inserta una molécula de ADN pequeña en un locus más grande.
J
K
L
Locus - Locus (plural: loci)
Lugar o localización física de un gen específico en un cromosoma
M
N
Nombre del locus - Locus name
Abreviatura asignada de manera informal que se utiliza en el proceso de mapeado para denominar un supuesto gen antes de que sea identificado. Una vez que el gen es identificado, el nombre del locus se sustituye generalmente por el símbolo genético asignado formalmente (que suele diferir del nombre del locus).
Ñ
Nombre del locus - Locus name
Abreviatura asignada de manera informal que se utiliza en el proceso de mapeado para denominar un supuesto gen antes de que sea identificado. Una vez que el gen es identificado, el nombre del locus se sustituye generalmente por el símbolo genético asignado formalmente (que suele diferir del nombre del locus).
O
P
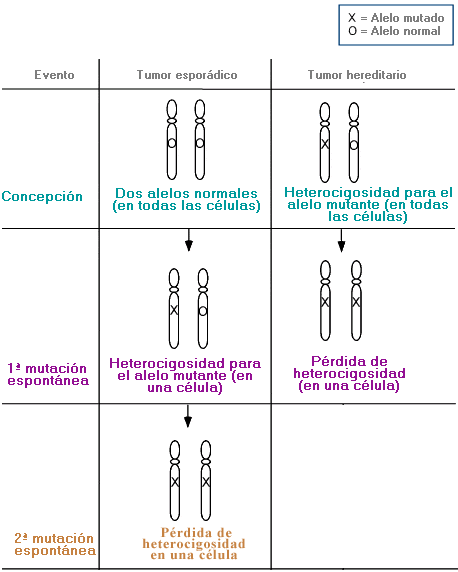
Pérdida de heterocigosidad - Loss of heterozygosity (LOH, de sus siglas en inglés)
Cuando en un locus determinado, que es heterocigoto para un alelo mutante deletéreo y un alelo normal, se produce una deleción, u otro evento mutacional, en el alelo normal que convierte a la célula bien en hemicigótica (es decir, con un único alelo deletéreo), bien en homocigótica para el alelo deletéreo.
La LOH es el mecanismo en el que se basa el desarrollo de algunos tipos de cáncer en los que una sola mutación en uno de los homólogos de un gen supresor tumoral no es suficiente para iniciar el crecimiento del tumor. Sin embargo, la deleción o inactivación del segundo alelo en el cromosoma homólogo, da lugar a un crecimiento celular incontrolado.
La LOH puede ser el fundamento del desarrollo del cáncer en tumores esporádicos y hereditarios. En los tumores esporádicos, los dos alelos son normales en el momento de la concepción, mientras que en los tumores hereditarios hay un alelo mutante en el momento de la concepción.
Portador - Carrier
Portador sintomático - Manifesting carrier
Individuo que en un locus determinado presenta un alelo recesivo patológico en un cromosoma y un alelo normal en el otro cromosoma y que manifiesta algunos síntomas de la enfermedad. Este término se usa con frecuencia para portadoras de una mutación recesiva ligada al cromosoma X que manifiestan signos de la enfermedad, aunque su fenotipo suele ser menos severo que el de los varones con la misma mutación.
Aunque con algunas excepciones, los portadores de una mutación patológica recesiva por regla general no manifiestan síntomas clínicos de la enfermedad. Sin embargo, los portadores de mutaciones recesivas ligadas al cromosoma X presentan ocasionalmente algunas manifestaciones clínicas de la enfermedad, por lo que se les denomina portadores sintomáticos. Se supone que el mecanismo subyacente en los portadores sintomáticos es una inactivación sesgada o no aleatoria del cromosoma X, de tal manera que en la mayoría de las células el cromosoma X activo es el que tiene la mutación.
Ejemplos de enfermedades ligadas al cromosoma X en las que son frecuentes las portadoras sintomáticas:
Hemofilia A: Tiempo de coagulación prolongado, hemorragia excesiva durante intervenciones quirúrgicas menores y menstruaciones abundantes.
Distrofia muscular de Duchenne: Debilidad muscular, hipertrofia de pantorrillas leve y/o miocardiopatía dilatada.
Síndrome X frágil (mutación con penetrancia completa): Retraso mental.
Q
R
S
Segregación - Segregation
Separación de los cromosomas homólogos y su distribución aleatoria en los diferentes gametos en la meiosis. Se aplica también a la separación de los alelos de un locus y su distribución en las células hijas que se produce durante la meiosis o en la mitosis.
Símbolo genético - Gene symbol
Abreviatura exclusiva del nombre de un gen, compuesta por letras del alfabeto latino mayúsculas y en cursiva y por números arábigos, que es asignada formalmente por el HUGO Gene Nomenclature Committee una vez identificado un gen (Nota: los posibles genes pueden ser denominados por su nombre de locus antes de que sean identificados).
La definición siguiente está adaptada de Genew: The Human Gene Nomenclature Database (www.gene.ucl.ac.uk/nomenclature/):
El “símbolo genético” debe estar compuesto preferentemente por un máximo de seis caracteres. Se pretende que el “nombre genético o nombre completo del gen” que es descriptivo y más largo, sea conciso y exprese el carácter o la función del gen. La primera letra del símbolo genético debe ser la misma que la del nombre del gen, con objeto de facilitar la ordenación y clasificación alfabética, p. ej. el gen denominado “cáncer de mama, comienzo precoz 1” tiene el símbolo “BRCA1”. Cada gen humano conocido tiene un nombre y símbolo genético aprobado por el comité HUGO. Todos los símbolos aprobados están incluidos en la base de datos Human Gene Nomenclature de Genew. Cada símbolo es único y se asegura que se proporciona a cada gen sólo un símbolo aprobado. Es necesario proporcionar un símbolo único a cada gen de manera que todos podamos hablar de ellos sabiendo de lo que se trata, esto también facilita la recuperación de datos de publicaciones por medios electrónicos. Preferentemente cada símbolo mantiene una estructura paralela en los diferentes miembros de una familia de genes y pueden ser utilizados también en otras especies, en particular el ratón. El HUGO ha aprobado ya los símbolos de más de 13.000 genes, lo que representa la tercera parte del total de los genes humanos, que se estiman en 30.000. Los científicos y responsables de publicaciones científicas (p. ej. Genomics, Nature Genetics and Cytogenetics and Cell Genetics) y bases de datos (p. ej. RefSeq, OMIM, GDB, MGD y LocusLink) siguen solicitando símbolos nuevos individuales. También hay una demanda de grupos de símbolos nuevos por parte de los que investigan familias de genes, segmentos de cromosomas o cromosomas enteros. Puesto que el análisis de la secuencia del genoma humano está casi completo, hay una demanda creciente por la aprobación rápida de símbolos génicos. Sin embargo, en todos los casos se están realizando esfuerzos considerables por utilizar un símbolo aceptable por todos aquellos que trabajan en este campo.