- Todos
- A
- B
- C
- D
- E
- F
- G
- H
- I
- J
- K
- L
- M
- N
- Ñ
- O
- P
- Q
- R
- S
- T
- U
- V
- W
- X
- Y
- Z
A
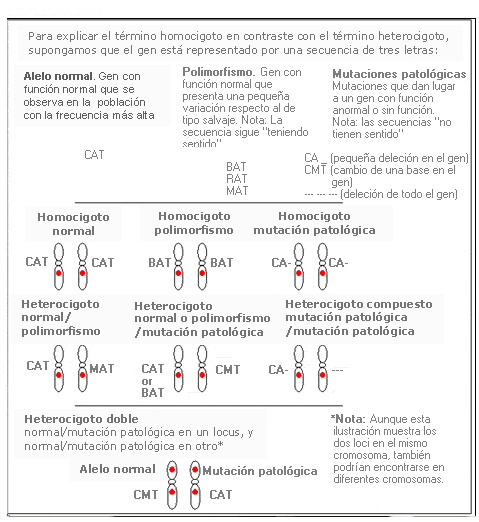
Alelo - Allele
Un alelo es cada una de las dos o más versiones de un gen. Un individuo hereda dos alelos para cada gen, uno del padre y el otro de la madre. Los alelos se encuentran en la misma posición dentro de los cromosomas homólogos. Si los dos alelos son idénticos, el individuo es homocigoto para este gen.
Tipos de alelos denominados según su función: En la figura siguiente se muestran las proporciones aproximadas de los alelos en un locus genético hipotético, en una población determinada. Nota: Las mutaciones patológicas, los polimorfismos y las variantes alélicas de significación desconocida se consideran alelos “mutantes” en todos los casos.
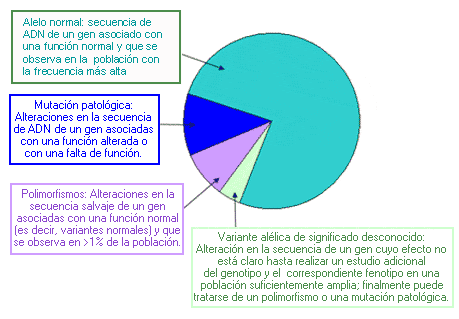
- Alelo materno: Heredado de la madre
- Alelo paterno: Heredado del padre
Los individuos pueden ser:
- Homocigóticos: cuando portan los alelos del gen iguales, cada uno procedente de un progenitor.
- Heterocigóticos: cuando los dos alelos del gen son diferentes.
Alelo de penetrancia completa - Full penetrance allele
Alelo que tiene una repercusión fenotípica en todos los individuos que son portadores de ese alelo.
Alelo de penetrancia incompleta (o reducida) - Incomplete (or reduced) penetrance allele
Alelo que no tiene una repercusión fenotípica en todos los individuos que son portadores, tan solo en una porción de ellos.
Alelo normal - Wild type alelle
El asociado al fenotipo normal, el más común en la población. Opuesto a alelo mutante. También llamado “alelo salvaje” o alelo silvestre”.
Alelo nulo - Null allele
Alelo que da lugar bien a la ausencia de producto génico, o a un producto sin actividad. Una deleción del gen es necesariamente un alelo nulo.
Alelo premutado - Mutable normal allele (sinónimos: alelo intermedio, premutación)
En trastornos causados por expansión de repeticiones de nucleótidos, alelo que no está asociado con síntomas clínicos, pero puede expandirse hasta convertirse en un alelo de gran tamaño con repercusión clínica. También denominado “premutación” o “alelo intermedio”.
En trastornos causados por expansión de repeticiones de nucleótidos, el alelo permutado no está asociado con síntomas clínicos. Su tamaño es superior al del alelo normal y puede expandirse cuando es transmitido a los descendientes, transformándose en una alelo mutado.
De www.acmg.net/Pages/ACMG_Activities/stds-2002/HD.htm (con acceso 17-12-03)
Análisis mutacional específico - Targeted mutation analysis (sinónimo: análisis mutacional dirigido)
Pruebas para detectar la presencia de una mutación específica (p. ej. Glu6Val en la Anemia de Células Falciformes), un tipo de mutación específica (p. ej. la expansión de trinucleótidos repetidos asociada a la Ataxia Espinocerebelosa tipo 1, deleciones asociadas a la Distrofia Muscular de Duchenne) o un conjunto de mutaciones (p. ej. un grupo de mutaciones en la Fibrosis Quística). Difiere de la secuenciación génica completa o el rastreo de mutaciones, en que éstos detectan la mayoría de las mutaciones en la región investigada.
El análisis mutacional se utiliza en las dos situaciones siguientes:
I. Estudio de enfermedades en las que:
La mayoría o todos los individuos afectados presentan la misma mutación (p. ej. Anemia de Células Falciformes).
La mayoría o todos los individuos afectados presentan el mismo tipo de mutación (p. ej. expansión de repeticiones de trinucleótidos).
Un grupo de mutaciones son las más comunes y explican un elevado porcentaje de los casos (véase Tabla).

II. Estudio de los miembros a riesgo de una familia tras haber identificado la mutación patológica específica de la familia en un pariente afectado. Por ejemplo, si en el análisis de la secuencia se identifica una mutación patológica en un miembro afectado de la familia, todos los familiares a riesgo deben ser sometidos sólo a un análisis mutacional dirigido a detectar esa mutación específica de la familia.
Algunas implicaciones clínicas
El análisis mutacional no es generalmente el primer estudio de genética molecular que se utiliza para la identificación de mutaciones en enfermedades que se sabe están asociadas con una proporción alta de mutaciones raras (por ejemplo, específicas de una familia).
Si en un individuo afectado no se puede detectar una mutación en el análisis mutacional, puede que sea necesario realizar un análisis de la secuencia o un rastreo de otras mutaciones para identificarla.
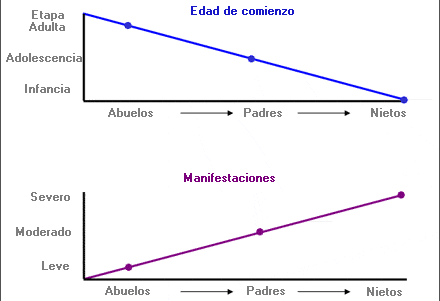
Anticipación - Anticipation
En determinadas enfermedades genéticas, tendencia a que los síntomas de una patología se presenten a edades más tempranas y/o sean más graves que en las generaciones anteriores. Se observa frecuentemente en enfermedades causadas por la expansión de trinucleótidos repetidos que tiende a aumentar de tamaño y a tener un efecto más significativo cuando se transmite de una generación a la siguiente.
ASO -
Asociación - Association
En epidemiología genética, situación en la que un alelo se encuentra más o menos representado en un grupo de individuos afectos de lo que se esperaría por su frecuencia en la población general.
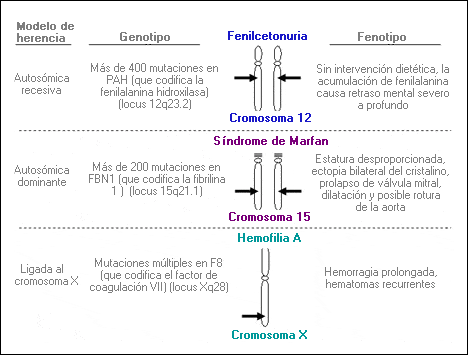
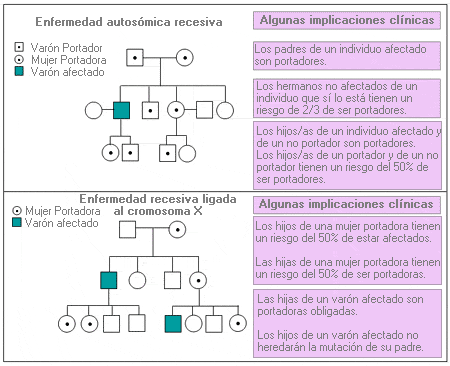
Autosómico dominante - Autosomal dominant
Término que se utiliza para describir un rasgo o patología asociados a un determinado alelo, que están presentes en todos los individuos que han heredado una sola copia de dicho alelo (heterocigotos). Se refiere específicamente a un gen de uno de los 22 pares de autosomas.
Arbol genealógico que representa el patrón de herencia autosómica dominante.
Probabilidad de que una mutación de un gen autosómico dominante sea transmitida a los descendientes.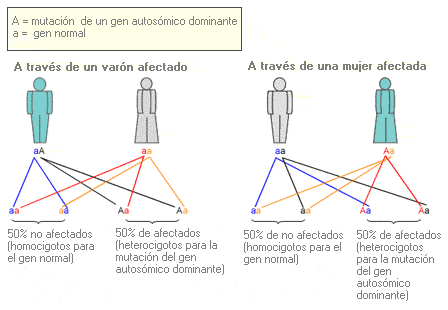
Autosómico recesivo - Autosomal recessive
Término que se utiliza para describir un rasgo o patología que requiere la presencia de las dos copias de un determinado alelo para que se exprese el fenotipo. Se refiere específicamente a los genes de uno de los 22 pares de autosomas.
Árbol genealógico que representa el patrón de herencia autosómica recesiva.
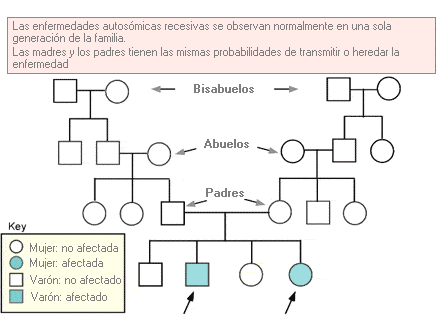
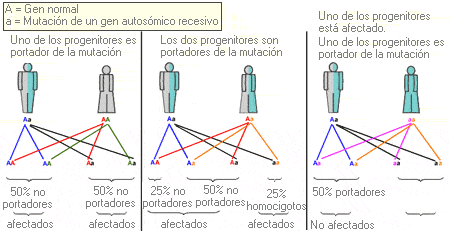
Probabilidad de que una mutación de un gen autosómico recesivo sea transmitida a los descendientes.
B
C
Codominante - Codominant
Situación genética en la que ambos alelos de un par, en estado de heterocigosis, se expresan igualmente, no existiendo dominancia de uno sobre otro.
Coeficiente de endogamia (F) - Coefficient of imbreeding
Probabilidad de que un individuo homocigoto en un locus determinado haya recibido ambos alelos del mismo ancestro (alelos idénticos por descendencia-identical by descent).
D
Deriva genética - Genetic Drift
Fluctuación (aumento o disminución) en la frecuencia de una variante genética determinada que se produce en una población concreta por azar. Típicamente ocurre en poblaciones pequeñas y aisladas y es común tras un fenómeno de “cuello de botella” en el que el tamaño de una población se reduce drásticamente, pudiéndose perder alelos raros y disminuir el pool genético. La deriva genética puede provocar que una población nueva sea genéticamente diferente de la población de origen y es uno de los mecanismos básicos en la evolución de las especies.
E
F
G
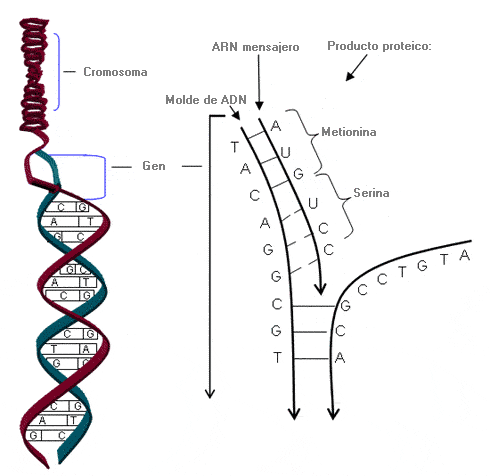
Gen - Gene
Gen dominante - Dominant gene
Gen que produce un efecto (o fenotipo) en el organismo independientemente del estado del alelo correspondiente.
Genotipaje - Genotyping
Pruebas que revelan los alelos específicos heredados por un individuo; son especialmente útiles en situaciones en las que más de una combinación de genotipos puede dar lugar a la misma presentación clínica, como en el caso del grupo sanguíneo ABO, en el que los genotipos AO y AA dan lugar al grupo sanguíneo A.
Genotipo - Genotype
Constitución genética de un organismo o célula; se refiere también al grupo específico de alelos heredados en un locus.
H
Haploinsuficiencia - Haploinsufficiency
Situación en la que un individuo está clínicamente afectado porque una sola copia del gen normal es incapaz de producir proteína en cantidad o calidad suficiente para asegurar la función normal. Puede deberse a que el individuo es heterocigótico para determinada mutación génica o es hemicigótico en un locus particular, debido normalmente a una deleción del alelo correspondiente.
Haplotipo - Haplotype
Conjunto de alelos contenidos en un locus (o en varios loci) de una misma dotación haploide. El haplotipo podemos referirlo a un solo locus o a un genoma completo pero siempre se refiere a uno de los dos alelos de cada gen.
Hardy-Weinberg (Ley) - Hardy-Weinberg law
Extensión de las leyes Mendelianas que describen las frecuencias de alelos y de genotipos para poblaciones en equilibrio génico.
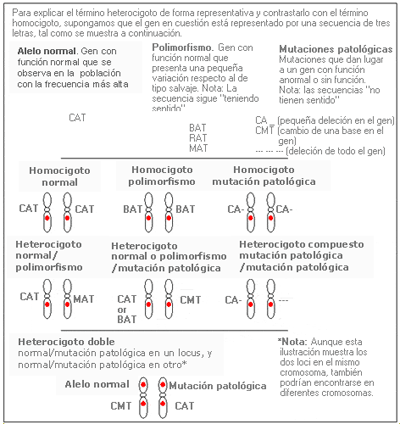
Heterocigoto - Heterozygote
Individuo que tiene dos alelos diferentes en un locus, uno en cada cromosoma. En el caso de una mutación patológica, un alelo es normal y otro anormal.
Heterocigoto compuesto: Individuo que tiene dos alelos anormales o mutados diferentes en un mismo locus, uno en cada cromosoma; se refiere normalmente a los individuos afectados por una enfermedad autosómica recesiva.
Heterocigoto doble: Individuo que presenta dos mutaciones génicas diferentes (es decir, es heterocigótico) en dos loci genéticos distintos.
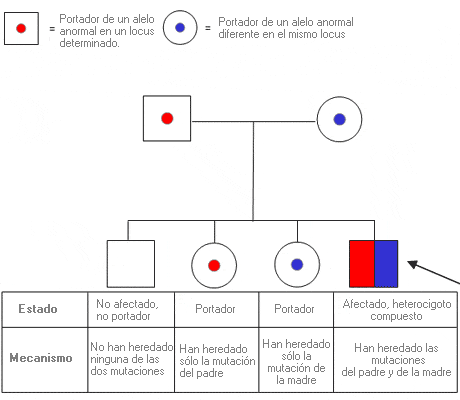
Heterocigoto compuesto - Compound heterozygote
Individuo que tiene dos alelos anormales o mutados diferentes en un mismo locus, uno en cada cromosoma; normalmente se refiere a los individuos afectados por una enfermedad autosómica recesiva.
Los hijos/as afectados de padres portadores de una mutación diferente en el mismo locus del gen son heterocigotos compuestos.
Heterogeneidad alélica - Allelic heterogeneity (sinónimo: heterogeneidad molecular)
Heterogeneidad de locus - Locus heterogeneity
Situación en que las mutaciones producidas en Genes que se encuentran en diferentes loci cromosómicos dan lugar al mismo Fenotipo.
Ejemplo de heterogeneidad de locus: Retinitis pigmentosa (RP)
Fenotipo: Las anomalías de los fotorreceptores (bastoncillos y conos) de la retina dan lugar a una adaptación inadecuada a la oscuridad, causan pérdida de visión periférica y finalmente pérdida progresiva de visión.
Loci: Las mutaciones producidas en los genes en más de 35 loci diferentes dan lugar a retinitis pigmentosa no sindrómica que es clínicamnete indistinguible, por lo que no es posible deducir el gen alterado basándose sólo en el exámen ocular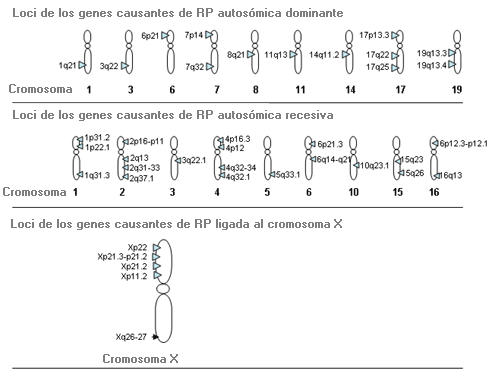
Comparar y contrastar el concepto de heterogeneidad de locus con lo siguiente:
Heterogeneidad alélica: Un solo fenotipo originado por diferentes mutaciones en el mismo gen y locus cromosómico (Nota: algunos trastornos muestran heterogeneidad de locus, otros heterogeneidad alélica y otros ambas características)
Expresividad: Variación de las manifestaciones clínicas (tipo y severidad) de una enfermedad genética entre individuos que presentan la misma alteración genética, incluso dentro de la misma familia.
(Nota: La heterogeneidad de locus implica que los individuos con diferentes genotipos tienen el mismo fenotipo; mientras que la expresividad variable implica que los individuos con el mismo genotipo presenten un fenotipo que muestra diferencias tanto en la severidad como en la edad de comienzo de sus manifestaciones).
Heterogeneidad genética - Locus heterogeneity
Situación en que las mutaciones producidas en genes distintos dan lugar al mismo fenotipo.
Fenotipo: Las anomalías de los fotorreceptores (bastoncillos y conos) de la retina dan lugar a una adaptación inadecuada a la oscuridad, causan pérdida de visión periférica y finalmente pérdida progresiva de visión.
Loci implicados: mutaciones producidas en más de 35 genes diferentes dan lugar a retinitis pigmentosa no sindrómica, que es clínicamente indistinguible. A esto se debe que no sea posible deducir qué gen es el alterado basándose sólo en el examen ocular.
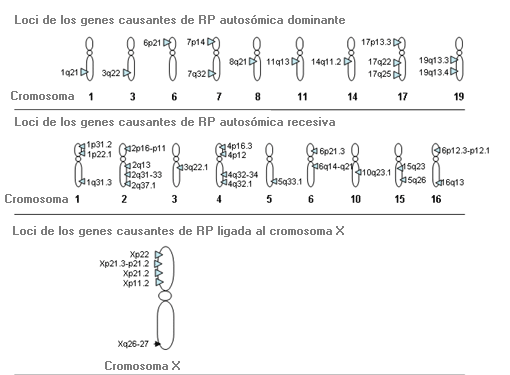
Heterogeneidad alélica: Un solo fenotipo originado por diferentes mutaciones en el mismo gen y locus cromosómico. (Nota: algunos trastornos muestran heterogeneidad genética, otros heterogeneidad alélica y otros ambas características).
Expresividad variable: variación de las manifestaciones clínicas (tipo y severidad) de una enfermedad genética entre individuos que presentan la misma alteración genética, incluso dentro de la misma familia.
(Nota: La heterogeneidad genética implica que los individuos con diferentes genotipos tienen el mismo fenotipo, mientras que la expresividad variable implica que los individuos con el mismo genotipo presenten un fenotipo que muestra diferencias tanto en la severidad como en la edad de comienzo de sus manifestaciones).
Homocigoto - Homozygote
Individuo que tiene dos alelos idénticos en un mismo locus determinado, uno en cada cromosoma.
I
Informatividad - Informativeness
En relación a un marcador genético, un descendiente de una pareja será informativo cuando la probabilidad de que se pueda deducir el origen parental de cada uno de los alelos de un locus en concreto es alta.
J
K
L
Ley de Hardy-Weinberg - Hardy-Weinberg Law
También conocido como equilibrio de Hardy-Weinberg. Principio que establece que la variación genética en una población se mantendrá constante de una generación a la siguiente en ausencia de factores perturbadores. Cuando el apareamiento es aleatorio en una gran población sin circunstancias perturbadoras, la ley predice que tanto el genotipo como la frecuencia de los alelos se mantendrán constantes porque están en equilibrio.
M
Marcador - Marker
Segmento identificable de ADN (p. ej. RFLP, VNTR, microsatélite) que al presentar una variación amplia entre los individuos, permite analizar su herencia y segregación con los alelos de un gen determinado. Se utiliza en el análisis de ligamiento.
Mutación dominante negativa - Dominant negative mutation
Mutación cuyo producto génico afecta de forma adversa al producto génico normal de tipo salvaje de la misma célula, normalmente al dimerizarse (combinarse) con él. En las moléculas poliméricas, tales como el colágeno, las mutaciones dominantes negativas suelen ser más deletéreas que las que no originan ningún producto génico (mutaciones nulas o alelos nulos).
N
Ñ
O
Oligonucleótidos específicos del alelo - Allele-specific oligonucleotide testing (ASO)
Prueba de genética molecular empleada para la detección de una mutación específica por medio de un segmento sintético de ADN de una longitud aproximada de 20 pares de bases (oligonucleótido). El investigador diseña la secuencia específica que se une a la secuencia complementaria en una muestra de ADN problema, lo que permite su identificación.
P
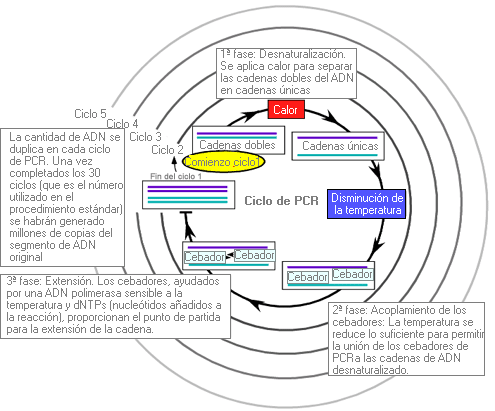
PCR - PCR (Polymerase chain reaction) (sinónimo: reacción en cadena de la polimerasa)
Procedimiento que genera millones de copias de un segmento corto de ADN mediante ciclos repetidos de: 1) desnaturalización del ADN, 2) acoplamiento de los oligonucleótidos y 3) extensión mediante la acción de la ADN polimerasa. La PCR es un procedimiento muy común en los estudios de genética molecular y se puede utilizar para: 1) generar una cantidad suficiente de ADN para realizar determinadas pruebas (p. ej. análisis de la secuencia, rastreo de mutaciones); o 2) puede ser una prueba diagnóstica en sí misma (por ejemplo amplificación específica del alelo, cuantificación del número de repeticiones de trinucleótidos).
Pérdida de heterocigosidad - Loss of heterozygosity (LOH, de sus siglas en inglés)
Cuando en un locus determinado, que es heterocigoto para un alelo mutante deletéreo y un alelo normal, se produce una deleción, u otro evento mutacional, en el alelo normal que convierte a la célula bien en hemicigótica (es decir, con un único alelo deletéreo), bien en homocigótica para el alelo deletéreo.
La LOH es el mecanismo en el que se basa el desarrollo de algunos tipos de cáncer en los que una sola mutación en uno de los homólogos de un gen supresor tumoral no es suficiente para iniciar el crecimiento del tumor. Sin embargo, la deleción o inactivación del segundo alelo en el cromosoma homólogo, da lugar a un crecimiento celular incontrolado.
La LOH puede ser el fundamento del desarrollo del cáncer en tumores esporádicos y hereditarios. En los tumores esporádicos, los dos alelos son normales en el momento de la concepción, mientras que en los tumores hereditarios hay un alelo mutante en el momento de la concepción.
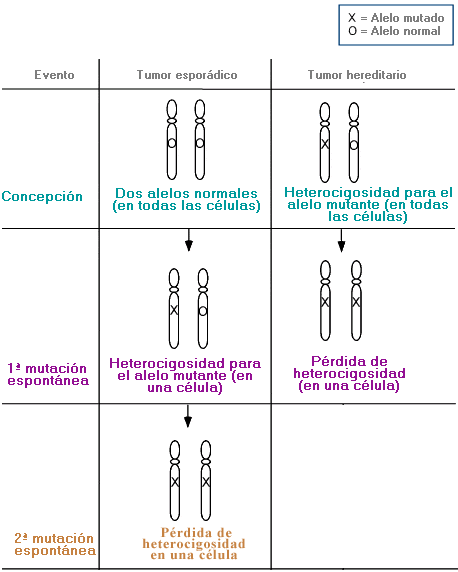
Polimorfismo - Polymorphism
Portador - Carrier
Portador sintomático - Manifesting carrier
Individuo que en un locus determinado presenta un alelo recesivo patológico en un cromosoma y un alelo normal en el otro cromosoma y que manifiesta algunos síntomas de la enfermedad. Este término se usa con frecuencia para portadoras de una mutación recesiva ligada al cromosoma X que manifiestan signos de la enfermedad, aunque su fenotipo suele ser menos severo que el de los varones con la misma mutación.
Aunque con algunas excepciones, los portadores de una mutación patológica recesiva por regla general no manifiestan síntomas clínicos de la enfermedad. Sin embargo, los portadores de mutaciones recesivas ligadas al cromosoma X presentan ocasionalmente algunas manifestaciones clínicas de la enfermedad, por lo que se les denomina portadores sintomáticos. Se supone que el mecanismo subyacente en los portadores sintomáticos es una inactivación sesgada o no aleatoria del cromosoma X, de tal manera que en la mayoría de las células el cromosoma X activo es el que tiene la mutación.
Ejemplos de enfermedades ligadas al cromosoma X en las que son frecuentes las portadoras sintomáticas:
Hemofilia A: Tiempo de coagulación prolongado, hemorragia excesiva durante intervenciones quirúrgicas menores y menstruaciones abundantes.
Distrofia muscular de Duchenne: Debilidad muscular, hipertrofia de pantorrillas leve y/o miocardiopatía dilatada.
Síndrome X frágil (mutación con penetrancia completa): Retraso mental.
Q
R
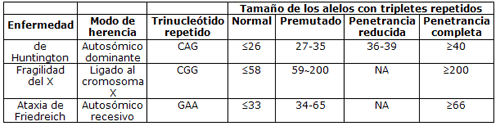
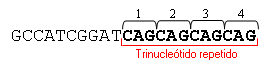
Repetición de trinucleótidos - Trinucleotide repeat
Secuencias de tres nucleótidos que se repiten varias veces en tándem en un mismo gen. La variación polimórfica normal en el número de repeticiones, que carece de significación clínica, ocurre normalmente entre los individuos. Los alelos anormalmente grandes se clasifican de menor a mayor tamaño en alelos mutables normales, alelos de penetrancia reducida y alelos de penetrancia completa, respectivamente.
La secuencia de un alelo que contiene cuatro repeticiones CAG tiene la estructura siguiente:
Un trinucleótido repetido con nueve repeticiones CCG tiene la estructura siguiente:
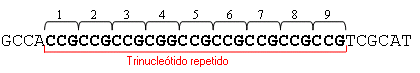
1. A diferencia de otros alelos que son estables de una generación a otra, los alelos que contienen repeticiones de trinucleótidos son inestables y dinámicos y pueden experimentar una expansión cuando son transmitidos de una generación a la siguiente. Las expansiones grandes del número de repeticiones pueden causar anomalías en la función y expresión de un gen. A continuación, se proporcionan algunos ejemplos de trastornos genéticos causados por expansiones de trinucleótidos repetidos:
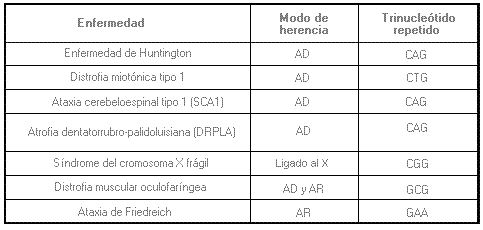
2. Las expansiones causantes de enfermedad, pueden ser pequeñas (A) o grandes (B).
A) Distrofia muscular oculofaríngea
Tamaño de los alelos normales: 6 repeticiones GCG
Tamaño de los alelos de penetrancia completa: 8-13 repeticiones GCG.
B) Distrofia miotónica1
Tamaño de los alelos normales: <30 repeticiones CTG
Tamaño de los alelos de penetrancia completa: 80-2000 repeticiones CTG
3. En algunos trastornos causados por repetición de trinucleótidos, tales como el síndrome del cromosoma X frágil, existe un rango de repeticiones de alelos mutables normales que son inestables y tienden a expandirse cuando son transmitidos de una generación a la siguiente. Los individuos con alelos mutables normales no suelen resultar afectados.
Síndrome del cromosoma X frágil
Tamaño de los alelos normales: ≤58 repeticiones CGG
Tamaño de los alelos mutables normales: 59- ~ 200 repeticiones CGG
Tamaño de los alelos de penetrancia completa: <200 repeticiones CGG
En algunos trastornos causados por repetición de trinucleótidos, tales como la enfermedad de Huntington, existe un rango de alelos de penetrancia reducida (36-39 alelos), por lo que uno de estos alelos podría dar lugar a una enfermedad de comienzo muy tardío y a ninguna manifestación clínica de la enfermedad durante la vida del individuo.
Enfermedad de Huntington
Tamaño de los alelos normales: ≤26 repeticiones CAG
Tamaño de los alelos mutables normales: 27-35 repeticiones CAG
Tamaño de los alelos de penetrancia reducida: 36-39 repeticiones CAG
Tamaño de los alelos de penetrancia completa: ≥40 repeticiones CAG
S
Segregación - Segregation
Separación de los cromosomas homólogos y su distribución aleatoria en los diferentes gametos en la meiosis. Se aplica también a la separación de los alelos de un locus y su distribución en las células hijas que se produce durante la meiosis o en la mitosis.
Silvestre - Wild type
El alelo que se encuentra con mayor frecuencia en la naturaleza, y que por tanto se designa arbitrariamente como "normal".
T
U
V
Variante alélica de significado desconocido - Allelic variant of unknown significance
Alteración de la secuencia normal de un gen, cuyo significado no está claro hasta realizar un estudio adicional del genotipo y de su correspondiente fenotipo en una población suficientemente amplia; la secuenciación completa de un gen permite identificar generalmente numerosas (a veces cientos) variantes alélicas de un gen determinado.
Ejemplos de variantes alélicas:
· Variante alélica normal: Cambio (mutación) que no interfiere en la función normal del gen y está asociado con un fenotipo normal; se conoce también como polimorfismo benigno. Nota: En GeneReviews, la composición del gen con función normal se describe generalmente bajo este epígrafe en el texto, lo que implica que los alelos salvajes y los polimorfismos benignos se incluyan en la misma categoría; sin embargo, el término “variante” implica técnicamente un cambio con respecto al tipo salvaje.
· Variante alélica patológica: Cambio (mutación) que altera la función normal del gen y da lugar a un fenotipo anormal; se conoce también como mutación patológica.
· Variante alélica de significado desconocido: Cambio (mutación) cuyo resultado es un fenotipo desconocido. Nota: Las variantes alélicas de significado desconocido deben ser estudiadas en poblaciones amplias, para determinar si se pueden clasificar definitivamente como variantes alélicas normales o patológicas.
Es probable que la secuenciación completa de cualquier gen revele numerosas variantes alélicas. Consideremos los ejemplos siguientes:
· Gen CFTR asociado a fibrosis quística: Se han documentado más de 2000 variantes alélicas con más de 900 mutaciones patológicas conocidas.
· Gen PAH asociado a fenilcetonuria: Se han documentado más de 3000 variantes alélicas con más de 400 mutaciones patológicas conocidas.
· Gen BRCA1 asociado a cáncer de mama y de ovario: Se han documentado más de 600 variantes alélicas de las cuales aproximadamente un tercio son de significado clínico desconocido.
W
Wild-type -
Wild-type significa en inglés 'salvaje' o 'silvestre' y se refiere a la forma más común, aquella que predomina en la población. Los genetistas llaman los individuos con la versión 'normal' de una característica individuos wild-type y los individuos con una característica única los denominan mutantes. Si nos referimos a variantes genéticas el alelo wild-type es el alelo que se encuentra en mayor frecuencia en la población general.