- Todos
- A
- B
- C
- D
- E
- F
- G
- H
- I
- J
- K
- L
- M
- N
- Ñ
- O
- P
- Q
- R
- S
- T
- U
- V
- W
- X
- Y
- Z
A
Ácido aspártico - Aspartic acid
El ácido aspártico o su forma ionizada, el aspartato (símbolos Asp y D) es uno de los veinte aminoácidos que pueden componer las proteínas. En el ARN se encuentra codificado por los codones GAU o GAC.
Ácido glutámico - Glutamic acid
El ácido glutámico, o en su forma ionizada, el glutamato (abreviado Glu o E) es uno de los 20 aminoácidos que forman parte de las proteínas. Es crítico para la función celular y no es nutriente esencial porque el ser humano puede sintetizarlo a partir de otros compuestos.
Acrocéntrico - Acrocentric
Tipo de cromosoma con el centrómero cercano a un extremo. Los cromosomas acrocéntricos en la especie humana son el 13, 14, 15, 21 y 22. Contienen brazos cortos satélites que albergan genes codificantes para el ARN ribosómico.
Adaptador (relativo a secuenciación masiva) - Adapter
Cebador que se une a los extremos 5´y 3´ de cada fragmento de ADN en una librería de secuenciación. Actúa en la amplificación y secuenciación del ácido nucleico adyacente.
Adenina - Adenine
ADN (Ácido desoxirribonucleico) - DNA ( Deoxyribonucleic acid)
Es la molécula portadora la información genética, que posibilita su transmisión de una generación a la siguiente.
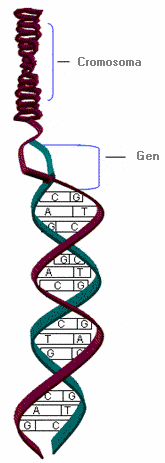
El ADN está constituido por dos cadenas de nucleótidos complementarias unidas entre sí por puentes de hidrógeno, enrollada la una sobre la otra formando una doble hélice. El elemento esencial del ADN es el nucleótido formado por un azúcar, la desoxirribosa, una base nitrogenada y un grupo fosfato. Las bases nitrogenadas son purinas: Adenina (A) y Guanina (G), y pirimidinas: Timina (T) y Citosina (C). Estas bases se unen al carbono 1 de la desoxirribosa para formar los nucleósidos, que a la vez se unirán con los grupos fosfato para formar los nucleótidos. Las bases nitrogenadas se unen de manera específica, guanina con citosina y adenina con timina, mediante puentes de hidrógeno.
El orden o secuencia de nucleótidos es la estructura primaria del ADN. La información genética reside en la secuencia de bases. Los genes son segmentos del ADN que codifican para proteínas. El orden de las bases en el gen determina la secuencia de aminoácidos que compondrán la proteína.
En las células eucariotas, el ADN se encuentra en el núcleo celular formando los cromosomas.
ADNc - cADN
ADN complementario, generado a partir del molde de ARN, mediante la retrotranscripción del ARNm maduro.
Afectado - Affected
Individuo que manifiesta síntomas de una enfermedad o patología determinada.
Alanina - Alanine
Alanina (Ala o A) es uno de los aminoácidos que forman las proteínas de los seres vivos, el más pequeño después de la glicina y se clasifica como hidrófobico. Es un aminoácido no esencial para el ser humano pero es de gran importancia y es codificada por los codones GCU, GCC, GCA y GCG.
Alelo - Allele
Un alelo es cada una de las dos o más versiones de un gen. Un individuo hereda dos alelos para cada gen, uno del padre y el otro de la madre. Los alelos se encuentran en la misma posición dentro de los cromosomas homólogos. Si los dos alelos son idénticos, el individuo es homocigoto para este gen.
Tipos de alelos denominados según su función: En la figura siguiente se muestran las proporciones aproximadas de los alelos en un locus genético hipotético, en una población determinada. Nota: Las mutaciones patológicas, los polimorfismos y las variantes alélicas de significación desconocida se consideran alelos “mutantes” en todos los casos.
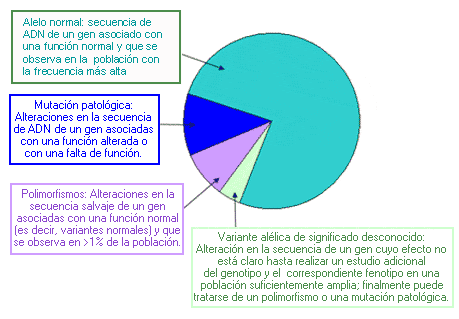
- Alelo materno: Heredado de la madre
- Alelo paterno: Heredado del padre
Los individuos pueden ser:
- Homocigóticos: cuando portan los alelos del gen iguales, cada uno procedente de un progenitor.
- Heterocigóticos: cuando los dos alelos del gen son diferentes.
Alelo de penetrancia completa - Full penetrance allele
Alelo que tiene una repercusión fenotípica en todos los individuos que son portadores de ese alelo.
Alelo de penetrancia incompleta (o reducida) - Incomplete (or reduced) penetrance allele
Alelo que no tiene una repercusión fenotípica en todos los individuos que son portadores, tan solo en una porción de ellos.
Alelo normal - Wild type alelle
El asociado al fenotipo normal, el más común en la población. Opuesto a alelo mutante. También llamado “alelo salvaje” o alelo silvestre”.
Alelo nulo - Null allele
Alelo que da lugar bien a la ausencia de producto génico, o a un producto sin actividad. Una deleción del gen es necesariamente un alelo nulo.
Alelo premutado - Mutable normal allele (sinónimos: alelo intermedio, premutación)
En trastornos causados por expansión de repeticiones de nucleótidos, alelo que no está asociado con síntomas clínicos, pero puede expandirse hasta convertirse en un alelo de gran tamaño con repercusión clínica. También denominado “premutación” o “alelo intermedio”.
En trastornos causados por expansión de repeticiones de nucleótidos, el alelo permutado no está asociado con síntomas clínicos. Su tamaño es superior al del alelo normal y puede expandirse cuando es transmitido a los descendientes, transformándose en una alelo mutado.
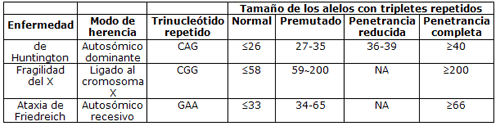
De www.acmg.net/Pages/ACMG_Activities/stds-2002/HD.htm (con acceso 17-12-03)
Alfa-fetoproteína (AFP) - Alpha-fetoprotein
Glicoproteína fetal que se excreta al líquido amniótico y alcanza niveles anormalmente altos en el mismo cuando el feto tiene ciertas anormalidades, especialmente defectos en la abertura de tubo neural.
Alineamiento de secuencias - Sequence alingment
Alteración de la secuencia - Sequence alteration: (sinónimo: mutación)
Cualquier cambio producido en el adn[o mas concretamente en un gen con respecto a su estado natural. Si no tiene repercusión clínica se denomina polimorfismo; cuando se trata de un cambio que tiene o puede tener repercusión clínica se suele denominar mutación. Cuando no se conoce si el cambio tiene o no repercusión clínica se habla de cambio o variante de significado desconocido.
Alu, Secuencias repetidas Alu - Alu, Alu element
Secuencias de ADN repetidas distribuidas a lo largo del genoma. Son las secuencias repetidas más frecuentes, representando el 5% del genoma humano. Su nombre se debe a que son reconocidas y cortadas por la enzima de restricción Alu.
Aminoácido - Amino acid
Un aminoácido es una molécula orgánica con un grupo amino (-NH2) y un grupo carboxilo (-COOH). Los aminoácidos son compuestos orgánicos que se combinan para formar proteínas. Los aminoácidos se clasifican en tres grupos: esenciales, no esenciales y condicionales.
Los 20 aminoácidos son:
Glicina (Gly, G)
Alanina (Ala, A)
Valina (Val, V)
Leucina (Leu, L)
Isoleucina (Ile, I)
Prolina (Pro, P)
Serina (Ser, S)
Treonina (Thr, T)
Cisteína (Cys, C)
Metionina (Met, M)
Asparragina (Asn, N)
Glutamina (Glu, Q)
Fenilalanina (Phe, F)
Tirosina (Tyr, Y)
Triptófano (Trp, W)
Lisina (Lys, K)
Arginina (Arg, R)
Histidina (His, H)
Aspartato (Asp, D)
Glutamato (Glu, E)
Aminoácidos, Secuencia de, - Amino acid sequence
Amniocentesis - Amniocentesis
Técnica de diagnóstico prenatal que utiliza las células del líquido amniótico para determinar el cariotipo del feto y realizar estudios bioquímicos.
Amniocito - Amniocyte
Células fetales obtenidas por amniocentesis.
Amplicón - Amplicon
Fragmento de ADN amplificado por la reacción en cadena de la polimerasa (PCR) o cualquier otro proceso que dé lugar a la producción de diferentes copias de ese fragmento.
Amplificación - Amplification
Cualquier proceso que causa la replicación de una secuencia concreta de ADN en cantidad desproporcionadamente elevada.
Amplificación puente (relativo a secuenciación masiva) - Bridge amplification
Generación de copias in situ de una molécula específica de ADN en un soporte sólido cubierto de cebadores.
Anafase - Anaphase
Es la tercera fase de la mitosis y meiosis en la que los cromosomas duplicados son separados. Las cromátidas son entonces desplazadas a polos opuestos de la célula en división por el huso mitótico o meiótico, para que cada célula hija herede una copia de cada cromosoma.
Análisis basados en ADN - DNA-based analysis
Pruebas que implican el estudio de ADN mediante un análisis de ligamiento, secuenciación o cualquiera de los diferentes métodos utilizados para la detección de mutaciones.
Análisis de dosis - Dosage analysis
Método para determinar la cantidad de diversos componentes, incluyendo ADN, ARN y proteínas, por comparación con un estándar conocido; se puede utilizar para determinar el número de copias de una secuencia de ADN (es decir, para investigar posibles mutaciones por duplicación y deleción), mediante la comparación visual de la intensidad de las bandas o la cuantificación numérica con densitometría. Si un gen presenta copias adicionales, la intensidad es de más del 100% en el gel o película; mientras que si se ha perdido una copia del gen, la intensidad es de aproximadamente un 50%.
Análisis de fragmentos de restricción de longitud polimórfica - Restriction fragment length polymorphism analysis (sinónimo: análisis RFLP, estudios RFLP)
Estudio de los fragmentos de ADN de tamaño previsible originados por la digestión (corte) de una cadena de ADN por una enzima de restricción específica. Las alteraciones (mutaciones o polimorfismos) producidas en la secuencia de ADN que destruyen o crean nuevos sitios de corte en el ADN, modifican el tamaño (y el número) de los fragmentos de ADN resultantes de la digestión por una enzima de restricción específica.

Análisis de ligamiento - Linkage analysis (sinónimo: análisis indirecto del ADN)
Estudio de los polimorfismos de las secuencias de ADN (variantes normales) que están próximos a/o dentro de un gen de interés (ligamiento) para identificar, en una misma familia, la segregación de una mutación patológica en un gen determinado.
EXPLICACIÓN:
Las tres fases del análisis de ligamiento son las siguientes:
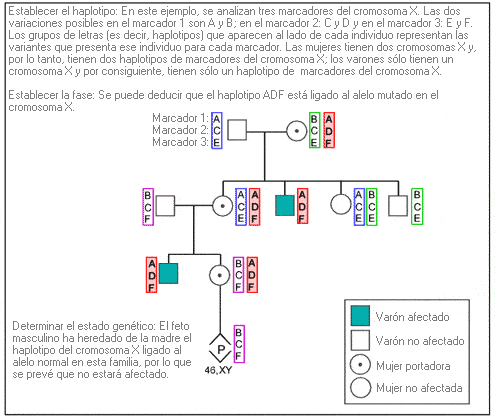
1. Establecer los haplotipos de cada individuo: Se analizan los múltiples marcadores de ADN dispuestos a cada lado (marcadores flanqueantes) o dentro (marcadores intragénicos) de la región del gen de interés, con objeto de determinar el conjunto de marcadores (haplotipos) de cada miembro de la familia.
2. Establecer la fase, comparando los haplotipos entre los miembros de la familia: Mediante la comparación de los haplotipos de los miembros de la familia con un estado genético conocido (es decir, afectados, no afectados), se puede identificar el haplotipo asociado con el alelo patológico.
3. Determinar el estado genético: Una vez establecido el haplotipo asociado con la enfermedad, es posible determinar el estado genético de los miembros de una familia de riesgo.
Ejemplo: Análisis de ligamiento para el diagnóstico prenatal de una enfermedad ligada al cromosoma X.
Algunas implicaciones clínicas
El análisis de ligamiento se utiliza generalmente cuando el análisis de ADN directo no es viable, porque se desconoce el gen de interés o no se puede detectar una mutación en ese gen en una familia específica.
En la mayoría de los casos, el haplotipo en sí carece de significación; sólo tiene sentido en el contexto de un estudio familiar. Sólo en el caso raro de que el desequilibrio del ligamiento sea un haplotipo específico, habrá una probabilidad muy elevada de que esté asociado con una mutación patológica específica.
La exactitud del análisis de ligamiento depende de:
La exactitud del diagnóstico clínico en un miembro o miembros de la familia que estén afectados.
La distancia entre la mutación patológica y los marcadores. El análisis de ligamiento puede proporcionar resultados falso positivo o falso negativo si, durante la formación de los gametos (meiosis), se produce recombinación que implique a los marcadores y suponga un intercambio entre el material genético de los cromosomas heredados de la madre o del padre. El riesgo de recombinación es proporcional a la distancia entre la mutación patológica y los marcadores. El riesgo de recombinación es más bajo si se utilizan marcadores intragénicos.
La informatividad de los marcadores genéticos en la familia del paciente. Si la secuencia de ADN de una variante determinada difiere entre los cromosomas heredados de la madre y del padre, este marcador es informativo. Por el contrario, si la secuencia de ADN de una variante determinada no difiere entre los dos cromosomas, el marcador no es informativo.
Análisis de matrices de ADN - DNA microarray analysis
Análisis de metilación - Methylation analysis
Métodos utilizados para evaluar el estado de metilación de un gen (unión de los grupos metilo a las citosinas del ADN). Los genes metilados no se expresan. La metilación desempeña un papel importante en la inactivación del cromosoma X y en la impronta genética.
1. Análisis con enzimas de restricción sensibles a metilación: El ADN es digerido (cortado) por una enzima de restricción que sólo puede digerir ADN no metilado. Se realizan análisis adicionales (por ejemplo, Southern blot y/o PCR) para determinar si el ADN se ha cortado. La presencia de ADN no cortado indica que el ADN está metilado, mientras que la presencia de ADN cortado indica que éste no está metilado.
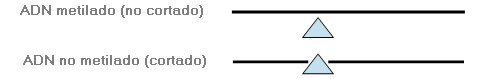
2. Análisis de secuencia tratada con bisulfito: El ADN es tratado para sustituir todos los nucleótidos citosina no metilados por nucleótidos timina. Se secuencia el ADN tratado. El ADN metilado conserva su secuencia original (normal), mientras que el ADN no metilado presenta una secuencia anormal en la que todos los nucleótidos citosina (C) se han sustituido por nucleótidos timina (T).

Análisis de microsatélites - Microsatellite analysis
Uso de secuencias repetitivas muy variables, que se encuentran en las regiones microsatélites adyacentes a los genes o intragénicas o en otras áreas de interés, como marcadores para el análisis de ligamiento, huellas moleculares de adn u otras aplicaciones diagnósticas.
Análisis de proteínas - Protein analysis
Uno de los diferentes métodos analíticos que revela la estructura o la función de un producto proteico determinado.
Análisis de replicación - Replication analysis (sinónimos: estudio de inactivación del cromosoma X)
Técnica citogenética basada en procedimientos de bandeo específicos (bandeo por replicación) para identificar el cromosoma X de replicación tardía (inactivo) en las células. En la actualidad, los estudios moleculares se utilizan con más frecuencia que el análisis de replicación para el estudio de inactivación del cromosoma X, puesto que son menos complicados, costosos y subjetivos.
Análisis de secuencias - Sequence analysis (sinónimo: secuenciación génica, secuenciación)
Proceso mediante el cual se determina la secuencia de nucleótidos en un segmento de ADN.
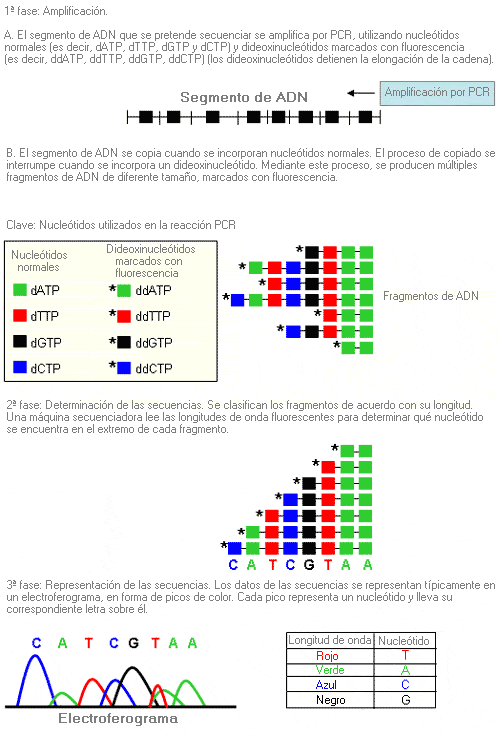
Se puede secuenciar un gen completo, pero la mayoría de las veces se secuencian sólo regiones específicas del gen que muy probablemente contendrán mutaciones (p. ej. los exones y los límites entre los intrones y los exones).
Al secuenciar un segmento de ADN, se identifican la mayoría de las variaciones producidas con respecto a la secuencia normal (por el contrario, el análisis mutacional identifica sólo mutaciones específicas en un segmento determinado de ADN).
Tipos de alteraciones de las secuencias que se pueden detectar:
Alteraciones de secuencia descritas en la literatura como patógenas
Alteraciones de secuencias previsiblemente patógenas, pero no descritas en la literatura
Alteraciones de secuencias de significación clínica imprevisible
Alteraciones de secuencias previsiblemente benignas, pero no descritas en la literatura
Alteraciones de secuencias benignas descritas en la literatura
Posibilidades en caso de que no se detecten alteraciones de las secuencias:
El paciente no presenta una mutación en el gen analizado (p. ej. hay una alteración de la secuencia en otro gen, en un locus diferente)
El paciente presenta una alteración de la secuencia que no se puede detectar mediante el análisis de secuencias (p. ej. deleción de tamaño grande)
El paciente presenta una alteración de la secuencia en una región del gen (p. ej. una región intrónica o reguladora) que no se ha detectado en el análisis de laboratorio
1Adaptado de ACGM Recommendations for Standards for Interpretation of Sequence Variations (2000)
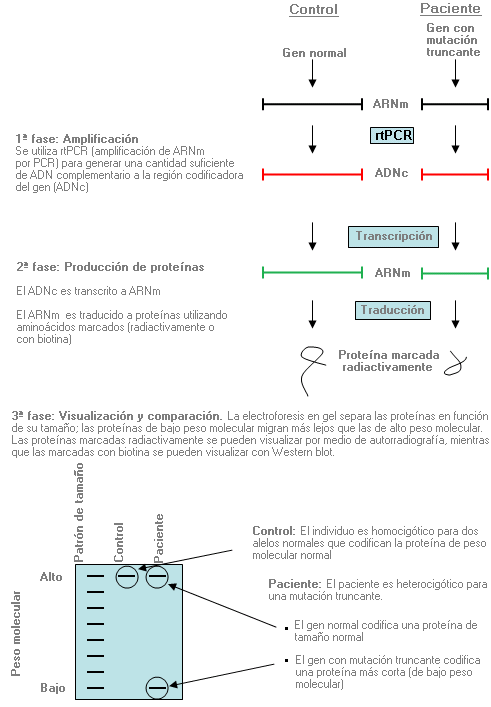
Análisis de truncación de proteínas - Protein truncation testing (sinónimo: PTT)
Método utilizado para identificar las proteínas que se acortan (truncan) a consecuencia de mutaciones que causan específicamente la terminación prematura del proceso de traducción del ARNm.
Análisis del haplotipo - Haplotype analysis
Estudio de genética molecular para identificar un conjunto de segmentos de adn que están estrechamente relacionados en su modo de herencia (ligados). Se utiliza en el análisis de ligamiento o cuando un rasgo o característica determinados presentan desequilibrio de ligamiento con un marcador genético o un grupo de [GLOSARIO]marcador[FIN_GLOSARIO]es.
Análisis del origen parental - Parental origen analysis
Análisis directo del ADN - Direct DNA analysis
La utilización de técnicas de secuenciación, análisis mutacional, rastreo de mutaciones o cualquier otro medio de estudio molecular genético, para detectar variantes en un gen que pueden estar asociadas a trastornos genéticos específicos. El análisis directo del adn es posible sólo cuando se conoce el gen o genes o la región genómica, asociados con la enfermedad.
Las técnicas utilizadas para el análisis de ADN directo pueden ser las siguientes:
Análisis de secuencias
Rastreo de mutaciones (p. ej. CSGE, DHPLC, SSCP)
Análisis mutacional (p. ej. PCR, Southern blot, análisis de repeticiones de trinucleótidos, FISH, análisis de truncación de proteínas (PTT), estudios de heterocigosidad, análisis RFLP, ASO)
El análisis de ADN directo contrasta con el análisis de ligamiento (que anteriormente se denominaba análisis de ADN indirecto)
Algunas implicaciones clínicas
El análisis de ADN directo sólo es posible cuando se conoce el gen (genes) o la región genómica asociados con una enfermedad
El análisis de ligamiento se puede realizar cuando no se conoce el gen o los genes asociados con una enfermedad
Análisis mutacional específico - Targeted mutation analysis (sinónimo: análisis mutacional dirigido)
Pruebas para detectar la presencia de una mutación específica (p. ej. Glu6Val en la Anemia de Células Falciformes), un tipo de mutación específica (p. ej. la expansión de trinucleótidos repetidos asociada a la Ataxia Espinocerebelosa tipo 1, deleciones asociadas a la Distrofia Muscular de Duchenne) o un conjunto de mutaciones (p. ej. un grupo de mutaciones en la Fibrosis Quística). Difiere de la secuenciación génica completa o el rastreo de mutaciones, en que éstos detectan la mayoría de las mutaciones en la región investigada.
El análisis mutacional se utiliza en las dos situaciones siguientes:
I. Estudio de enfermedades en las que:
La mayoría o todos los individuos afectados presentan la misma mutación (p. ej. Anemia de Células Falciformes).
La mayoría o todos los individuos afectados presentan el mismo tipo de mutación (p. ej. expansión de repeticiones de trinucleótidos).
Un grupo de mutaciones son las más comunes y explican un elevado porcentaje de los casos (véase Tabla).

II. Estudio de los miembros a riesgo de una familia tras haber identificado la mutación patológica específica de la familia en un pariente afectado. Por ejemplo, si en el análisis de la secuencia se identifica una mutación patológica en un miembro afectado de la familia, todos los familiares a riesgo deben ser sometidos sólo a un análisis mutacional dirigido a detectar esa mutación específica de la familia.
Algunas implicaciones clínicas
El análisis mutacional no es generalmente el primer estudio de genética molecular que se utiliza para la identificación de mutaciones en enfermedades que se sabe están asociadas con una proporción alta de mutaciones raras (por ejemplo, específicas de una familia).
Si en un individuo afectado no se puede detectar una mutación en el análisis mutacional, puede que sea necesario realizar un análisis de la secuencia o un rastreo de otras mutaciones para identificarla.
Análisis mutacional individualizado - Custom mutation analysis
Estudios de Genética molecular desarrollados especialmente en un laboratorio clínico en función de las necesidades, para detectar una mutación específica que predispone a una enfermedad que no se puede identificar por otros medios clínicos. Las situaciones en las que puede utilizarse un análisis mutacional individualizado son las siguientes: a) estudios de miembros de una familia que tienen riesgo de desarrollar una enfermedad específica, al haberse identificado una mutación patológica en un miembro afectado de esa misma familia; b) estudios prenatales en familias en las que se ha identificado una mutación patológica en un miembro afectado.
Aneuploidía - Aneuploidy
Un aneuploide es un individuo cuyo número de cromosomas difiere del tipo silvestre o euploide en parte de su dotación cromosómica, debido a un cromosoma extra o ausente, que siempre se asocia con una deficiencia en el desarrollo físico, mental o ambos. Generalmente, la dotación cromosómica aneuploide solo difiere de la salvaje en uno o pocos cromosomas. La aneuploidía se puede observar frecuentemente en células cancerosas.
Anotación (relativo a secuenciación) - Annotation
Información adjunta (texto, comentarios, otros) que se añade a una secuencia genómica específica.
Antecedentes familiares - Family history
Anticipación - Anticipation
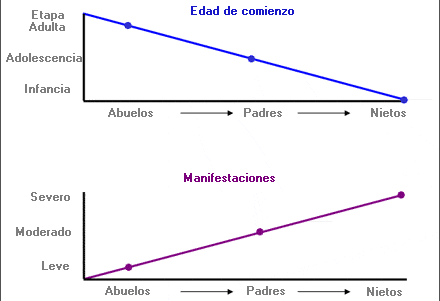
En determinadas enfermedades genéticas, tendencia a que los síntomas de una patología se presenten a edades más tempranas y/o sean más graves que en las generaciones anteriores. Se observa frecuentemente en enfermedades causadas por la expansión de trinucleótidos repetidos que tiende a aumentar de tamaño y a tener un efecto más significativo cuando se transmite de una generación a la siguiente.
Anticodon - Anticodon
Unidad de tres bases de ARN de transferencia, complementaria a un codón en el ARN mensajero.
Anticuerpo - Antibody
Proteína sintetizada por las células plasmáticas en respuesta a un antígeno. Cada anticuerpo se une específicamente a un solo antígeno con el objetivo de destruirlo, bien directamente o bien promoviendo la acción de los glóbulos blancos sobre el mismo.
Antígeno - Antigen
Sustancia ajena al cuerpo que el sistema inmunitario reconoce como “extraña” y que hace que se induzca una respuesta inmunitaria que conlleva la formación de anticuerpos. Los antígenos incluyen toxinas, sustancias químicas, bacterias, virus, etc.
Apoptosis -
Muerte celular programada, la ruta normal por la que el organismo se libera de las células dañadas, no deseadas o innecesarias.
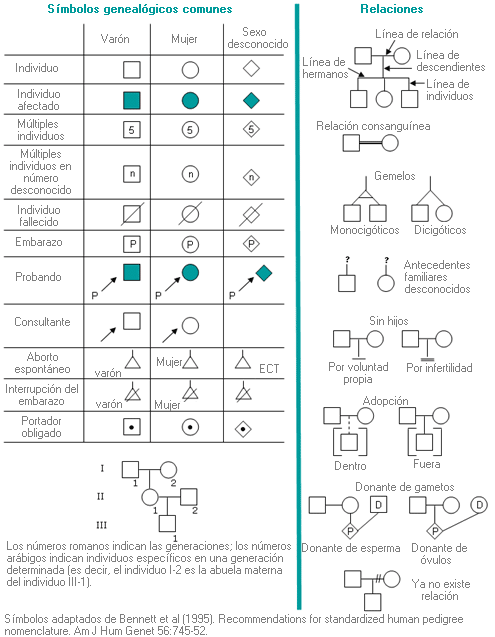
Árbol genealógico - Genealogía - Árbol familiar - Pedigree - Genograma - Pedigree
Arginina - Arginine
La arginina (arg o R) es uno de los 20 aminoácidos que se encuentran formando parte de las proteínas. Es un aminoácido condicionalmente esencial (se necesita en la dieta solo bajo ciertas condiciones), y puede estimular la función inmunológica al aumentar el número de leucocitos. La arginina está involucrada en la síntesis de creatina, poliaminas y en el ADN. En el ARNm está codificado por los codones CGU, CGC, CGA, CGG, AGA, y AGG.
ARN (ácido ribonucleico) - RNA (ribonucleic acid)
El ácido ribonucleico (ARN o RNA) es un ácido nucleico formado por una cadena de ribonucleótidos. Está formado por una cadena simple de ribonucleótidos, cada uno de ellos formado por ribosa, un fosfato y una de las cuatro bases nitrogenadas (adenina, guanina, citosina y uracilo). El ARN celular es lineal y monocatenario (de una sola cadena), pero en el genoma de algunos virus es de doble hebra.
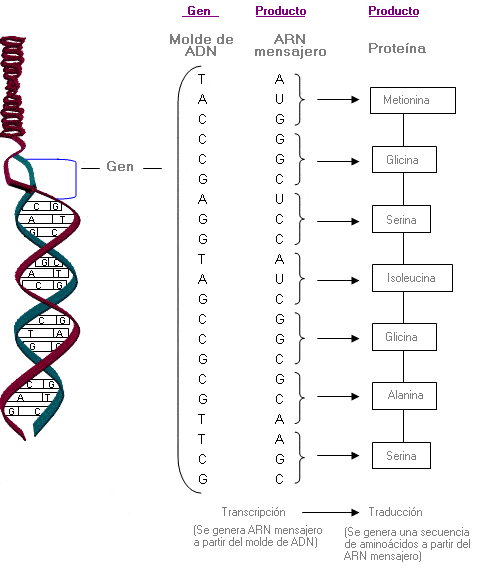
ARN mensajero (ARNm) - Messenger RNA (mRNA)
Es el ácido ribonucleico que transfiere el código genético procedente del núcleo celular a un ribosoma en el citoplasma. Se forma durante el proceso de transcripción del ADN al ARN, durante el cual la ARN polimerasa debe unirse al ADN con ayuda de cofactores de transcripción. El proceso de transcripción se lleva a cabo en el núcleo en eucariotas y en el citoplasma en procariotas.
ARNt - tRNA
ARN de transferencia (ARNt) es una pequeña molécula de ARN que participa en la síntesis de proteínas. Su función es suministrar los aminoácidos al ribosoma para que éste lleve a cabo la síntesis de la proteína según la secuencia especificada de un ARN mensajero. Una vez el ribosoma usa el aminoácido transportado por el ARNt, este se separa del ribosoma y se desplaza por el citoplasma buscando nuevos aminoácidos.
Asesoramiento genético - Genetic counseling
ASO -
Asociación - Association
En epidemiología genética, situación en la que un alelo se encuentra más o menos representado en un grupo de individuos afectos de lo que se esperaría por su frecuencia en la población general.
Asparagina - Asparagine
La asparagina (abreviada como Asn o N) es uno de los 20 aminoácidos codificados en el código genético, y los codones que la codifican son AAU y AAC. En el ser humano no es un aminoácido esencial.
Atlas del Genoma del Cáncer - The Cancer Genome Atlas TCGA
Proyecto internacional iniciado en el año 2005 con el objetivo de catalogar los cambios moleculares, a nivel de ADN, ARN, proteico y epigenético, existentes en los diferentes tipos de tumores mediante plataformas de análisis masivo y potentes análisis bioinformáticos. Con este proyecto se pretende mejorar el diagnóstico y el tratamiento de los pacientes oncológicos además de proporcionar información para prevenir y entender cuáles son los mecanismos moleculares asociados al desarrollo de esta enfermedad (https://cancergenome.nih.gov/)
Autosoma - Autosome
Autosómico - Autosomal
Se refiere a cualquiera de los cromosomas que no son los determinantes del sexo (es decir, X e Y) o a los genes de esos cromosomas.
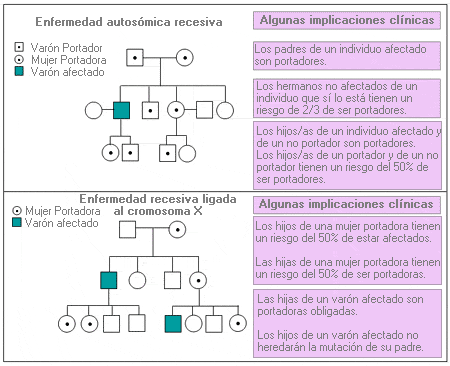
Autosómico dominante - Autosomal dominant
Término que se utiliza para describir un rasgo o patología asociados a un determinado alelo, que están presentes en todos los individuos que han heredado una sola copia de dicho alelo (heterocigotos). Se refiere específicamente a un gen de uno de los 22 pares de autosomas.
Arbol genealógico que representa el patrón de herencia autosómica dominante.
Probabilidad de que una mutación de un gen autosómico dominante sea transmitida a los descendientes.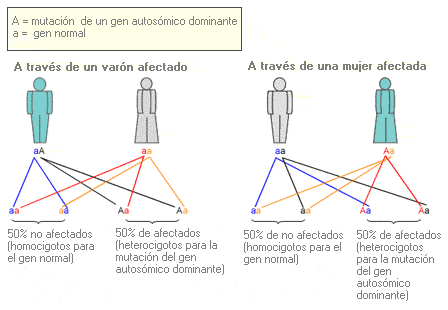
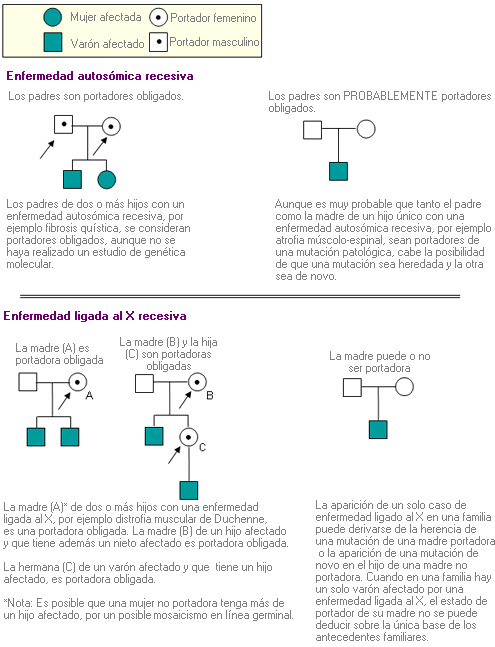
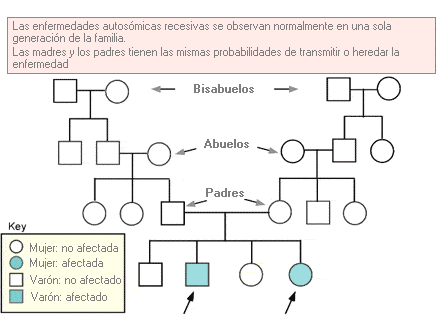
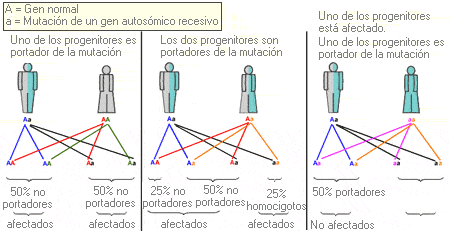
Autosómico recesivo - Autosomal recessive
Término que se utiliza para describir un rasgo o patología que requiere la presencia de las dos copias de un determinado alelo para que se exprese el fenotipo. Se refiere específicamente a los genes de uno de los 22 pares de autosomas.
Árbol genealógico que representa el patrón de herencia autosómica recesiva.
Probabilidad de que una mutación de un gen autosómico recesivo sea transmitida a los descendientes.
B
BAC, Cromosoma Artificial Bacteriano - Bacterial Artificial Chromosome
Segmento grande de ADN, generalmente constituido por 100.000-200.000 bases, de cualquier especie que es clonado e introducido en una bacteria. Una vez dentro de la bacteria, pueden obtenerse múltiples copias del mismo.
Bacteriofago - Bacteriophage
Virus que infecta y sólo se reproduce en el interior de bacterias. Es habitual referirse a él como “fago”.
BAM (Binary Aligned Mapped) -
Formato binario para el almacenamiento de datos de secuenciación. La extensión de archivo (.bam) contiene información sobre lecturas de secuencias después de haber sido estas alineadas contra un genoma de referencia. Muy utilizado para almacenar datos de secuenciación masiva.
Banco de ADN - DNA banking
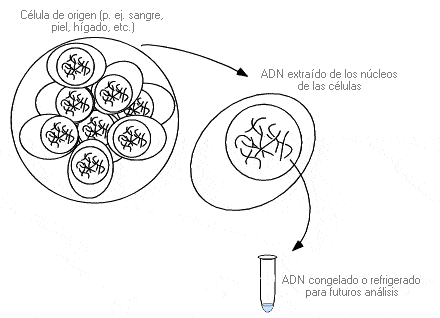
Proceso de almacenaje durante un periodo indefinido del adn extraído de cualquiera de las posibles fuentes celulares, conservado bajo congelación o refrigeración para futuros análisis.
Barr, Cuerpo de - Barr body
Base nitrogenada - Nitrogenated base
Bioética - Bioethics
Estudio de las cuestiones éticas surgidas a partir de los avances en la investigación biológica y sus aplicaciones, especialmente en medicina, en campos tales como el trasplante de órganos, la ingeniería genética, la inseminación artificial, etc.
Bioinformática - Bioinformatics
Análisis computacional y almacenamiento de datos biológicos y experimentales aplicados a estudios ómicos.
Biomarcador - Biomarker
Característica biológica, normalmente una molécula, que puede ser medida y evaluada objetivamente como indicador de un proceso biológico normal o patológico o de respuesta a una intervención terapéutica. Algunos ejemplos de biomarcadores son los perfiles de expresión génica, niveles de proteínas en fluidos corporales o cambios en la actividad eléctrica en el cerebro.
Bloque haplotípico - Haplotype Block
Región del genoma donde los diferentes polimorfismos genéticos como los SNPs son heredados en bloque.
C
cADN, Librería de - cDNA library
Colección de secuencias de adn generadas a partir de la secuencia del ARN mensajero (mARN). Estas librerías contienen sólo adn codificante de proteínas (genes), quedando excluídas las secuencias de adn que no codifican.
Cambio conservador - Conservative change
Cambio en la secuencia de aminoácidos que no afecta significativamente a la función de la proteína.
Cap (relativo a RNA mensajero) -
Nucleótido modificado que se añade al extremo 5´ de una cadena de ARN mensajero creciente y que es necesario para su normal procesamiento, estabilidad y traducción del ARN mensajero.
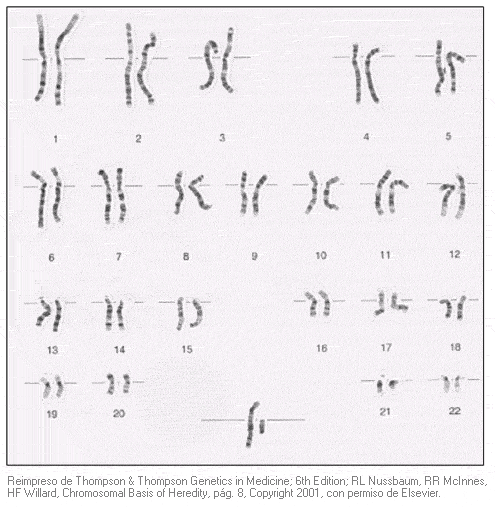
Cariotipo - Karyotype
Fotografía de los cromosomas de una célula, cortados y dispuestos en pares en función de su tamaño y el patrón de bandas y de acuerdo con una clasificación estándar.
Ejemplo de un cariotipo masculino normal, denominado 46, XY
46 se refiere al número total de cromosomas
XY indica un cariotipo masculino; XX indicaría un cariotipo femenino
Un cariotipo humano normal consta de 22 pares de autosomas y dos cromosomas sexuales. Obsérvese que cada par tiene un tamaño y un patrón rayado (en bandas) similares. Los pares de cromosomas autosómicos están numerados y organizados de mayor a menor tamaño. La flexión y doblamiento son característicos de los cromosomas y no representan ninguna anomalía.
Cariotipo espectral, SKY - Spectral Karyotype, SKY
Representación gráfica del conjunto de los cromosomas de un organismo teñidos con colores diferente. Es una técnica muy útil para identificar alteraciones cromosómicas complejas.
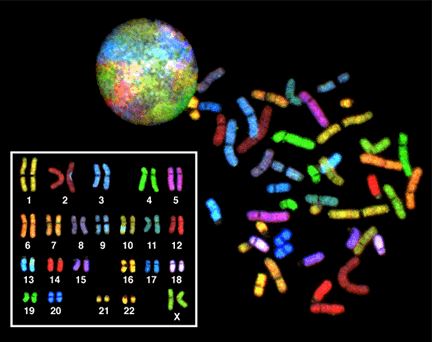
Fuente: National Human Genome Research Institute.
Caso esporádico - Sporadic case
Aparición de un trastorno de carácter, malformación o enfermedad en un solo miembro de una familia.
Caso-control (estudio) - Case-control study
En genética, estudio retrospectivo en el que se estima el riesgo relativo a desarrollar una enfermedad o condición en presencia de una variante genética específica. En este tipo de estudios, se comparan individuos que presentan una enfermedad o condición (casos) con individuos que no la presentan (controles). Si la prevalencia de la variante genética es diferente entre los dos grupos, es posible inferir que esta se asocia con un aumento o disminución del riesgo a que ocurra dicha condición.
Cebador - Primer
Una secuencia corta de oligonucleótidos que se utiliza en la reacción en cadena de la polimerasa (PCR) para la creación de moléculas más largas de ADN.
Célula - Cell
La unidad básica de un organismo vivo. Todas las eucariotas (con núcleo diferenciado), excepto las germinales, contienen una copia completa del genoma del organismo al que pertenecen.
Célula diploide - Diploid cell
Las células diploides (2n) son las células que tienen un número doble de cromosomas (a diferencia de los gametos), es decir, poseen dos series de cromosomas.
Célula haploide - Haploid cell
Una célula haploide es aquella que contiene un solo juego de cromosomas o la mitad (n, haploide) del número normal de cromosomas, en células diploides (2n, diploide). Las células reproductoras, como los óvulos y los espermatozoides de los mamíferos, la etapa asexual de hongos, briófitos y algunas algas contienen un solo juego de cromosomas.
Centimorgan (abreviado cM) -
Es la unidad de los mapas genéticos de ligamiento realizados por recombinación en eucariotas diploides. Equivale a un 1% de frecuencia de recombinación, o lo que es lo mismo, a 1 unidad de mapa.
Centrómero - Centromere
Región de constricción en el cromosoma, donde se unen las fibras del huso acromático durante la división celular. En esta región se unen las dos cromátidas del cromosoma. La presencia del centrómero determina la división del cromosoma en dos brazos (p y q), y su localización a lo largo del cromosoma sirve para clasificar los cromosomas en acrocéntricos, metacéntricos, submetacéntricos, etc.
Centrosomas - Centrosomes
Pareja de centros que organizan el crecimiento de los microtúbulos en el huso mitótico. Son visibles en los polos de las células en división en profase tardía.
ChIP (chromatin immunoprecipitation) -
Ciclo celular - Cell cycle
Estadío entre dos mitosis sucesivas. Se compone de las fases G1, S, G2 y M.
Cigoto - Zygote
Célula diploide formada por la unión de dos gametos haploides.
Cisteina - Cysteine
La cisteína (abreviada como Cys o C) es un aminoácido no esencial, lo que significa que puede ser sintetizado por los humanos. Los codones que codifican la cisteína son UGU y UGC.
Citogenética - Cytogenetics
Estudio de la estructura, función y anomalías de los cromosomas.
Citosina - Cytosine
ClinVar -
Repositorio público de variantes genéticas relacionadas con fenotipos concretos que tiene como objetivo la interpretación de su significado clínico. Las clasificaciones incluidas en ClinVar presentan varios niveles de confianza representados por estrellas.

Clon - Clone
Copia idéntica de una secuencia de ADN, o de todo un gen, o de un ser vivo. Una o más células derivadas de un solo antecesor e idénticas a éste.
Clonación - Cloning
Aislar un gen o una secuencia específica de ADN.
Clonación posicional - Positional cloning (sinónimo: Genética inversa)
Clonación o identificación de un gen causante de una enfermedad específica, basándose en su localización en el genoma, determinada por diversos métodos, incluyendo análisis de ligamiento, mapeo genómico (físico) y bioinformática, cuando no se conoce la información sobre la base bioquímica de la enfermedad. Se distingue de la estrategia más común de clonación génica en que en ésta se parte de un producto protéico conocido, se determina su secuencia de aminoácidos y se utiliza esa información para aislar el gen.
Cobertura - Coverage
Número de veces que una porción del genoma es secuenciada en una reacción de secuenciación. Frecuentemente expresado como profundidad de lectura o “depth coverage” y numéricamente como 1X, 2X, 50X, 100X, etc.
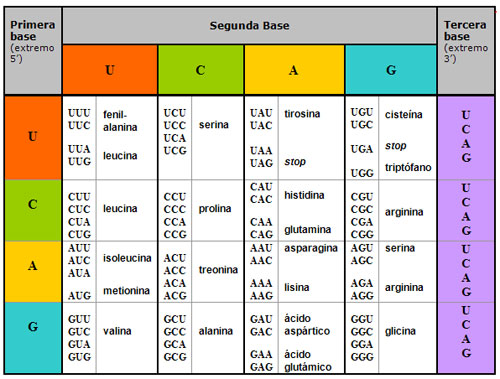
Código genético - Genetic code
Las reglas por las que la secuencia de nucleótidos de un determinado gen se traduce finalmente en la secuencia de aminoácidos de una proteína.
Codominante - Codominant
Situación genética en la que ambos alelos de un par, en estado de heterocigosis, se expresan igualmente, no existiendo dominancia de uno sobre otro.
Codón - Codon
En el adn o ARN, secuencia de tres nucleótidos que codifica determinado aminoácido o indica el comienzo o la terminación del proceso de traducción (codón de inicio, parada o terminación).
Coeficiente de endogamia (F) - Coefficient of imbreeding
Probabilidad de que un individuo homocigoto en un locus determinado haya recibido ambos alelos del mismo ancestro (alelos idénticos por descendencia-identical by descent).
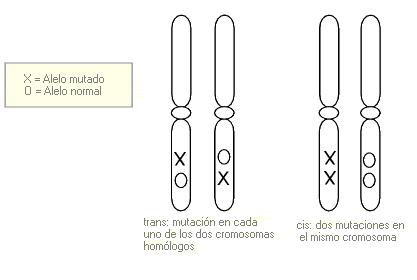
Configuración en cis - Cis configuration (sinónimos: cis, acoplamiento)
Término que indica que un individuo que es heterocigoto en dos loci adyacentes presenta esas dos mutaciones en el mismo cromosoma, en vez de una en cada cromosoma homólogo (configuración en trans).
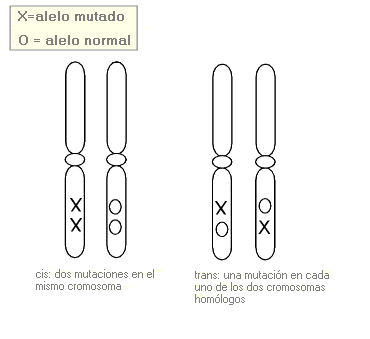
Configuración en trans - Trans configuration
Congénito - Congenital
Alteración presente desde el nacimiento, pero no necesariamente de origen genético; puede tener también origen ambiental, infeccioso, etc.
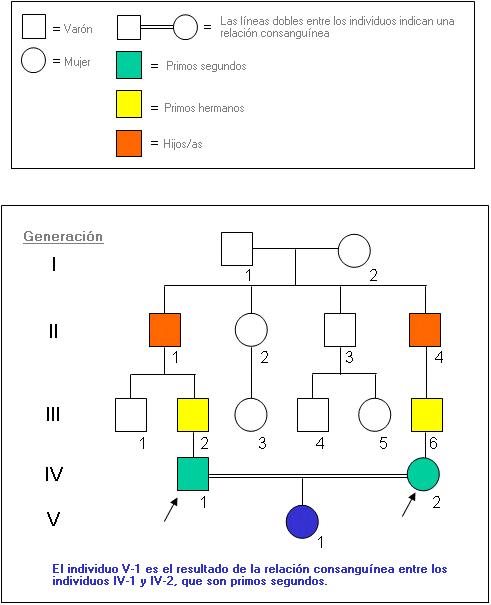
Consanguinidad - Consanguinity
Consejo genético - Genetic counselling
El proceso por el cual un individuo o los miembros de una familia, a riesgo para una enfermedad que puede ser hereditaria, son informados de las consecuencias de la enfermedad, de la probabilidad de padecerla y transmitirla, y de las formas de prevenirla o reducir sus efectos.
Consentimiento informado - Informed consent
Permiso otorgado por un individuo para ser sometido a una prueba o procedimiento específicos una vez entendidos los riesgos, beneficios, limitaciones y posibles repercusiones del procedimiento y sus resultados.
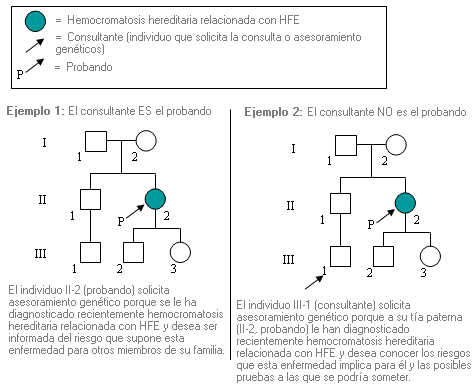
Consultante - Consultand
Individuo (no necesariamente afectado) que solicita asesoramiento genético.
Contaminación materna - Maternal contamination
Situación que se da en los estudios de diagnóstico prenatal cuando muestras de vellosidad corial, líquido amniótico o sangre de cordón, resultan contaminadas por células maternas (normalmente sanguíneas), lo que puede confundir la interpretación de los resultados del análisis genético.
Contig -
Tramo de secuencia continua in silico generada por alineamiento de lecturas de secuencias solapantes.
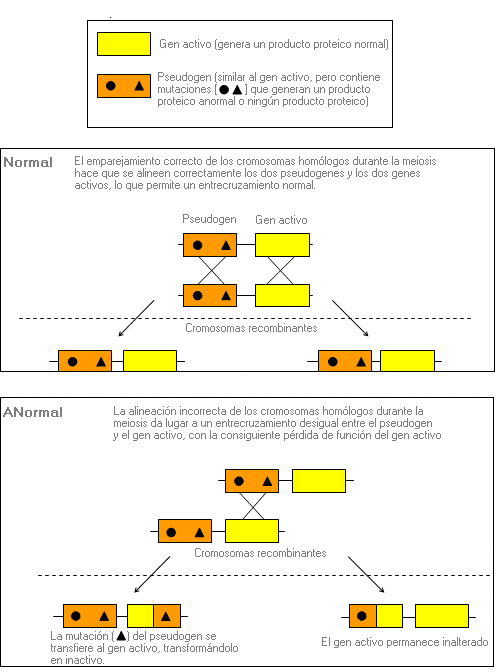
Conversión génica - Gene conversion
Transferencia de secuencias de ADN entre dos genes muy similares, que se produce la mayoría de las veces por un entrecruzamiento desigual durante la meiosis. Puede ser un mecanismo que da lugar a una mutación si la transferencia de material altera la secuencia codificadora del gen o si el mismo material transferido contiene una o más mutaciones.
Puesto que los pseudogenes guardan una gran similitud con el gen activo, puede producirse un emparejamiento erróneo. La recombinación entre el pseudogen y el gen activo podría transferir una mutación desde el pseudogen al gen activo.
Correlación genotipo - fenotipo - Genotype - Phenotype correlation
Asociación entre determinada mutación o mutaciones (genotipo) y el fenotipo resultante.
Las posibles conclusiones sobre las correlaciones genotipo-fenotipo son las siguientes:
La estrecha correlación entre el genotipo y el fenotipo permite predecir el fenotipo de forma fiable, basándose en el genotipo.
La severidad y las manifestaciones clínicas específicas del fenotipo no se pueden predecir con seguridad debido a la amplia variabilidad de presentaciones fenotípicas entre los individuos que tienen el mismo genotipo.
No se han realizado estudios de correlación genotipo-fenotipo o bien éstos no son viables en la práctica debido al elevado número de mutaciones diferentes posibles.
A la inversa, la pregunta para la correlación fenotipo-genotipo es la siguiente: dado un fenotipo determinado ¿cuál es el genotipo más probable? En este caso, la evaluación del fenotipo sirve de orientación al médico/laboratorio respecto a las técnicas más apropiadas para la detección de mutaciones.
Ejemplo: Síndrome de deleción 22q11.2
Manifestaciones clínicas: Defectos cardíacos (en particular conotruncales), ausencia o función anormal del timo y/o las glándulas paratiroideas, paladar fisurado, rasgos faciales característicos, trastornos del habla y retraso del desarrollo.
Correlación genotipo-fenotipo: BAJA Un feto en el que se ha identificado la deleción 22q11.2 podría presentar todas las manifestaciones clínicas asociadas, sólo algunas de ellas o sólo trastornos del habla. No se puede predecir el fenotipo basándose en el genotipo.
Correlación fenotipo-genotipo: ALTA Niño nacido con defecto cardíaco conotruncal y ausencia de timo. La probabilidad de detectar una deleción en 22q11.2 es aproximadamente del 95%. El genotipo se puede predecir con seguridad basándose en el fenotipo.
Cosegregación - Cosegregation
Transmisión conjunta de dos o más genes ligados en un mismo cromosoma.
Ejemplo de cosegregación y recombinación: Pareja de cromosomas en meiosis que contienen los marcadores A, B y C en uno de los dos cromosomas homólogos y a, b y c en el otro. Recombinación entre los loci A y B (y a y b) que están muy separados; cosegregación de los loci B y C (y b y c) que están estrechamente ligados.
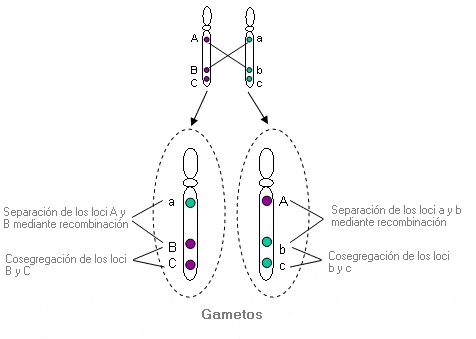
Debido a la gran distancia entre A y B (y entre a y b), la probabilidad de recombinación entre los dos loci es alta. Puesto que B y C (y b y c) están más juntos, la probabilidad de recombinación entre estos loci es baja; por tanto, B y C (y b y c) tienden a ser heredados juntos en el mismo cromosoma (es decir, cosegregan en la meiosis) con más frecuencia que A y B (y a y b).
Cribado - Screening
Pruebas destinadas a identificar a los individuos en una población determinada que presentan un riesgo más alto de tener o desarrollar un trastorno específico o de presentar una mutación génica por la cual desarrollarían un trastorno específico.
En el diagrama siguiente se representan las pruebas de detección prenatal del síndrome de Down, defectos del tubo neural abiertos y la trisomía 18 en la población embarazada, basadas en el análisis de marcadores séricos maternos.
Dependiendo de la prueba realizada, es posible que no se detecten alteraciones en algunos individuos afectados (resultado falso negativo), o que se identifiquen en individuos que no están afectados (resultado falso positivo).
1000 mujeres embarazadas (en total) sometidas al análisis de marcadores séricos maternos para la detección de posibles trastornos.
*Tasa de detección positiva, la sensibilidad y la especificidad varían en función del punto de corte de riesgo positivo de la prueba de detección y del número de análisis evaluados y determinados por cada laboratorio. 
Cribado neonatal - Newborn screening
Pruebas realizadas en los días siguientes al nacimiento con objeto de identificar si un neonato presenta un mayor riesgo de desarrollar un trastorno genético específico y de ser así, iniciar lo antes posible un tratamiento. Cuando el resultado de una prueba de cribado neonatal es positivo, normalmente es necesario realizar otros estudios diagnósticos para confirmar o concretar los resultados y ofrecer asesoramiento a los padres.
- Hiperplasia suprarrenal congénita
- Hipotiroidismo congénito
- Fibrosis quística
- Galactosemia
- Homocistinuria
- Enfermedad de la orina en jarabe de arce (MSUD)
- Deficiencia de acil-coenzima A deshidrogenasa de cadena media (MCAD)
- Fenilcetonuria (PKU)
- Anemia de células falciformes
- Tirosemia
Cromátidas - Chromatids
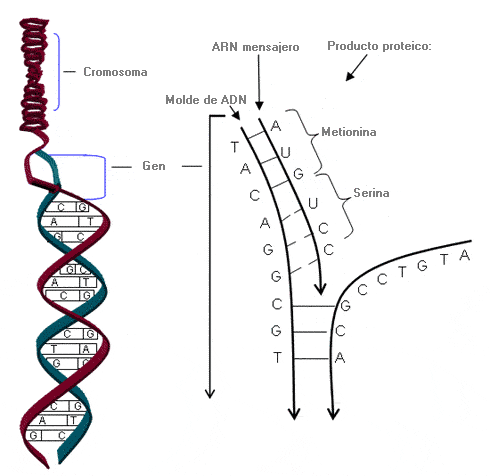
Cromosoma - Chromosome
Estructura física, también llamada cromatina, que consiste en una molécula de adn compactado organizada en genes y mantenida por proteínas llamadas histonas.
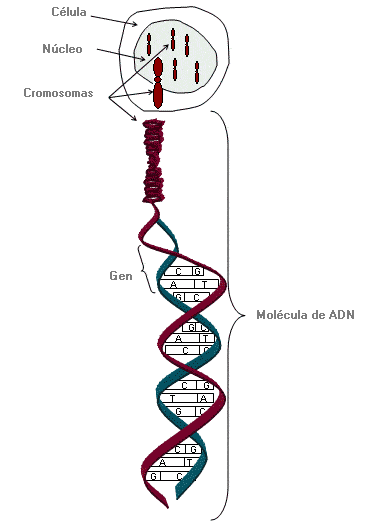 Los cromosomas están localizados en el núcleo de una célula. Presentan una estructura en forma de hélice compacta, excepto durante el proceso de división celular en el que se alargan.
Los cromosomas están localizados en el núcleo de una célula. Presentan una estructura en forma de hélice compacta, excepto durante el proceso de división celular en el que se alargan.
Las células humanas contienen normalmente 46 cromosomas dispuestos en 23 pares, 22 de los cuales son autosomas (cromosomas no sexuales) y un par son los cromosomas sexuales.
Diagrama adaptado de “Understanding Genetic Testing”, National Institutes of Health
Cromosoma derivado - Derivative chromosome
Término que se utiliza para designar un cromosoma cuya composición o morfología difiere del patrón normal debido a su participación en algún tipo de reordenamiento cromosómico como, por ejemplo una deleción, una traslocación entre 2 o más cromosomas o una inserción.
Cromosoma marcador - Marker chromosome
Cromosoma cuya morfología no corresponde al patrón estándar y cuyo origen no puede ser explicado de forma evidente a partir de un reordenamiento cromosómico. Un cromosoma marcador tiene significación clínica variable dependiendo de su contenido génico. La evaluación de su posible significación clínica puede resultar complicada, en particular si el cromosoma se detecta en estudios prenatales.
Cromosoma supernumerario - Supernumerary chromosome
Cromosoma de pequeño tamaño que contiene un centrómero y se observa ocasionalmente en cultivos celulares, generalmente en estado de mosaicismo (es decir, está presente en algunas células pero no en otras). Un cromosoma supernumerario tiene significación clínica variable dependiendo de su contenido génico. La evaluación de su posible significación clínica puede resultar complicada, en particular si el cromosoma se detecta en estudios prenatales.
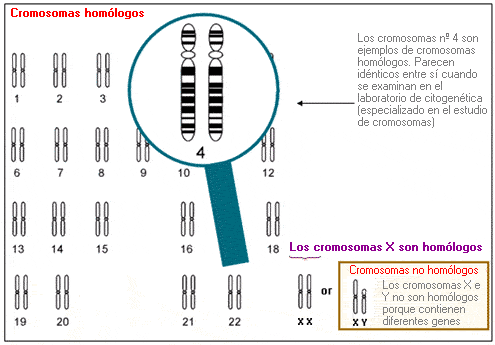
Cromosomas homólogos - Homologous chromosomes (sinónimo: homólogos)
Los dos cromosomas que constituyen una pareja cromosómica, uno heredado de la madre y el otro del padre, y que contienen los mismos loci genéticos en idéntico orden.
En los varones, cada par de cromosomas autosómicos es homólogo. Los cromosomas sexuales (X e Y) no son homólogos, puesto que contienen diferentes genes.
CSGE - (Conformation-sensitive gel electrophoresis) (sinónimo: variante de electroforesis en gel sensible a la conformación)
Cuello de botella - Population Bottleneck
Drástico descenso en el número de miembros de una población.
D
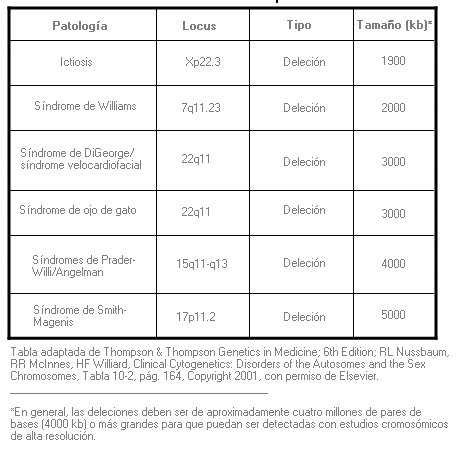
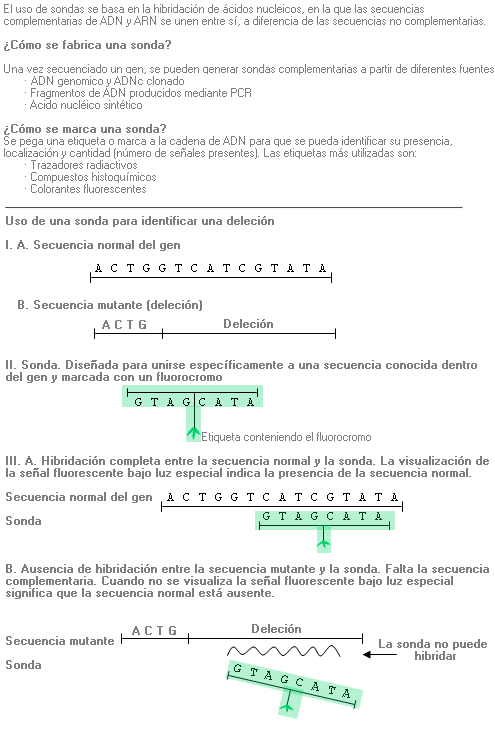
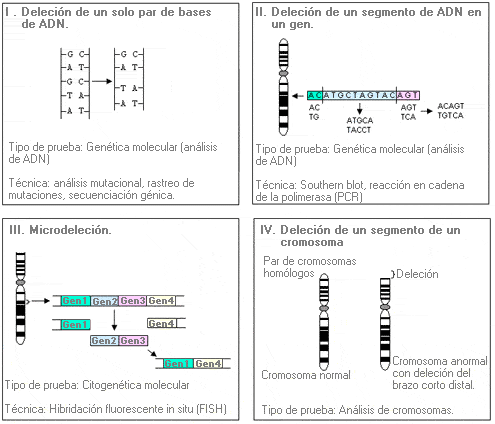
Deleción - Deletion
Alteración genética consistente en la pérdida de un segmento de ADN. Su magnitud es variable, pudiendo ser tan pequeñas como un solo par de bases o tan grandes que afecten a uno o más genes.
Densitometría - Densitometry
Método para identificar la dosis o la expresión génica mediante la medición de la absorción de la luz en una autorradiografía (película) de una banda (o mancha) que representa el ADN, el ARN o una muestra de proteínas. Es una técnica útil para detectar mutaciones por duplicación y mutaciones por deleción heterocigótica.
Deriva genética - Genetic Drift
Fluctuación (aumento o disminución) en la frecuencia de una variante genética determinada que se produce en una población concreta por azar. Típicamente ocurre en poblaciones pequeñas y aisladas y es común tras un fenómeno de “cuello de botella” en el que el tamaño de una población se reduce drásticamente, pudiéndose perder alelos raros y disminuir el pool genético. La deriva genética puede provocar que una población nueva sea genéticamente diferente de la población de origen y es uno de los mecanismos básicos en la evolución de las especies.
Desequilibrio de ligamiento - Linkage disequilibrium
En una población, la herencia simultánea y transmitida de un conjunto de marcadores de adn específicos a lo largo de generaciones sucesivas sin que se produzca recombinación entre ellos.
DGGE - (Denaturing gradient gel electrophoresis) (sinónimo: Electroforesis en gel con gradiente desnaturalizante)
Técnica de electroforesis para muestras de adn de cadena doble que utiliza un gel en el que se crea un gradiente desnaturalizante mediante una sustancia química, por ejemplo, urea. Se emplea para la identificación de mutaciones que afectan al patrón de migración del adn en esas condiciones de electroforesis. La presencia de la mutación se detecta mediante comparación del patrón electroforético (punto de desnaturalización) de la muestra problema con la secuencia salvaje u original.
DHPLC (Denaturing high-performance liquid chromatography) -
Cromatografía líquida desnaturalizante de alto rendimiento. En genética es un método utilizado para la detección de mutaciones de uno o pocos nucleótidos. La base de la técnica es la diferencia de migración que presentan fragmentos homodúplex (secuencia doble de ADN que presenta un 100% de apareamiento entre las bases) frente a los heterodúplex (apareamiento no total) durante su migración por un gradiente en una columna.
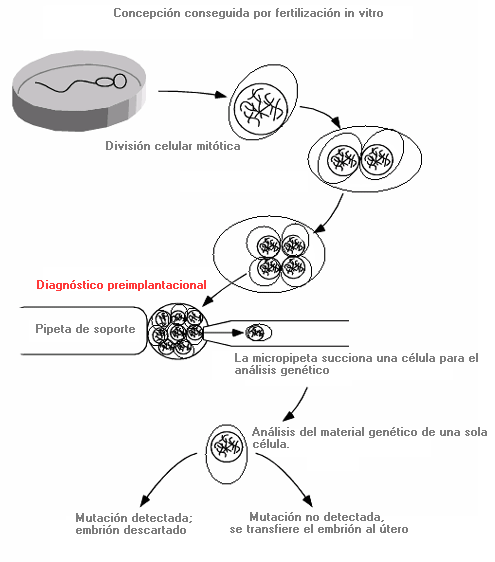
Diagnóstico preimplantacional - Preimplantation diagnosis (sinónimo: pruebas preimplantación)
Procedimiento de análisis o diagnóstico genético que se utiliza para estudiar la presencia de una patología genética determinada en un embrión. El estudio se realiza mediante el análisis de una célula extraída en las fases precoces del desarrollo del embrión (blastómero), todo ello en el entorno de una concepción realizada mediante fertilización in vitro. Una vez concluido el estudio se pueden transferir al útero de la madre aquellos embriones no portadores de las alteraciones genéticas analizadas.
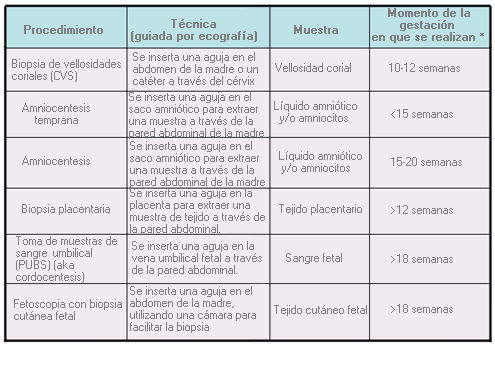
Diagnóstico prenatal - Prenatal diagnosis
Pruebas realizadas durante el embarazo para determinar si el feto está afectado por una determinada patología genética, cromosómica o de otra naturaleza. La biopsia de vellosidades coriales (CVS), la amniocentesis, la recogida de sangre umbilical por funiculocentesis (PUBS), la ecografía y la fetoscopia, son ejemplos de los procedimientos realizados para obtener muestras para análisis o para evaluar la anatomía fetal.
Examen mediante ecografía fetal: La ecografía permite el examen del feto mediante ondas de sonido de alta frecuencia. Se utiliza rutinariamente para calcular la edad fetal y detectar embarazos múltiples. También puede realizarse en el primer y segundo trimestre para detectar anomalías, tales como onfalocele, o signos indicativos de una enfermedad genética subyacente. Los hallazgos de la ecografía fetal pueden indicar la necesidad de realizar pruebas adicionales y/o solicitar asesoramiento genético.
Pruebas de cribado basadas con múltiples marcadores (que también se conocen como pruebas de cribado de AFP ampliadas, perfil de riesgo del embarazo o triple o cuádruple cribado). Este conjunto de pruebas de cribado prenatal incluyen la determinación de los niveles de alfafetoproteína (AFP), gonadotropina coriónica humana (hCG), estriol no conjugado (uE3) y, dependiendo del laboratorio en el que se realicen, inhibina-A (DIA), en el suero de las mujeres embarazadas. Estas pruebas se realizan normalmente entre las semanas 15 y 17 de la gestación, para evaluar el riesgo de anomalías fetales, incluyendo defectos abiertos del tubo neural, defectos de la pared abdominal, síndrome de Down y trisomía 18.
Diploide - Diploid
Dismorfología - Dysmorphology
Estudio clínico de los síndromes malformativos.
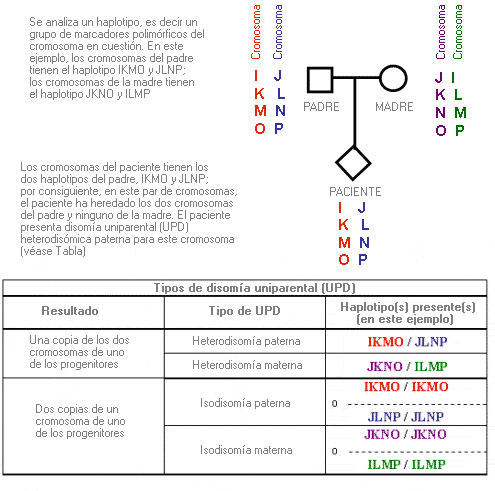
Disomía uniparental - Uniparental disomy (sinónimo: UPD)
Situación en la que un par de cromosomas homólogos o algún segmento de un par de cromosomas homólogos son heredados de sólo uno de los progenitores. La disomía uniparental puede dar lugar a un fenotipo anormal en algunos casos. También se ha descrito disomía uniparental somática que afecta sólo a las células que componen un determinado tejido. En estos casos el origen de la disomía no es la herencia directa sino un proceso anómalo de replicación del ADN.
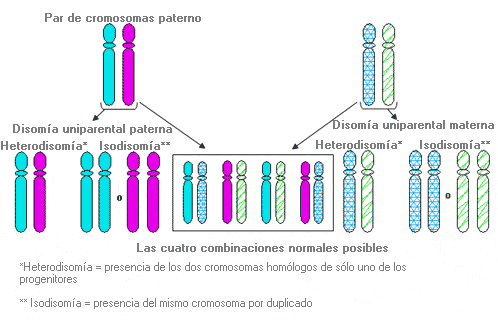
La disomía uniparental da lugar a un fenotipo anormal cuando los cromosomas implicados llevan impronta, por lo que los genes de estos cromosomas sólo tienen un alelo activo (es decir, sólo el alelo materno o paterno del par es activo).
Síndromes asociados con disomía uniparental (UPD)
Síndrome de Prader-Willi (UPD15 materno)
Síndrome de Angelman (UPD15 paterno)
Diabetes mellitus neonatal transitoria (UPD6 paterno)
Síndrome de Russell-Silver (UPD7 materno)
Síndrome de Beckwith-Wiedeman (UPD11 materno)
Síndrome de MUPD14 (UPD14 materno)
Dominante - Dominant
Dominante ligado al cromosoma X - X-linked dominant
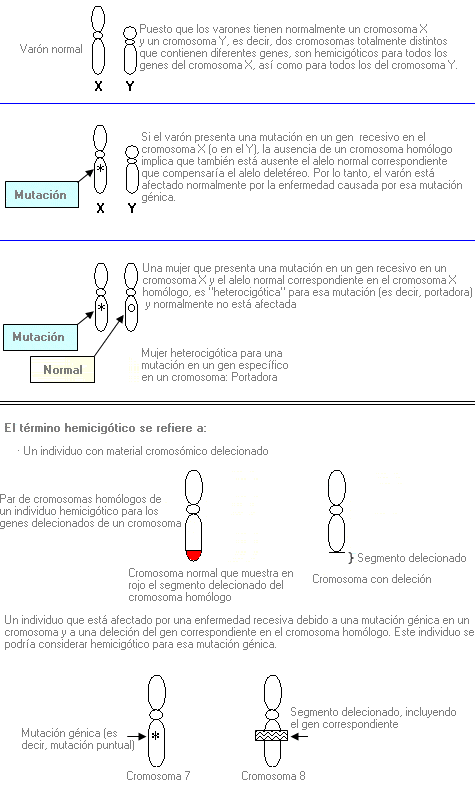
Describe un rasgo o trastorno de manifestación dominante causado por una [GLOSARIO]mutación[FIN_GLOSARIO] en un gen del cromosoma X. El fenotipo está expresado en mujeres heterocigóticas, así como en varones hemicigóticos (que sólo tienen un cromosoma X); los varones afectados suelen presentar un fenotipo más severo que las mujeres afectadas.

Probabilidad de transmisión de una mutación de un gen dominante ligado al cromosoma X.

Dominio - Domain
Región específica o secuencia de aminoácidos de una proteína, que está asociada a una función determinada o al segmento de adn correspondiente.
Duplicación - Duplication
Modificación de la secuencia genética (patológica o no) consistente en la presencia de un segmento adicional de adn que da lugar a copias repetidas de una parte de un gen, un gen entero o una serie de genes. Su origen más probable es un entrecruzamiento desigual durante la replicación del segmento duplicado durante la formación de los gametos en la meiosis.
E
Edición génica o genómica - Gene/genome editing
Técnica que permite a los investigadores alterar el ADN de las células u organismos para insertar, eliminar o modificar un gen o secuencia de genes para silenciar, mejorar o cambiar las características del gen.
La edición del genoma o la ingeniería del genoma es un tipo de ingeniería genética en la que el ADN se altera en el genoma de una célula u organismo vivo usando nucleasas modificadas genéticamente o "tijeras moleculares". Estas nucleasas crean roturas de doble cadena específicas de sitio (double strand breaks, DSB) en ubicaciones deseadas en el genoma. Las roturas inducidas de doble cadena se reparan a través de los sistemas de reparación de “non homology en joining” (NHEJ) o la recombinación homóloga (HR), dando como resultado mutaciones dirigidas.
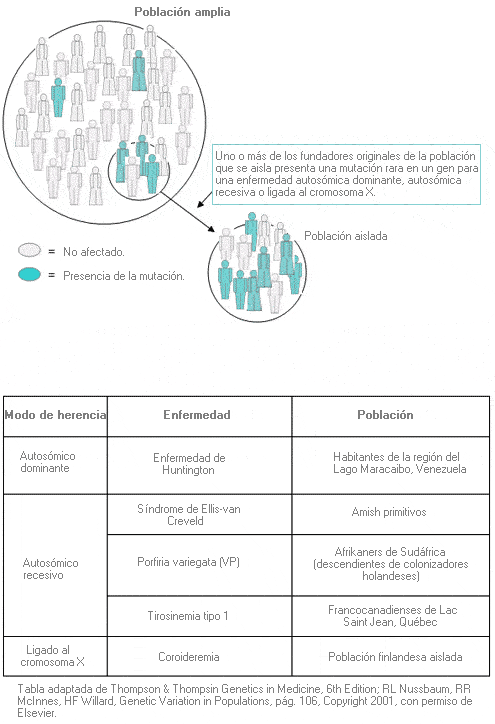
Efecto fundador - Founder effect
Mutación producida en un gen que se observa ligada a un haplotipo común que se presenta con una frecuencia alta en una población específica. Su origen está en la aparición de la mutación en un solo ancestro o en un número reducido de ancestros que se mantiene ligada al mismo haplotipo en todos los individuos que la poseen.
Eficacia biológica - Biological fitness
Eficacia es la representación cuantitativa de la selección natural y sexual dentro de la biología evolutiva. Se puede definir ya sea con respecto a un genotipo o a un fenotipo en un ambiente dado. En cualquier caso, describe el éxito reproductivo individual y es igual a la contribución promedio al conjunto de genes de la siguiente generación que realizan los individuos del genotipo o fenotipo especificado.
Electroforesis - Electrophoresis
Electroforesis en gel con gradiente desnaturalizante - Denaturing gradient gel electrophoresis (sinónimo: DGGE)
Técnica de electroforesis para muestras de ADN de cadena doble que utiliza un gel en el que se crea un gradiente desnaturalizante mediante una sustancia química, por ejemplo, urea. Se emplea para la identificación de mutaciones que afectan al patrón de migración del ADN en esas condiciones de electroforesis. La presencia de la mutación se detecta mediante comparación del patrón electroforético (punto de desnaturalización) de la muestra problema con la secuencia salvaje u original.
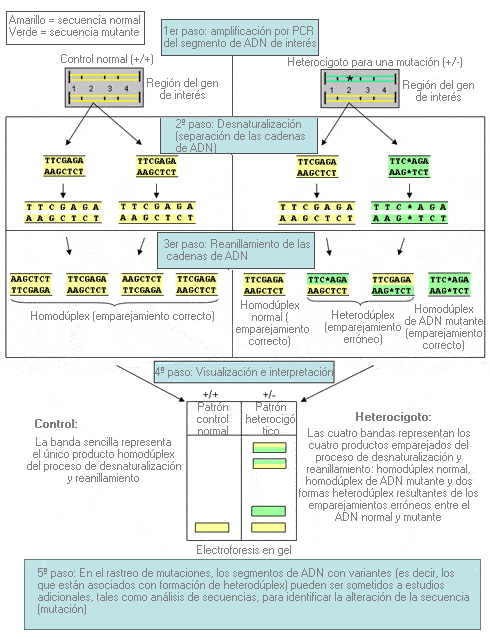
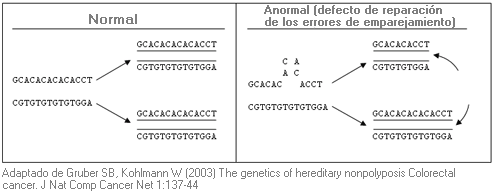
Electroforesis en gel sensible a conformación - Conformation-sensitive gel electrophoresis (sinónimo: CSGE)
Técnica electroforética que consiste en el análisis de un segmento de ADN para detectar errores de emparejamiento entre pares de bases normales y mutadas (ver explicación esquemática). Se emplea para el rastreo de mutaciones.
Elemento GC - GC element
Elemento de promotores eucariontes con secuencia consenso 5'-GGGCGGG-3' encontrados upstream respecto del sitio de inicio de la transcripción.
Elemento silenciador - Silencing element
Elemento regulador transcripcional de eucariotas que disminuyen la transcripción en vez de estimularla como los elementos de tipo enhancer.
Embrionaria (célula madre) - Embryonic stem cells (ESC)
Líneas celulares cultivadas que se establecen desde embriones muy tempranos y que son esencialmente totipotentes; es decir, estas células se pueden implantar en un embrión huésped y poblar muchos o todos los tejidos del animal en desarrollo, lo que incluye la línea germinal.
Endógeno - Endogenous
Se origina desde dentro de una célula o un organismo.
Endonucleasa - Endonuclease
Enzima que cataliza el corte de los enlaces fosfodiéster del interior de una cadena nucleotídica.
Endonucleasa de restricción - Restriction endonuclease
Una enzima de restricción o endonucleasa de restricción es una enzima que escinde ADN en fragmentos en o cerca de sitios de reconocimiento específicos dentro de la molécula conocida como sitios de restricción. Las enzimas de restricción se clasifican comúnmente en cuatro tipos. Para cortar el ADN, todas las enzimas de restricción hacen dos incisiones, una a través de cada cadena principal de azúcar-fosfato (es decir, cada cadena) de la doble hélice de ADN.
Endopoliploidia - Endopolyploidy
Estado poliploide en el que los cromosomas de una célula se han dividido y duplicado repetidamente sin sufrir división del núcleo ni de la célula.
Enfoque isoeléctrico - Isoelectric focusing
Método de detección de mutaciones basado en la separación de proteínas en función del valor de pH en el que su carga neta es cero (punto isoeléctrico). Se suele utilizar conjuntamente con la técnica western blot para permitir la identificación de productos proteicos de tipo salvaje frente a mutante. La alteración de una secuencia de adn que dé lugar a una sustitución de aminoácidos puede variar el punto isoeléctrico de una proteína.
Enlace fosfodiester - Phosphodiester bond
Enlace covalente en ácidos nucleicos que une a los grupos fosfatos de nucleótidos adyacentes, uniendo el C5' de una pentosa con el C3' de la otra pentosa. Los enlaces fosfodiesteres junto con los azúcares forman el esqueleto de las moléculas de ácidos nucleicos.
Enlace peptídico - Peptide bond
Enlace de tipo covalente que ocurre entre el grupo carboxilo de un primer aminoácido con el grupo amino de un segundo aminoácido. Así, las cadenas polipeptídicas crecen con la polaridad de extremo amino libre hacia extremo carboxilo libre.
Ensamblaje - Splicing
Mecanismo por el cual los intrones son eliminados durante el proceso de maduración de un ARN mensajero.
Ensayo enzimático - Enzyme assay
Determinación de la actividad enzimática con un sustrato dado; se puede evaluar de diferentes maneras, incluyendo la cuantificación del producto final o análisis colorimétrico.
Ensayo funcional de proteínas - Protein functional assay
Método para determinar la velocidad de una reacción química que se produce en presencia de una enzima contenida en una muestra tomada de un individuo. Una actividad enzimática reducida puede indicar el estado portador o el diagnóstico de una enfermedad genética determinada.
Entrecruzamiento - Crossing over (sinónimo: recombinación)
Intercambio de un segmento de adn entre los dos cromosomas homólogos durante la meiosis. Tiene como resultado una combinación nueva de material genético en el gameto.
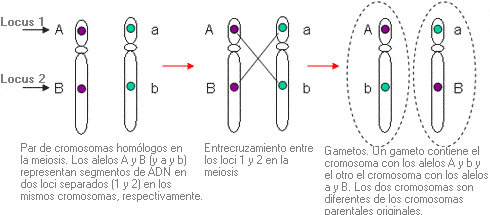 Dos genes localizados en el mismo cromosoma pueden segregarse juntos o no. El entrecruzamiento entre los cromosomas homólogos durante la meiosis da lugar a combinaciones nuevas de genes en el gameto, y por lo tanto, en los descendientes.
Dos genes localizados en el mismo cromosoma pueden segregarse juntos o no. El entrecruzamiento entre los cromosomas homólogos durante la meiosis da lugar a combinaciones nuevas de genes en el gameto, y por lo tanto, en los descendientes.
Gracias al proceso de entrecruzamiento, cada cromosoma transmitido de un progenitor a su descendiente contiene algunos segmentos heredados de las dos ramas ascendientes de ese progenitor. Por ello, el entrecruzamiento desempeña un papel importante asegurando la variabilidad genética entre individuos, incluso de la misma familia.
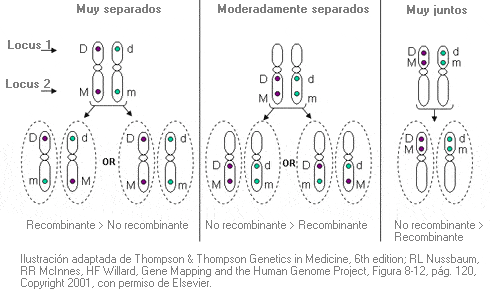
El comportamiento durante la meiosis de los alelos en dos loci (1 y 2) del mismo cromosoma depende de su proximidad.
Enzima - Enzyme
Molécula de naturaleza proteica que promueve o permite que tenga lugar una reacción química en las células (una reacción bioquímica). Las enzimas son esenciales para la correcta función del metabolismo del cuerpo.
Epidemiología genética - Genetic epidemiology
La epidemiología genética estudia la interacción entre los factores genéticos y ambientales que dan origen a las enfermedades del ser humano. Utiliza marcadores de ADN que son estudiados mediante análisis molecular. Dado el volumen de datos (que hacen referencia a poblaciones numerosas) lo habitual es que sean almacenados en bases de datos y tratados mediante herramientas bioinformáticas. Las estrategias de investigación incluyen, entre otras, análisis de segregación familiar, asociaciones alélicas y estudios de interacción gen y medio ambiente.
Epigenética - Epigenetics
Cambios reversibles de ADN (modificaciones químicas) que alteran la expresión de los genes que son objeto de los mismos. Los tipos de cambios epigenéticos más frecuentes incluyen la metilación de la citosina del ADN así como la metilación, acetilación y fosforilación de las proteínas histonas que conforman la cromatina. La metilación más estudiada es una modificación del ADN, en la que un grupo metilo es trasferido desde S-adenosilmetionina a una posición C-5 de citosina por una ADN-5 metiltransferasa. La metilación del ADN ocurre, casi exclusivamente, en dinucleótidos CpG, teniendo un importante papel en la regulación de la expresión del gen. Estos factores genéticos son determinados por el ambiente celular en lugar de por la herencia.
Episoma - Episome
Elementos genéticos replicantes extracromosómicos que se pueden replicar de forma autónoma o que pueden ser insertados (mediante un proceso de recombinación) en el cromosoma del organismo que los porta y replicarse con el mismo. Los episomas más conocidos son los plásmidos.
Epistasia - Epistasis
Interacción de varios genes al expresar un determinado carácter fenotípico. Se denomina epistasia cuando la expresión de uno o más genes dependen de la expresión de otro gen.
Epitopo - Epitope
La parte de una molécula de antígeno que es reconocida por una inmunoglobulina específica.
Escisión reparación - Excision repair
Especificidad - Specificity
Esporádico - Sporadic
La aparición casual de un trastorno o anomalía en un individuo cuyo riesgo de recurrencia no es probable en su familia.
Los trastornos esporádicos pueden ser de etiología genética o no genética.
Trastornos esporádicos de etiología genética
Disomía uniparental – El individuo afectado ha heredado los dos genes o cromosomas de un mismo par de cromosomas procedentes de uno de sus progenitores y ninguno del otro. No es probable que la aparición casual de una anomalía genética implique un riesgo de recurrencia en otros miembros de la familia.
Tumores esporádicos – Todos los tumores son de origen genético a nivel celular; sin embargo, NO todos son hereditarios. Es probable que un único tumor en un individuo de más edad esté causado por mutaciones adquiridas (no heredadas) de genes reguladores celulares, que dan lugar a una proliferación celular incontrolada.
Trastornos esporádicos de etiología no genética:
Síndrome de rubeola congénita en un niño cuya madre ha padecido rubeola durante el embarazo
Ataxia causada por alcoholismo
El término “esporádico” puede ser utilizado vagamente (e incorrectamente) para referirse a lo siguiente:
Trastornos autosómicos dominantes o ligados al cromosoma X que aparecen en un solo individuo de la familia, generalmente a consecuencia de una mutación nueva. Puesto que el riesgo de recurrencia aumenta en los hermanos del individuo afectado y es de hasta un 50% en los descendientes de este individuo, el uso del término “esporádico” no es apropiado. En este caso, el término correcto que debe utilizarse es “caso único”.
Estudio de disomía uniparental - Uniparental disomy study (sinónimos: análisis UPD, estudio UPD)
Pruebas realizadas para identificar si algún par de cromosomas o de segmentos específicos de un cromosoma han sido heredados ambos de la madre o del padre. Estos estudios pueden servir para confirmar el diagnóstico clínico de determinados trastornos en los que la UPD es una posible etiología subyacente.
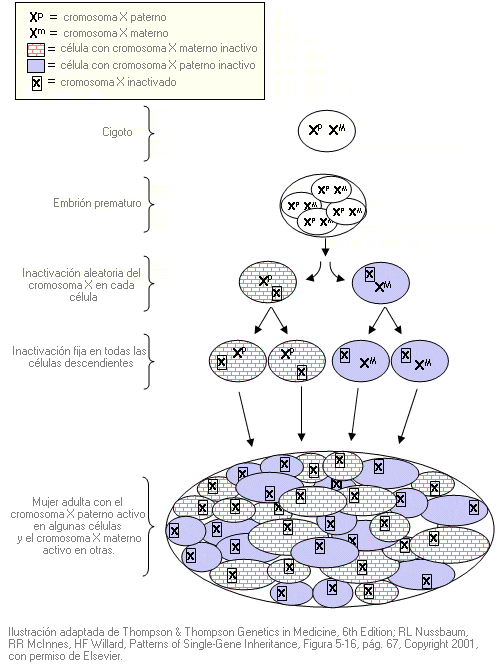
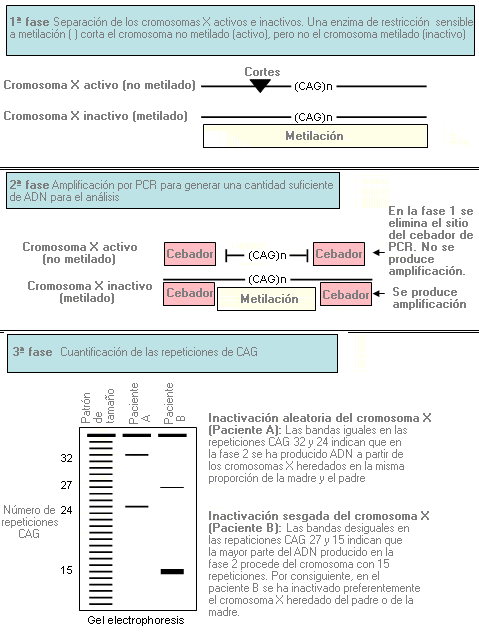
Estudio de inactivación del cromosoma X - X-chromosome inactivation study
Estudios de genética molecular para evaluar la proporción relativa de cromosomas X inactivos (metilados) y activos (no metilados). Se utiliza para determinar si la inactivación del cromosoma X es aleatoria o sesgada.
Un ejemplo de estudio de inactivación es analizar el tamaño de las repeticiones de trinucleótidos CAG polimórficas en el gen del receptor de andrógenos (AR) localizado en el cromosoma X, para determinar si la inactivación de este cromosoma es aleatoria o sesgada. El 90% de las mujeres son heterocigóticas para el número de repeticiones CAG en los dos cromosomas X (es decir, el número de repeticiones CAG en el cromosoma X heredado de la madre difiere del número observado en el cromosoma X heredado del padre). Cuando el número de repeticiones CAG en los dos cromosomas X de una mujer es diferente, se puede utilizar este número para determinar si la inactivación del cromosoma X es aleatoria (es decir, la misma proporción de cromosomas X heredados del padre y la madre están inactivados) o si es sesgada (es decir, uno de los cromosomas X se ha inactivado preferentemente).
Estudio de predisposición - Predispositional testing
Pruebas realizadas a un individuo asintomático en el que la identificación de una mutación indicará que es probable, pero no seguro, que ese individuo desarrolle en el futuro síntomas relacionados con una enfermedad específica. Un resultado negativo no descarta la posibilidad de que el individuo desarrolle la enfermedad en un futuro por otras causas.
Estudios cromosómicos de alta resolución - High-resolution chromosome studies
Análisis del número y la estructura de los cromosomas cuando se ha detenido el proceso de división celular y se han teñido los cromosomas en una fase temprana de la mitosis (prometafase). En un estudio de alta resolución, los cromosomas parecen más largos y muestran de 700 a 1200 bandas, lo que permite un análisis más detallado de la estructura de los cromosomas, a diferencia de la técnica de bandas en metafase rutinaria, en la que aparecen normalmente de 300 a 600 bandas.
Estudios de cigosidad - Zygosity testing
Proceso mediante el cual se comparan las secuencias de adn para determinar si los individuos nacidos en una gestación múltiple (mellizos, trillizos, etc.) son monocigóticos (idénticos) o dicigóticos (fraternales). Estos estudios se utilizan frecuentemente para identificar un donante apropiado para trasplante de órganos o para calcular el riesgo de susceptibilidad a desarrollar una enfermedad.
Estudios de genética molecular - Molecular genetic testing (sinónimos: análisis de ADN, análisis basados en ADN, estudios moleculares)
Pruebas que implican el estudio del ADN, mediante análisis de ligamiento, secuenciación o detección de mutaciones, utilizando uno o varios de los diferentes métodos disponibles.
Estudios de inestabilidad de microsatélites - Microsatellite instability testing (sinónimo: estudios MSI)
Análisis genéticos realizados en muestras tumorales que detectan la deficiencia en el sistema de reparación de errores de emparejamiento de bases del ADN. La deficiencia se manifiesta al comparar el número de repeticiones de nucleótidos en un grupo de marcadores tipo microsatélite en una muestra problema (tumor) frente al número observado en una muestra normal del mismo paciente. Se considera que hay estabilidad de microsatélites (MSS) cuando el número de repeticiones en cada marcador, tanto en el tejido tumoral como en el normal, es el mismo. Existe inestabilidad de microsatélites (MSI) cuando el número de repeticiones en el tejido tumoral y el normal es diferente.
Replicación de microsatélites
Procedimiento
Se extrae ADN de tejido tumoral y normal del mismo individuo.
Se evalúa un grupo de marcadores microsatélites. Generalmente, este grupo incluye los marcadores BAT25, BAT26, D2S123, D5S346 y D17S250; la mayoría de los laboratorios clínicos utilizan un grupo de 10 marcadores.
Interpretación1
Variación del número de repeticiones de nucleótidos entre el ADN extraído de tejido tumoral y normal:
MSI de frecuencia alta (MSI-H): 30% de los marcadores analizados muestran diferencias entre el ADN tumoral y el normal.
MSI de frecuencia baja (MSI-L): Mayor que cero, pero 30% de los marcadores analizados muestran diferencias.
Estabilidad de los microsatélites (MSS): Ninguno de los marcadores analizados muestra diferencias.
Seguimiento.
El hallazgo de inestabilidad de microsatélites (MSI) en el tumor de un individuo con cáncer de colon o endometrio diagnosticado a una edad temprana, con unos antecedentes familiares de cáncer relacionado con HNPCC o la presencia de tumores primarios múltiples en el mismo individuo, sugiere una mutación en línea germinal en un gen de reparación de errores de emparejamiento (p. ej. MLH1, MSH2, MSH6, PMS2, PMS1). En estos casos están indicados los estudios de genética molecular para la búsqueda de mutaciones en línea germinal.
Puesto que aproximadamente el 20% de los cánceres de colon esporádicos son MSI-H debido a mutaciones somáticas o a la metilación del gen MLH1, la edad a la que se desarrolla el tumor y los antecedentes familiares son factores a tener en cuenta a la hora de distinguir entre los cánceres de colon esporádicos que son MSI-H y los que están relacionados con HNPCC.
1The National Cancer Institute Workshop on Microsatellite Instability and RER Phenotypes in Cancer Detection and Familial Predisposition MSI testing recommendations include [Boland et al 1998]
Estudios de portadores - Carrier testing (sinónimos: detección de portadores, estudios de heterocigotos)
Pruebas que se realizan para identificar a los individuos asintomáticos que presentan una mutación en un gen causante de una enfermedad autosómica recesiva o recesiva ligada al cromosoma X.
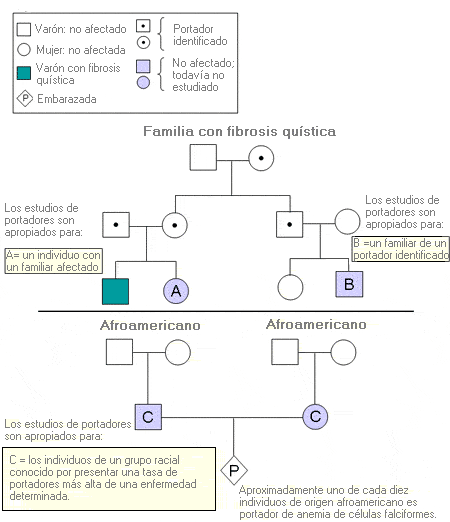 Los estudios de portadores se proponen a los individuos (A) con familiares afectados por una enfermedad genética; (B) con familiares que son portadores identificados y (C) que pertenecen a grupos étnicos o raciales conocidos por presentar una tasa de portadores más alta para una patología particular.
Los estudios de portadores se proponen a los individuos (A) con familiares afectados por una enfermedad genética; (B) con familiares que son portadores identificados y (C) que pertenecen a grupos étnicos o raciales conocidos por presentar una tasa de portadores más alta para una patología particular.
Estudios de radiosensibilidad - Radiosensitivity testing (sinónimos: ensayo de supervivencia de colonias, CSA)
Pruebas específicas para detección de ataxia-telangiectasia (AT) y otros trastornos de origen genético en los que las células son particularmente sensibles a la radiación ionizante. Se ha demostrado que la formación de colonias en una línea celular de linfocitos de sangre periférica tras irradiación es anormal en aproximadamente el 99% de los pacientes con diagnóstico clínico de AT.
Estudios de repeticiones de trinucleótidos - Trinucleotide repeat testing
Análisis genético cuyo objetivo es la cuantificación del número de repeticiones de grupos de tres nucleótidos en un segmento determinado de ADN.
Para cuantificar las repeticiones de trinucleótidos, se pueden utilizar dos tipos de análisis mutacional que se representan en el diagrama siguiente:
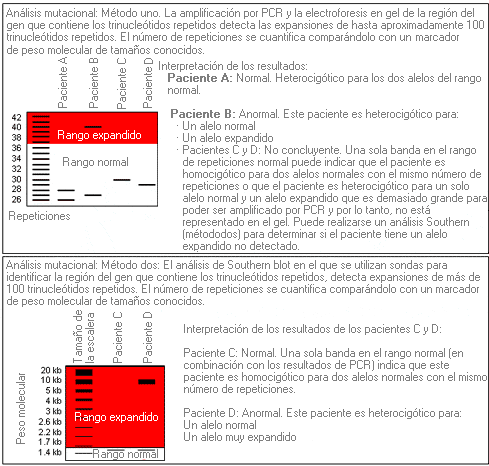
Algunas implicaciones clínicas:
El análisis Southern blot no se realiza de forma rutinaria en todos los laboratorios para clarificar la interpretación de los resultados de las bandas no concluyentes por PCR. Cuando se reciba un informe de un número de repeticiones “homocigóticas” normales, particularmente en un niño sintomático de corta edad que podría presentar una expansión grande, es recomendable que el médico confirme que se ha realizado el análisis Southern blot para evaluar la posibilidad de que haya una repetición de trinucleótidos muy expandida.
Si no está disponible el análisis Southern blot, se puede realizar un análisis mutacional basado en PCR/electroforesis en gel de los padres de un paciente con un número de repeticiones “homocigóticas” normales, para determinar si el paciente podría haber heredado de sus padres dos alelos con el mismo número de repeticiones.
Estudios diagnósticos - Diagnostic testing
Pruebas realizadas con objeto de confirmar o descartar una enfermedad genética conocida o que se sospecha en un individuo sintomático o, prenatalmente, en un feto con riesgo de desarrollar una patología genética específica.
Algunos ejemplos clínicos de estudios diagnósticos.
Distrofia muscular de Duchenne (DMD): Dado que se detectan deleciones o duplicaciones en el gen DMD en el 65% de los casos, el análisis de ADN de este gen en muestras de sangre es un procedimiento diagnóstico primario menos costoso e invasivo que el análisis de distrofina en biopsia muscular.
Hemocromatosis hereditaria (HHC): Dado que las manifestaciones clínicas y/o la saturación de transferrina sérica y la concentración de hierro sérico son compatibles con HHC, los estudios de genética molecular resultan menos costosos e invasivos que la biopsia hepática para confirmar el diagnóstico. Las mutaciones patológicas en HFE conocidas se detectan en aproximadamente el 87% de los casos de HHC.
Fibrosis quística. Mediante el estudio de las mutaciones en CFTR más comunes, se podrán detectar mutaciones en aproximadamente el 90% de los pacientes caucasianos descendientes de judíos no askenazi; sin embargo, en los individuos de cualquier etnia, los análisis de cloruro en muestras de sudor son una prueba diagnóstica más exacta.
Estudios predictivos - Predictive testing
Pruebas que se proponen a individuos asintomáticos que tienen antecedentes familiares de una patología genética y presentan un riesgo potencial de desarrollar esa patología con el tiempo.
Los dos tipos de estudios predictivos son los siguientes:
Estudios presintomáticos: Pruebas realizadas en un individuo asintomático en el que la detección de una mutación génica indica que con seguridad, ese individuo desarrollará en algún momento de su vida, síntomas relacionados con una enfermedad específica. Por el contrario, el resultado negativo descarta el desarrollo de la enfermedad en cuestión.
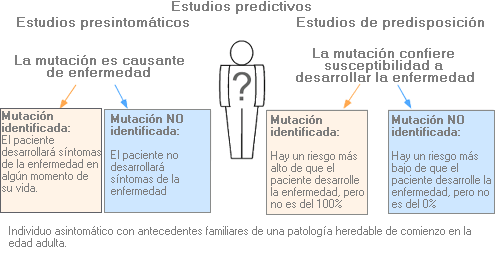
Estudios de predisposición: Pruebas realizadas en un individuo asintomático en el que la detección una mutación génica indica que es probable, pero no seguro, que ese individuo desarrollará con el tiempo síntomas relacionados con una enfermedad específica. El resultado negativo no descarta la posibilidad de que el individuo desarrolle la enfermedad en el futuro por otras causas.
Algunas implicaciones clínicas.
Las estudios predictivos ESTAN INDICADOS CLÍNICAMENTE si un diagnóstico precoz posibilita una intervención que reduzca la morbilidad o mortalidad.
Incluso en ausencia de indicaciones clínicas, los estudios predictivos pueden influir en las decisiones para planificar la vida.
Puesto que los estudios predictivos pueden tener repercusiones psicológicas, la evaluación, asesoramiento y seguimiento minuciosos del paciente son aspectos importantes.
Muchos laboratorios no realizan estos estudios sin contar previamente con un consentimiento informado y si no disponen de un servicio de asesoramiento genético.
Antes de realizar los estudios predictivos, se debe haber identificado la mutación específica en algún familiar afectado o haber establecido el ligamiento dentro de la familia.
Es desaconsejable realizar estudios predictivos en niños asintomáticos con riesgo de desarrollar enfermedades de comienzo en la edad adulta, cuando no son posibles intervenciones médicas que mejoren el manejo o seguimiento de estos pacientes.
Estudios prenatales individualizados - Custom prenatal testing
Pruebas de diagnóstico prenatal ofrecidas a familias en las que se han identificado mutaciones patológicas en un miembro afectado.
Estudios presintomáticos - Presymptomatic testing
Pruebas realizadas a un individuo asintomático en el que la identificación de una mutación va a indicar que es seguro que el individuo en cuestión desarrollará síntomas relacionados con esa enfermedad específica. Un resultado negativo descarta la probabilidad de que el individuo desarrolle la enfermedad.
Etiqueta de secuencia expresada - Expressed sequence tag, EST
Una etiqueta de secuencia expresada (o "expressed sequence tag", EST) es una subsecuencia corta de secuencia conocida dentro de una secuencia de cADN.
Eucromatina - Euchromatin
Forma de la cromatina ligeramente compactada (a diferencia de la heterocromatina que son regiones más compactadas). Las regiones eucromáticas suelen presentar una gran concentración de genes y generalmente son regiones transcripcionalmente activas.
Euploidía - Euploidy
Alteración numérica que afecta al número completo de juegos cromosómicos de una especie. Se suele producir por un reparto desigual de los cromosomas durante la meiosis, normalmente cuando no hay separación de los cromosomas homólogos.
Evaluación del riesgo - Risk assessment
Cálculo por medio de ecuaciones matemáticas apropiadas del riesgo de que un individuo haya heredado una mutación génica determinada, de que desarrolle un trastorno específico, o de que tenga un hijo con un trastorno determinado. El cálculo se basa en el análisis de múltiples factores, incluyendo la historia clínica familiar y los antecedentes étnicos entre otros.
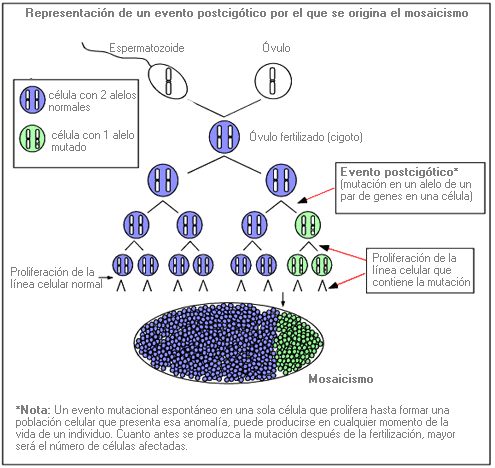
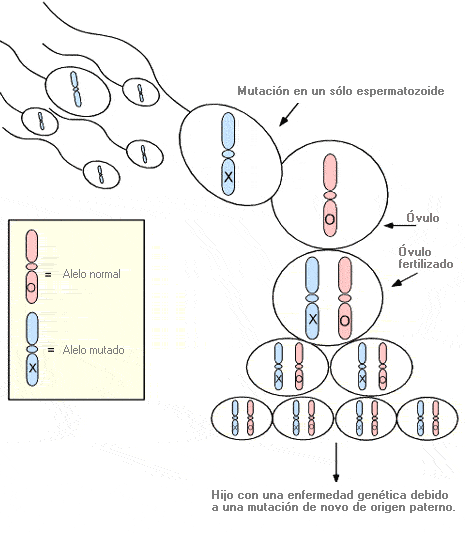
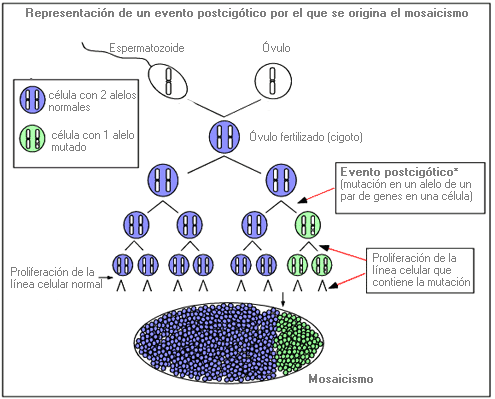
Evento post-cigótico - Post-zygotic event
Acontecimiento mutacional o alteración en la replicación/segregración de los cromosomas, que se produce una vez que un óvulo ha sido fertilizado por un espermatozoide y generalmente da lugar a mosaicismo (presencia de dos o más líneas celulares genéticamente distintas en el mismo organismo).
Exógeno - Exogenous
Introducido u originado desde el exterior de una célula o un organismo.
Exón - Exon
Secuencia codificante de ADN. El exón es la región de un gen que no se separa durante el proceso de splicing (corte y empalme) manteniéndose en el ARN mensajero.
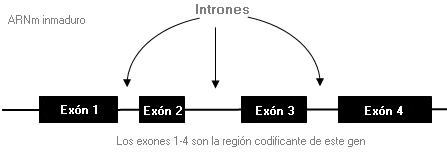
Figura 1. El ADN transcrito inicialmente a ARN mensajero inmaduro (ARNm) consta de secuencias codificantes (exones) y no codificantes (intrones).
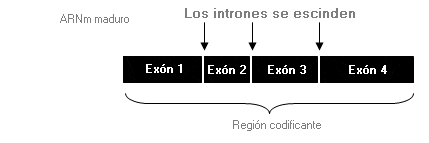
Figura 2. Los intrones se han escindido del ARNm inmaduro para formar ARNm maduro, con lo cual quedan sólo los exones que codificarán finalmente el producto aminoacídico.
Ilustraciones adaptadas de Thompson & Thompson Genetics in Medicine, 6th edition; RL Nussbaum, RR McInnes, HF Willard, The Human Genome: Structure and Function of Genes and Chromosomes, Figura 3-6, pág. 20, Copyright 2001, con permiso de Elsevier.
Exonucleasa - Exonuclease
Enzimas que catalizan la rotura de enlaces fosfodiéster en los extremos de cadenas nucleotídicas.
Expresión ectópica - Ectopic expression
Expresión de un gen en un tejido en el que normalmente no se expresa. Esta expresión ectópica puede ser causada por la yuxtaposición de nuevos elementos reguladores (enhancers o moduladores) a un gen.
Expresión génica - Gene expression
Proceso mediante el cual el ARN y las proteínas se elaboran a partir de las instrucciones codificadas en los genes. La expresión génica está controlada por proteínas y moléculas de ARN que se unen al genoma o al ARN y regulan sus niveles de producción y los de sus productos. Las alteraciones en la expresión génica cambian las funciones de las células, tejidos, órganos u organismos completos.
Expresividad - Expressivity
Es la fuerza o grado con el que se manifiesta un gen particular en el fenotipo.
Expresividad variable - Variable expressivity
Variación de las manifestaciones clínicas (tipo y severidad) de un trastorno genético entre individuos afectados, incluso de la misma familia.
Síndrome de Treacher Collins como ejemplo de expresividad variable
Patrón de herencia: Autosómica dominante Gen: TCOF1 Locus: 5q32-q33.1
Manifestaciones clínicas principales:
Fisuras palpebrales en sentido oblicuo y hacia abajo
Coloboma palpebral
Ausencia parcial o total de pestañas en el párpado inferior
Hipoplasia malar
Paladar fisurado
Hipoplasia mandibular
Deformidades auriculares
Pérdida de audición
Posibles explicaciones para la expresividad variable:
Diferentes mutaciones en el mismo gen (heterogeneidad alélica)
Diferentes mutaciones en varios genes en diferentes locus (heterogeneidad genética)
Existencia de genes modificadores (otros genes que afectan a la expresión del gen de interés)
Factores medioambientales
Factores desconocidos
¿Cuál es la diferencia entre expresividad variable y penetrancia?
La expresividad variable se refiere a la diversidad de manifestaciones clínicas observadas en individuos que presentan un trastorno determinado. El término expresividad variable se aplica a los trastornos que siguen todos los patrones de herencia.
La penetrancia es la proporción de individuos con una mutación patológica que presentan manifestaciones clínicas del trastorno. El término penetrancia se aplica normalmente a los trastornos autosómicos dominantes.
¿De qué forma influye la expresividad variable en la penetrancia?
La penetrancia depende de las manifestaciones de la enfermedad que se utilizan para determinar si un individuo está afectado. Por ejemplo, se puede suponer que los individuos con manifestaciones leves de un trastorno autosómico dominante no están afectados, por lo que la penetrancia reducida del trastorno es aparente. Consideremos lo siguiente:
Se sabe que una paciente presenta una mutación en FBN1 causante del síndrome de Marfan porque tanto su padre como su hijo están afectados. Los resultados de la exploración física de esta paciente son normales y se cree que no presenta manifestaciones del síndrome de Marfan. Sin embargo, el ecocardiograma revela dilatación de la raíz aórtica, que es una manifestación clínica característica del síndrome de Marfan. Los estudios basados en los resultados de la exploración física y el ecocardiograma revelarán una penetrancia más alta del síndrome de Marfan que los estudios basados sólo en la exploración física.
F
F1 -
Progenie de la primera generación filial de un cruce.
F2 -
Progenie de la segunda generación filial de un cruce, producida por el entrecruzamiento de individuos de la F1.
Factor de transcripción - Transcription factor
Proteína que facilita o da inicio a la transcripción de uno o varios genes.
Falso negativo - False negative
Falso positivo - False positive
Resultado de una prueba que indica que un individuo está afectado y/o presenta una mutación génica determinada, cuando en realidad no está afectado y/o no presenta tal mutación; es decir, cuando el resultado de una prueba realizada en un individuo que realmente no está afectado es positivo.
Familia génica - Gene family
Conjunto de genes similares originados por duplicación a partir de un gen ancestral común. Generalmente tienen funciones bioquímicas similares.
Familiar - Familial
Una característica o condición que tiende a ser heredada en familias.
Farmacogenética - Pharmacogenetics
Disciplina genética que se ocupa de investigar el vínculo entre la constitución genética de las personas y su respuesta a xenobióticos (drogas). Su método de investigación tiene una hipótesis definida a priori: está basado en el análisis de marcadores genéticos predeterminados de los que sospecha previamente que tengan una incidencia directa sobre el metabolismo de las drogas.
El concepto de farmacogenética surge de la observación clínica de que existían pacientes a los que, tras administrarles una determinada droga, la concentración observada en plasma u orina era anormalmente variable. Tras realizar en esos pacientes pruebas bioquímicas y metabólicas se demostraba que estas variaciones tienen origen genético. Su principal objetivo es poder anticipar la efectividad del tratamiento indicado a cada individuo y minimizar, simultáneamente, los eventos adversos.
Farmacogenómica - Pharmacogenomics
Disciplina genética que se ocupa de investigar las diferentes reacciones de los individuos a los fármacos basándose en los patrones genéticos de cada uno. Su método de investigación está basado en una aproximación distinta a la farmacogenética. No hay hipótesis a priori sobre marcadores genéticos candidatos. Se utilizan abordajes que tienen en cuenta las características de las secuencias genómicas (de miles de genes o marcadores), mediante una visión integradora que incluiría interacciones entre genes El objetivo de la farmacogenómica (también compartido por la farmacogenética) es la creación de fármacos a medida para cada paciente y adaptados a sus condiciones genéticas.
Fenilalanina - Phenylalanine
La fenilalanina es un aminoácido (abreviado frecuentemente como Phe o F). Se encuentra en las proteínas como L-fenilalanina (LFA), siendo uno de los 10 aminoácidos esenciales para el ser humano. Los codones que la expresan son UUC y UUU.
Fenocopia - Phenocopy
Variación en el fenotipo no hereditaria (generalmente referido a un único rasgo) que es causado por condiciones ambientales. Imita a un fenotipo producido por un genotipo específico. No es un tipo de mutación, ya que no es hereditaria.
Fenotipaje o tipificación fenotípica - Phenotyping
Clasificación de la diferencias fenotípicas entre individuos con la intención de inferir el genotipo.
Fenotipo - Phenotype
Características físicas y/o bioquímicas observables de la expresión de uno o varios genes. Conjunto de rasgos clínicos de un individuo con un genotipo determinado.
Algunas implicaciones clínicas
En algunas patologías, numerosos genotipos pueden dar lugar a un fenotipo similar debido a heterogeneidad alélica y/o de locus.
En algunas enfermedades, puede observarse una gran variabilidad fenotípica entre individuos que tienen un genotipo idéntico, es decir, la correlación genotipo-fenotipo es baja (véase expresividad variable).
FISH - Fluorescent in situ hybridization (sinónimo: hibridación in situ con fluorescencia)
Técnica que se utiliza para identificar, en una muestra problema, la presencia de cromosomas determinados o regiones específicas de cromosomas cuya secuencia es conocida, (sondas de ADN), y que marcamos con fluorescencia. La detección se realiza tras la hibridación (reconocimiento de la secuencia y emparejamiento base a base) de la Sonda de ADN marcada sobre el ADN cromosómico desnaturalizado de la muestra problema.
1ª fase. Preparación de la sonda. Una sonda es un segmento de ADN marcado con fluorescencia que es complementario a la región cromosómica de interés.
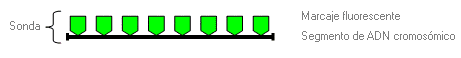
2ª fase. Hibridación. Los cromosomas desnaturalizados, fijados en el portaobjetos del microscopio, son expuestos a la sonda marcada con fluorescencia. La hibridación (fusión) tiene lugar entre la sonda y el ADN cromosómico complementario (es decir, se acopla).
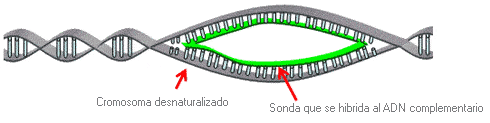
3ª fase. Visualización. Tras la hibridación, se examina el portaobjetos al microscopio con luz fluorescente. Las señales fluorescentes indican la presencia de ADN cromosómico complementario, la ausencia de tales señales implica que no hay ADN cromosómico complementario.
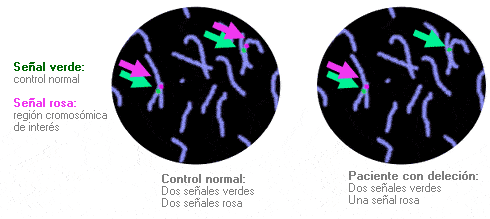
FISH en interfase - Interphasic FISH
Técnica que se utiliza para identificar, en una muestra problema, la presencia de cromosomas determinados o regiones específicas de cromosomas cuya secuencia es conocida, (sondas de ADN), y que marcamos con fluorescencia. La detección se realiza tras la hibridación (reconocimiento de la secuencia y emparejamiento base a base) de la sonda de ADN marcada sobre el ADN cromosómico desnaturalizado de la muestra problema. En la FISH en interfase, las sondas se introducen en el núcleo interfásico. La FISH en interfase se utiliza, por ejemplo para detectar tipos específicos de aneuploidía en células fetales, así como determinadas deleciones, duplicaciones y otras anomalías en células tumorales. Al contrario que la FISH en metafase, la FISH en interfase no permite visualizar los cromosomas individualizados; por lo tanto, no se podrán detectar determinados reordenamientos estructurales.
FISH en metafase - Metaphase FISH
Técnica que se utiliza para identificar, en una muestra problema, la presencia de cromosomas determinados o regiones específicas de cromosomas cuya secuencia es conocida, (sondas de ADN, y que marcamos con fluorescencia). La detección se realiza tras la hibridación (reconocimiento de la secuencia y emparejamiento base a base) de la sonda de ADN marcada sobre el ADN cromosómico desnaturalizado de la muestra problema. En la FISH en metafase, se utiliza como muestra cromosomas obtenidos a partir de células en mitosis. La división celular tiene varias fases y en la fase denominada metafase, los cromosomas se condensan y se pueden distinguir de forma individual. Al contrario que la FISH en interfase, la FISH en metafase permite visualizar la hibridación de las sondas de ADN marcado sobre cromosomas individualizados, lo que facilita la localización de la anomalía en el cromosoma específico en el que se produce.
FISH subtelomérica - Subtelomeric FISH
Técnica en la que se utilizan sondas de ADN específicas para las áreas subteloméricas de los brazos largos y cortos de cada cromosoma, que permite la detección de deleciones y translocaciones subteloméricas crípticas y submicroscópicas, que pueden ser causa de retraso mental.
Flujo genético - Gene flow
Intercambio o transferencia de variación genética entre dos poblaciones de la misma especie. Origina cambios en las frecuencias alélicas de ambas poblaciones, y es una posible causa de evolución. Se produce como resultado de la migración.
Footprinting - Footprinting
Técnica empleada para la identificación de secuencias de ADN que ligan proteínas específicas. Al unirse la proteína ésta protege algunos enlaces fosfodiesteres del ADN y por lo tanto también protege de la acción de ADNasas.
Frecuencia alélica - Allele frequency (sinónimo: frecuencia génica)
Proporción de individuos de una población que han heredado una mutación o variante génica específica.
Frecuencia de portadores - Carrier rate
Proporción de individuos en una población que tienen una sola copia de una variante genética (mutación o polimorfismo).
Los individuos portadores de una sola mutación de un gen recesivo no se pueden identificar normalmente por su fenotipo (es decir, no están afectados); la frecuencia de portadores se puede calcular utilizando la ley de Hardy-Weinberg cuando se conoce la incidencia de la enfermedad (es decir, la frecuencia de homocigotos).

Cálculo de la tasa de portadores (2pq) para una enfermedad autosómica recesiva utilizando la ley de Hardy-Weinberg cuando se conoce la frecuencia de la enfermedad (q2):
Frecuencia de la enfermedad (q2)=1/2000; Puesto que p+q = 1; p =1-0,022 = 0,978.
Por lo tanto, 2pq = 2(0,978)(0,022) = 0,043 o aproximadamente 4%.
La tasa de portadores de esta enfermedad recesiva en particular es de aproximadamente un 4% o de 1 entre 25 individuos, dada una frecuencia o incidencia de la patología en esta población de 1 entre 2000.
Las tasas de portadores se pueden determinar de forma similar en enfermedades recesivas ligadas al cromosoma X.
Frecuencia de recombinación - Recombination frequency
Parámetro genético que se emplea como indicador cuantitativo de la distancia que existe entre dos loci dados. Se mide en centiMorgan (cM).
G
Gametogénesis - Gametogenesis (sinónimos: oogénesis, espermatogénesis)
Proceso meiótico mediante el cual se forman los gametos maduros (óvulos y espermatozoides). Los términos oogénesis y espermatogénesis se refieren a la producción de óvulos y de espermatozoides, respectivamente.
Ganancia de función - Gain of function
Tipo de mutación que resulta en un producto genético alterado que posee una nueva función molecular o un nuevo patrón de expresión genética
Gap - Gap
Ausencia de una o más bases en una de las hebras del DNA duplex.
Gemelos dicigóticos - Dizygous tweens
Dos hermanos nacidos del mismo embarazo y desarrollados a partir de dos óvulos independientes fecundados al mismo tiempo. Pueden ser del mismo sexo o del sexo opuesto, diferir tanto física como genéticamente.
Gemelos monocigóticos - Monozygous tweens
Dos hermanos nacidos del mismo embarazo y desarrolladas a partir de un solo óvulo fertilizado que se divide en dos mitades iguales durante una fase temprana de escisión en el desarrollo embrionario, dando lugar a fetos separados. Dichos gemelos son siempre del mismo sexo, tienen la misma constitución genética, poseen grupos sanguíneos idénticos y se asemejan mucho en características físicas, psicológicas y mentales.
Gen - Gene
Gen constitutivo - Housekeeping gene
Gen que codifica para una proteína indispensable para la función celular. Se expresa en todas o la mayoría de las estirpes celulares. También se los conoce como genes estructurales, en oposición a los genes específicos de tejido.
Gen de fusión - Fusion gene
Unión accidental del ADN de dos genes. Los genes de fusión pueden ocurrir como resultado de una translocación, deleción o inversión cromosómica. Las fusiones génicas pueden dar lugar a proteínas híbridas o a la regulación incorrecta de la unidad de transcripción de un gen por los elementos reguladores en cis (potenciadores) de otro.
Gen de susceptibilidad - Susceptibility gene
Gen que cuando sufre una mutación incrementa la probabilidad de que un individuo desarrolle determinada enfermedad o trastorno.
Gen dominante - Dominant gene
Gen que produce un efecto (o fenotipo) en el organismo independientemente del estado del alelo correspondiente.
Gen letal - Lethal gene
Gen que produce un genotipo que lleva a la muerte del organismo antes de que la reproducción sea posible o imposibilita la reproducción; para un gen recesivo, el estado homocigoto o hemicigótico es letal.
Gen ligado al sexo - Sex-linked gene
Gen localizado en un cromosoma sexual, especialmente un cromosoma X.
Gen mapeado - Mapped gene
Gen para el que se ha establecido su posición relativa en un segmento de ADN o en un cromosoma.
Gen recesivo - Recesive gene
Gen que produce un efecto en el organismo solo cuando es transmitido por ambos padres, es decir, solo cuando el individuo es homocigoto.
Gen regulador - Regulator gene
Gen involucrado en el control de la expresión de uno o más genes.
Gen reportero - Reporter gene
Gen cuyo producto es fácilmente detectable o medible y, por lo tanto, puede usarse como un indicador de si una construcción de ADN se ha transferido con éxito. Por ejemplo: cloranfenicol transacetilasa.
Gen supresor de tumores - Tumour suppressor gene
Gen implicado en procesos de crecimiento y/o diferenciación celular. Cuando este gen sufre una mutación que causa una pérdida o reducción en su función, la célula puede progresar a cáncer, generalmente en combinación con otros cambios genéticos.
Genealogía - Genealogy
Pedigrí. Historia familiar. Diagrama que muestra las relaciones y características de individuos de una misma familia durante dos o más generaciones.
Generación filial - Filial generation
Generación de individuos productos de cruzamientos. La primera generación se denomina F1, la segunda generación F2 y así sucesivamente. Son relativos a la generación parental.
Genética - Genetics
Disciplina científica que estudia el significado, propiedades y función del material genético.
Genética inversa - Reverse genetics
Método utilizado para ayudar a comprender la función de un gen mediante el análisis de los efectos fenotípicos de secuencias genéticas modificadas.
Genoma - Genome
Conjunto de los genes de un individuo o una especie contenida en un juego haploide de cromosomas.
Genómica - Genomics
La genómica se encarga del estudio de los genomas. Se considera a un genoma como el conjunto de información genética (ADN) de un organismo. A diferencia de la genética clásica, en la que a partir de un fenotipo se busca el o los genes responsables de dicho fenotipo, la genómica tiene como objetivo predecir la función de los genes a partir de su secuencia. Para conseguir este tipo de objetivos emplea herramientas basadas en análisis genéticos masivos (del orden de miles de genes) frecuentemente de forma simultánea (en un reducido número de experimentos).
Genómica funcional - Functional Genomics
La genómica funcional se ocupa de recoger de forma sistemática datos e información sobre la función de genes. Estos datos son obtenidos mediante la aplicación de aproximaciones experimentales globales (genómicas). Se caracteriza por la combinación de metodologías experimentales a gran escala con estudios computacionales de los resultados.
Genotipaje - Genotyping
Pruebas que revelan los alelos específicos heredados por un individuo; son especialmente útiles en situaciones en las que más de una combinación de genotipos puede dar lugar a la misma presentación clínica, como en el caso del grupo sanguíneo ABO, en el que los genotipos AO y AA dan lugar al grupo sanguíneo A.
Genotipo - Genotype
Constitución genética de un organismo o célula; se refiere también al grupo específico de alelos heredados en un locus.
Glicina - Glycine
La glicina o glicocola (Gly, G) es uno de los aminoácidos que forman las proteínas de los seres vivos. En el código genético está representada por los codones GGU, GGC, GGA o GGG. Es el aminoácido más pequeño y el único no quiral de los 20 aminoácidos presentes en la célula. La glicina es un aminoácido no esencial.
Glutamina - Glutamine
La glutamina (abreviada Gln o Q, y con frecuencia nombrada como L-glutamina) es uno de los 20 aminoácidos que intervienen en la composición de las proteínas y que tienen codones referentes en el código genético. Está codificada en el ARN mensajero como 'CAA' o 'CAG'. Se trata de un aminoácido no esencial.
Guanina - Guanine
Una de las cuatro bases que componen el ADN. Las otras tres son adenina, citosina y timina. La guanina siempre se empareja con la citosina.
Guía RNA - Guide RNA
Segmentos cortos de ARN utilizados para dirigir la enzima de corte de ADN a la ubicación de destino en el genoma. Los segmentos de gARN contienen la región de homología con la secuencia diana (normalmente 20 bases) y una secuencia que interacciona con la nucleasa (por ejemplo, Cas9). Los gARN utilizados en la edición del genoma son sintéticos y no ocurren en la naturaleza.
H
Haploide - Haploid
La mitad del número diploide o normal de cromosomas en una célula somática. Coincide con el número de cromosomas en un gameto (óvulo o espermatozoide), que son 23 en el ser humano; un cromosoma de cada par.
Haploinsuficiencia - Haploinsufficiency
Situación en la que un individuo está clínicamente afectado porque una sola copia del gen normal es incapaz de producir proteína en cantidad o calidad suficiente para asegurar la función normal. Puede deberse a que el individuo es heterocigótico para determinada mutación génica o es hemicigótico en un locus particular, debido normalmente a una deleción del alelo correspondiente.
Haplotipo - Haplotype
Conjunto de alelos contenidos en un locus (o en varios loci) de una misma dotación haploide. El haplotipo podemos referirlo a un solo locus o a un genoma completo pero siempre se refiere a uno de los dos alelos de cada gen.
Hardy-Weinberg (Ley) - Hardy-Weinberg law
Extensión de las leyes Mendelianas que describen las frecuencias de alelos y de genotipos para poblaciones en equilibrio génico.
Helicasa - Helicase
Enzima que participa en el proceso de replicación de la molécula de ADN desenrrollando la doble hélice cerca del punto de bifurcación de la horquilla replicadora.
Hemicigoto - Hemizygous
Heredabilidad - Heritability
Estadístico utilizado para la estimación del grado de variación en un rasgo fenotípico en una población que se debe a la variación genética entre individuos en esa población. Proporción de la variación fenotípica que es debida a la variación genética total. Da una idea del grado en que un carácter fenotípico está determinado genéticamente.
Herencia cualitativa - Cualitative heritage
Codifica para características fenotípicas que se miden en escala cualitativa. Generalmente depende de un par de genes. Ejemplo: Color de ojos.
Herencia cuantitativa - Cuantitative heritage
Codifica para características fenotípicas que se miden en escala cuantitativa. También conocida como herencia poligénica. Ejemplo: peso, altura, intensidad.
Herencia mitocondrial - Mitochondrial inheritance
Las mitocondrias son organelas citoplasmáticas que producen ATP (la energía que origina la mayoría de las reacciones químicas en el organismo) y contienen su propio genoma. Las mutaciones producidas en los genes mitocondriales son responsables de diversos síndromes reconocidos y se heredan siempre de la madre, puesto que los óvulos contienen mitocondrias, mientras que los espermatozoides no.
Herencia multifactorial - Multifactorial inheritance (sinónimo: herencia poligénica)
Contribución combinada de uno o más genes y de factores medioambientales, generalmente desconocidos, al desarrollo de un rasgo o enfermedad determinada.
Herencia pseudodominante - Pseudodominant inheritance
Trastorno autosómico recesivo presente en dos o más generaciones de una misma familia; es decir, el patrón de herencia puede confundirse con un modelo dominante. Posibles explicaciones: 1- alta frecuencia de portadores en la población, y 2- consanguinidad en la familia.
Heterocigoto - Heterozygote
Individuo que tiene dos alelos diferentes en un locus, uno en cada cromosoma. En el caso de una mutación patológica, un alelo es normal y otro anormal.
Heterocigoto compuesto: Individuo que tiene dos alelos anormales o mutados diferentes en un mismo locus, uno en cada cromosoma; se refiere normalmente a los individuos afectados por una enfermedad autosómica recesiva.
Heterocigoto doble: Individuo que presenta dos mutaciones génicas diferentes (es decir, es heterocigótico) en dos loci genéticos distintos.
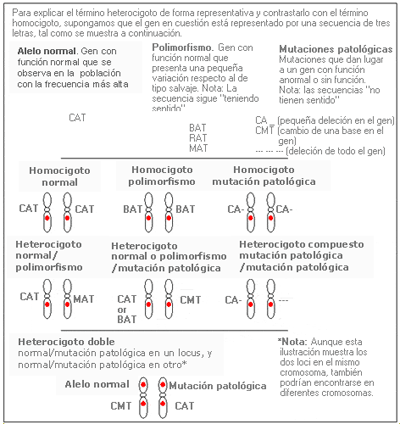
Heterocigoto compuesto - Compound heterozygote
Individuo que tiene dos alelos anormales o mutados diferentes en un mismo locus, uno en cada cromosoma; normalmente se refiere a los individuos afectados por una enfermedad autosómica recesiva.
Los hijos/as afectados de padres portadores de una mutación diferente en el mismo locus del gen son heterocigotos compuestos.
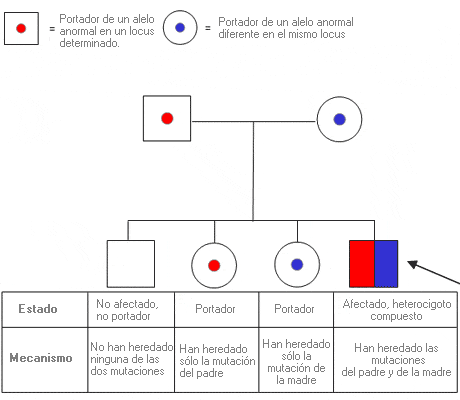
Heterocigoto doble - Double heterozygote
Individuo que es heterocigoto para mutación en dos loci genéticos diferentes. Es decir, es portador de dos mutaciones en genes distintos.
Cuando el padre y la madre presentan una mutación en dos loci genéticos diferentes, sus hijos pueden ser heterocigotos dobles. Los loci pueden estar en el mismo cromosoma o en diferentes cromosomas, según se muestra a continuación.
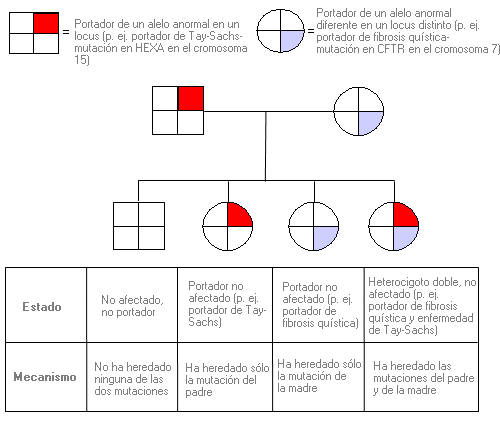
Nota: Puesto que en el ejemplo anterior se incluyen dos loci diferentes (dos alelos para cada uno), en cada hijo/a pueden darse 16 combinaciones posibles de los cuatro alelos de los padres.
Heterocigoto obligado - Obligate heterozygote (sinónimo: portador obligado)
Individuo que de acuerdo al análisis de los antecedentes familiares debe ser portador de una mutación genética, aunque clínicamente pueda no estar afectado.
Heterocromatina - Heterochromatin
Forma compacta de ADN, que se presenta en múltiples variedades. Estas variedades se encuentran en un continuo entre los dos extremos de heterocromatina constitutiva y facultativa.
Heteroduplex - Heteroduplex
Molécula híbrida de ácido nucleico en la que cada una de las hebras tiene un origen diferente.
Heterogeneidad alélica - Allelic heterogeneity (sinónimo: heterogeneidad molecular)
Heterogeneidad de locus - Locus heterogeneity
Situación en que las mutaciones producidas en Genes que se encuentran en diferentes loci cromosómicos dan lugar al mismo Fenotipo.
Ejemplo de heterogeneidad de locus: Retinitis pigmentosa (RP)
Fenotipo: Las anomalías de los fotorreceptores (bastoncillos y conos) de la retina dan lugar a una adaptación inadecuada a la oscuridad, causan pérdida de visión periférica y finalmente pérdida progresiva de visión.
Loci: Las mutaciones producidas en los genes en más de 35 loci diferentes dan lugar a retinitis pigmentosa no sindrómica que es clínicamnete indistinguible, por lo que no es posible deducir el gen alterado basándose sólo en el exámen ocular
Comparar y contrastar el concepto de heterogeneidad de locus con lo siguiente:
Heterogeneidad alélica: Un solo fenotipo originado por diferentes mutaciones en el mismo gen y locus cromosómico (Nota: algunos trastornos muestran heterogeneidad de locus, otros heterogeneidad alélica y otros ambas características)
Expresividad: Variación de las manifestaciones clínicas (tipo y severidad) de una enfermedad genética entre individuos que presentan la misma alteración genética, incluso dentro de la misma familia.
(Nota: La heterogeneidad de locus implica que los individuos con diferentes genotipos tienen el mismo fenotipo; mientras que la expresividad variable implica que los individuos con el mismo genotipo presenten un fenotipo que muestra diferencias tanto en la severidad como en la edad de comienzo de sus manifestaciones).
Heterogeneidad genética - Locus heterogeneity
Situación en que las mutaciones producidas en genes distintos dan lugar al mismo fenotipo.
Fenotipo: Las anomalías de los fotorreceptores (bastoncillos y conos) de la retina dan lugar a una adaptación inadecuada a la oscuridad, causan pérdida de visión periférica y finalmente pérdida progresiva de visión.
Loci implicados: mutaciones producidas en más de 35 genes diferentes dan lugar a retinitis pigmentosa no sindrómica, que es clínicamente indistinguible. A esto se debe que no sea posible deducir qué gen es el alterado basándose sólo en el examen ocular.
Heterogeneidad alélica: Un solo fenotipo originado por diferentes mutaciones en el mismo gen y locus cromosómico. (Nota: algunos trastornos muestran heterogeneidad genética, otros heterogeneidad alélica y otros ambas características).
Expresividad variable: variación de las manifestaciones clínicas (tipo y severidad) de una enfermedad genética entre individuos que presentan la misma alteración genética, incluso dentro de la misma familia.
(Nota: La heterogeneidad genética implica que los individuos con diferentes genotipos tienen el mismo fenotipo, mientras que la expresividad variable implica que los individuos con el mismo genotipo presenten un fenotipo que muestra diferencias tanto en la severidad como en la edad de comienzo de sus manifestaciones).
Heteromorfismo cromosómico - Chromosomal heteromorphism
Par de cromosomas homólogos que muestran alguna diferencia en forma o tamaño.
Heterosis - Heterosis
Aquella situación en la que el heterocigoto presenta superioridad respecto a los homocigotos para un carácter determinado.
Hibridación in situ con fluorescencia - Fluorescent in situ hybridization
Véase FISH
Híbrido - Hybrid
Resultante del cruzamiento entre un homocigoto dominante y un homocigoto recesivo.
Hiperploide - Hyperploid
Presencia de uno o más cromosomas o segmentos cromosómicos ganados respecto al estado normal (nótese la diferencia con "poliploide"). Dependiendo del grado de ploidía se puede definir estado hiperhaploide, hiperdiploide, hipertriploide, etc.
Hipoploide - Hypploid
Presencia de uno o más cromosomas o segmentos cromosómicos perdidos respecto al estado normal (nótese la diferencia con "haploide"). Dependiendo del grado de ploidía se puede definir estado hiperhaploide, hiperdiploide, hipertriploide, etc.
Histidina - Histidine
La histidina es un aminoácido esencial en animales (no puede ser fabricado por su propio organismo y debe ser ingerido en la dieta), mientras que bacterias, hongos y plantas pueden sintetizarlo internamente. Es uno de los 20 aminoácidos que forman parte de las proteínas codificadas genéticamente. Se abrevia como His o H. Los codones que la expresan son CAU y CAC.
Histonas - Histones
Proteínas altamente alcalinas que se encuentran en los núcleos de células eucarióticas y cuya función es el empaquetamiento y ordenamiento del ADN en unidades estructurales llamadas nucleosomas. Son los principales componentes proteicos de la cromatina, que actúan como carretes alrededor de los cuales se enrolla el ADN y desempeñan un papel en la regulación genética.
Homocigoto - Homozygote
Individuo que tiene dos alelos idénticos en un mismo locus determinado, uno en cada cromosoma.
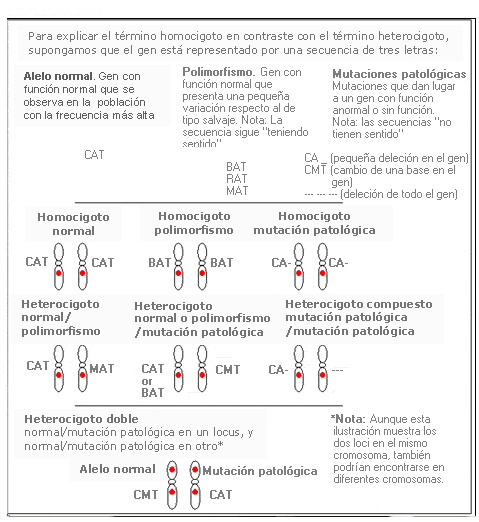
Homoduplex - Homoduplex
Secuencia de ácido nucleico donde el apareamiento entre las bases es de un 100%.
I
Impronta - Imprinting
Proceso mediante el cual los cromosomas de origen materno y paterno son modificados químicamente por separado. Esto da lugar a la expresión diferencial de determinados genes, dependiendo del origen parental del cromosoma.
*Las células somáticas de varones y mujeres contienen normalmente los cromosomas con impronta materna y paterna.
**En los gametos, la impronta se cambia de acuerdo a la siguiente regla: en los hijos varones, la impronta materna se transforma en impronta masculina en todos los espermatozoides, y en los descendientes de sexo femenino, la impronta paterna se transforma en impronta femenina en todos los óvulos. Este proceso de conversión está controlado por el “centro de impronta”, localizado en la misma región que el gen/genes regulados por impronta.
In vitro -
Referente a un experimento que tiene lugar fuera de un organismo vivo.
In vivo -
Referente a un experimento que tiene lugar en un organismo vivo.
Inactivación del cromosoma X - X-chromosome inactivation
Incidencia - Incidence
Número de nuevos pacientes o individuos que desarrollaron la enfermedad durante un cierto período de tiempo en una población específica.
Normalmente se expresa en forma de tasa (ver fórmula de abajo) por 100.000 habitantes y año:
Número de casos nuevos de enfermedad o evento.
Número de personas a riesgo de desarrollar la enfermedad por el tiempo que cada una de ellas permanece en riesgo.
Inducción - Induction
El inicio o intensificación de la síntesis de una enzima por parte de una célula que tiene lugar con la provisión del sustrato para la enzima.
Inductor - Inductor
Un agente ambiental que desencadena la transcripción de un operón.
Inestabilidad de microsatélites - Microsatellite instability (sinónimos : MSI, fenotipo de error de replicación, RER)
Discrepancia entre el tamaño de los microsatélites en el adn de tejido tumoral comparado con el no tumoral de la misma persona, a consecuencia de las mutaciones producidas en un gen en la vía de reparación de los errores de emparejamiento de bases del adn (MMR). Estos gen es si no estuviesen mutados corregirían normalmente estos errores.
Influido por el sexo - Sex-influenced
Rasgo fenotípico condicionado por el sexo del individuo sin que éste esté determinado por un gen ligado al sexo.
Informatividad - Informativeness
En relación a un marcador genético, un descendiente de una pareja será informativo cuando la probabilidad de que se pueda deducir el origen parental de cada uno de los alelos de un locus en concreto es alta.
Inhibición por contacto - Contact inhibition
Propiedad de las células en cultivo por la que dejan de proliferar cuando entran en contacto entre sí.
Inmunofluorescencia - Inmunofluorescence
Un método que determina la ubicación del antígeno o anticuerpo en un tejido mediante el patrón de fluorescencia que resulta cuando el tejido se expone al anticuerpo o antígeno específico marcado con un fluorocromo.
Inmunoglobulina - Inmunoglobulin
Ver anticuerpo.
Inmunohistoquímica - Inmunohistochemistry
Técnica que usa anticuerpos o antisueros como herramientas histológicas para identificar patrones de distribución de proteínas en un tejido o un organismo. Un anticuerpo (o mezcla de anticuerpos) que se une a una proteína específica se marca con una enzima que puede convertir un sustrato en un colorante visible. El anticuerpo etiquetado se incuba con el tejido, el anticuerpo no unido se elimina por lavado y luego se agrega el sustrato enzimático, revelando el patrón de localización de la proteína (antígeno).
Inmunosupresión - Inmunosuppression
La prevención o la disminución artificial de la respuesta inmune. Ésta puede lograrse por irradiación o por administración de antimetabolitos, suero anti-linfocitos o anticuerpos específicos.
Inserción - Insertion
Anomalía cromosómica en la que el material de un cromosoma se inserta en otro cromosoma; mutación en la que se inserta un segmento de ADN en un gen o en otro segmento de ADN, alterando potencialmente la secuencia codificante.
Integración - Integration
Proceso de recombinación que inserta una molécula de ADN pequeña en un locus más grande.
Interacción Ligando-Receptor - Ligand-Receptor Interaction
Las interacciones entre una molécula (generalmente de origen extracelular) y una proteína en o dentro de una célula diana.
Intercambio de cromátidas hermanas - Sister chromatid exchange
Intercambio de material genético entre dos cromátidas de un mismo cromosoma durante el proceso de división celular; es similar al entrecruzamiento (recombinación) con la diferencia de que el intercambio implica a dos cromátidas hermanas de un mismo cromosoma, mientras que el entrecruzamiento supone el intercambio de material genético entre los dos cromosomas homólogos.
Interfase - Interphase
La etapa del ciclo celular entre las divisiones nucleares, cuando los cromosomas se extienden y son funcionalmente activos.
Interferencia - Interference
Ocurrencia no aleatoria de cruces entre cromosomas homólogos durante la meiosis.
Interferón - Interferon
Una proteína que es sintetizada por células animales en respuesta a la infección viral y no inhibe específicamente la replicación de los virus. Se encuentra en el suero al inicio de la infección y mucho antes de la producción de los anticuerpos.
Intrón - Intron
Secuencia no codificante de ADN que se transcribe a ARN mensajero (ARNm) en su estado inmaduro, pero es escindida del mismo al transformarse en ARNm maduro antes de la traducción.

Figura 1. El ADN transcrito inicialmente a ARN mensajero inmaduro (ARNm) consta de secuencias codificadoras (exones) y no codificadoras (intrones).
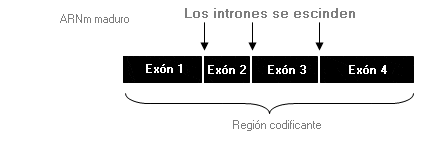
Figura 2. Los intrones se han escindido del ARNm inmaduro para formar ARNm maduro, con lo cual quedan sólo los exones que codificarán finalmente el producto del aminoácido.
Ilustraciones adaptadas de Thompson & Thompson Genetics in Medicine, 6th edition; RL Nussbaum, RR McInnes, HF Willard, The Human Genome: Structure and Function of Genes and Chromosomes, Figura 3-6, pág. 20, Copyright 2001, con permiso de Elsevier.
Inversión - Inversion
Reordenamientos cromosómicos en los que un segmento de un cromosoma se invierte totalmente y se reinserta en el mismo sitio de rotura del cromosoma. Las inversiones equilibradas (sin pérdida ni adición neta de material genético) no suelen estar asociadas con anomalías fenotípicas; sin embargo, en algunos casos, las alteraciones de un gen en los puntos de rotura pueden causar efectos fenotípicos adversos, incluyendo algunas enfermedades genéticas conocidas. Las inversiones no equilibradas (con pérdida o adición de material cromosómico) casi siempre dan lugar a un fenotipo anormal.
Inversión paracéntrica - Paracentric inversion
Inversión en la que los dos puntos de rotura están situados en el mismo brazo de un cromosoma. El segmento invertido no incluye nunca el centrómero del cromosoma.
Inversión pericéntrica - Pericentric inversion
Inversión en la que los puntos de rotura se producen en los dos brazos del cromosoma. El segmento invertido incluye el centrómero del cromosoma.
Isocromosoma - Isochromosome
Cromosoma metacéntrico anormal que se produce durante la meiosis o mitosis, cuando la división del centrómero se produce según el plano horizontal en vez de vertical. Ambos brazos del isocromosoma son genéticamente idénticos pero en sentido inverso.
Isoformas - Isoforms
Productos proteicos distintos creados a partir del mismo gen. Por splicing alternativo la transcripción de un gen puede implicar a grupos diferentes de exones, resultando en distintas proteínas.
Isótopo - Isotope
Una de las varias formas de un átomo que tiene el mismo número atómico pero diferente masa atómica.
J
Judíos askenazi - Ashkenazi Jewish (sinónimo: judíos del este de Europa)
Población de judíos del este de Europa, originaria principalmente de Alemania, Polonia y Rusia, en comparación con los judíos sefardíes que proceden principalmente de España, determinadas zonas de Francia, Italia y norte de África.
K
Kilobase (kb) - Kilobase
Unidad de longitud de los ácidos nucleicos que corresponde a 1000 nucleótidos. En DNA bicatenario sería 1000 pares de bases (Kbp).
Knockout - Knockout
Organismo que ha sido genéticamente diseñado para carecer de uno o más genes específicos. Los knockouts se diseñan a menudo en ratones para que se pueda estudiar el impacto de los genes que faltan y comprender su función.
L
L1 -
Familia de elementos nucleares largos dispersos (LINE), presente en el genoma humano en número moderado de copias (alrededor de 80.000). Formada por unidades de unas 6 kb que pueden comportarse como retrotransposones.
Lastre genético - Genetic Load
Diferencia entre la aptitud de un genotipo promedio en una población y la aptitud del mejor genotipo presente en una población (genotipo de referencia u óptimo).
Leptoteno - Leptotene
Etapa de división celular durante la profase de la meiosis en la cual los cromosomas no son distintos sino que aparecen como una masa de hilos enredados.
Lesión - Lesion
Un área dañada en un gen (i.e., mutación), un cromosoma o una proteína.
Letal ligado al cromosoma X - X-linked lethal
Trastorno causado por una mutación dominante en un gen del cromosoma X que se observa casi exclusivamente en las mujeres, porque la mayoría de las veces es letal en los varones que heredan la mutación génica.
Leucina - Leucine
La leucina, abreviada leu o L, es uno de los aminoácidos esenciales. En el ARN mensajero está codificada como UUA, UUG, CUU, CUC, CUA o CUG.
Ley de Hardy-Weinberg - Hardy-Weinberg Law
También conocido como equilibrio de Hardy-Weinberg. Principio que establece que la variación genética en una población se mantendrá constante de una generación a la siguiente en ausencia de factores perturbadores. Cuando el apareamiento es aleatorio en una gran población sin circunstancias perturbadoras, la ley predice que tanto el genotipo como la frecuencia de los alelos se mantendrán constantes porque están en equilibrio.
Leyes de Mendel - Mendel´s Laws
Principios que describen la herencia cromosómica: principio de uniformidad (1ª Ley), principio de segregación (2ª Ley) y principio de distribución independiente (3ª Ley).
Librería - Librery
Colección desordenada de clones de ADN de un organismo particular cuya relación entre sí puede establecerse mediante un mapeo físico.
Ligado al sexo - Sex-linked
Fenotipo que se expresa solo en individuos de un determinado sexo.
Ligado al X - X-linked
Fenotipo determinado por un gen localizado en el cromosoma X.
Ligado al Y - Y-linked
Fenotipo determinado por un gen localizado en el cromosoma Y.
Ligamiento - Linkage
Véase desequilibrio de ligamiento.
Ligasa - Ligase
Enzima que puede unirse con un enlace roto fosfodiéster a un ácido nucleico.
Limitado por el sexo - Sex-limited
Fenotipos diferentes según el sexo a pesar de tener el mismo genotipo. Ello es debido a genes que están presentes en ambos sexos de especies que se reproducen sexualmente pero que se expresan en un solo sexo y permanecen "desactivados" en el otro.
Línea - Line
Grupo de organismos diploides o poliploides, de reproducción pura, idénticos, distinguidos de otros individuos de la misma especie por algún fenotipo y genotipo únicos.
Línea germinal - Germline
Línea celular de la que proceden o derivan directamente los óvulos o los espermatozoides (gametos).
Linfocito - Lymphocyte
Leucocito que se origina en la médula ósea y completa su diferenciación en los ganglios linfáticos. Tiene un solo núcleo y un protoplasma no granular.
Linfocito (leucocito del sistema inmunitario humano)
Lisina - Lysine
La lisina, abreviada lys o K, es un aminoácido esencial para los seres humanos. En el ARNm está codificada por los codones AAA y AAG.
Lisosoma - Lysosome
Orgánulo celular ligado a la membrana que contiene enzimas digestivas. Los lisosomas están involucrados en diversos procesos celulares. Descomponen partes de la célula sobrantes. Se pueden usar para destruir virus y bacterias invasores. Si la célula se daña irreparablemente, los lisosomas pueden ayudarla a autodestruirse en un proceso llamado muerte celular programada o apoptosis.
Locus - Locus (plural: loci)
Lugar o localización física de un gen específico en un cromosoma
LOD score -
Abreviatura del logaritmo de probabilidades. Medida obtenida al dividir la probabilidad de que dos loci estén vinculados en una fracción de recombinación específica por la probabilidad de que estén desvinculados.
Lugar de ensamblaje del donante - Donor splicing site
Límite entre el extremo 3' de un exón y el extremo 5' del intrón siguiente (en sentido 5' a 3').
Lugar de ensamblaje del receptor - Acceptor splicing site
Límite entre el extremo 3' de un intrón y el extremo 5' del exón siguiente (en sentido 5' a 3').
Lugar de ensamblaje oculto - Cryptic splicing site
Sitios desfavorables que están inactivos o se usan solo en niveles bajos a menos que se activen por mutación de sitios de empalme auténticos o ventajosos cercanos.
Lugar de poliadenilación - Polyadenylation site
Posición de un RNA mensajero en la que se añade una cola de poliadeninas, durante el proceso de maduración del mismo.
Lugar de restricción - Restriction Site
Ubicaciones en una molécula de ADN que contiene secuencias de nucleótidos específicas (4-8 pares de bases de longitud), que son reconocidas por enzimas de restricción.
M
Mapeo génico - Gene mapping
Proceso gracias al cual se determina las posiciones relativas de genes en una molécula de ADN, de un cromosoma o de un plásmido, y de la distancia, dada en unidades de ligamiento o física, que hay entre ellos.
Marcador - Marker
Segmento identificable de ADN (p. ej. RFLP, VNTR, microsatélite) que al presentar una variación amplia entre los individuos, permite analizar su herencia y segregación con los alelos de un gen determinado. Se utiliza en el análisis de ligamiento.
Marcador flanqueante - Flanking marker
Región polimórfica identificable de ADN (es decir, un marcador) localizada en un gen, a diferencia del marcador intragénico, que está localizado dentro de un mismo gen. Los marcadores flanqueantes se utilizan en el análisis de ligamiento para analizar la herencia del gen en cuestión.
Marcador intragénico - Intragenic marker
Región polimórfica identificable de ADN (es decir, un marcador) localizado dentro de la extensión de la secuencia de un gen (es decir, intragénico), a diferencia del marcador flanqueante, que está localizado en uno de los dos lados de un gen. Los marcadores intragénicos se utilizan en el análisis de ligamiento para investigar la herencia del gen en cuestión.
Marco de lectura - Reading frame (sinónimo: exón)
Secuencia de ARN mensajero que es traducido a una cadena de aminoácidos, tres bases al mismo tiempo (triplete) codifican un solo aminoácido.
Marco de lectura abierta - Open reading frame (sinónimo: ORF)
Todos los exones de un gen que contribuyen a los productos proteicos del gen.
Mecanismo de reparación de bases mal emparejadas - Mismatch repair mechanism (sinónimo: reparación de emparejamientos erróneos)
Sistema de control de la integridad del ADN, en el que intervienen determinados genes, que identifica, escinde y corrige los errores de emparejamiento de las bases durante la replicación del ADN. Las mutaciones producidas en los genes responsables de este mecanismo pueden dar lugar a determinadas enfermedades genéticas y algunos tipos de cáncer.
Medicina Personalizada de Precisión - Personalized Precision Medicine
La Medicina Personalizada de Precisión (MPP) se entiende como la identificación y aplicación del abordaje preventivo, diagnóstico y terapéutico más efectivos para cada paciente, utilizando como herramienta la Medicina de Precisión.
Megabase, Mb - Megabase
Unidad de longitud para fragmentos de ADN que equivale a 1 millón de nucleótidos (aproximadamente 1 centimorgan, cM).
Meiosis - Meiosis
Proceso que consiste en dos divisiones celulares consecutivas en los progenitores diploides de las células germinales. Es el proceso por el cual se originan los gametos, que poseen la mitad de cromosomas. Cada una de las dos divisiones de la meiosis tiene las mismas cuatro fases de una mitosis normal: profase, metafase, anafase y telofase.
Metacéntrico - Metacentric
Cromosoma que tiene el centrómero situado aproximadamente en el centro, de modo que los brazos son de longitud similar.
Metafase - Metafase
Fase de la mitosis o la meiosis durante la cual los cromosomas se colocan en el plano ecuatorial de la célula.
Metilación - Methylation
Unión de los grupos metilo a las citosinas del ADN. Se relaciona con una transcripción reducida del gen y se considera el mecanismo principal en la inactivación del cromosoma X y en la impronta genómica.
Metionina - Methionine
La metionina (abreviada como Met o M) es un aminoácido hidrófobo, y esencial. Es uno de los dos aminoácidos codificados por un único codón del código genético, el AUG (el otro es el triptófano, que está codificado por el codón UGG), que es también el mensaje que indica al ribosoma el inicio de la traducción de una proteína desde el ARNm.
Micro RNA (miRNA) - Micro RNA (miRNA)
Los microRNAs (miRNAs) son pequeños RNAs no codificantes. Una de sus funciones conocidas es la regulación postranscripcional de la expresión de genes, mediante la unión a secuencias diana en las regiones 3’UTR (no traducidas) de los ARN mensajeros.
Microarray - Microarray
Superficie sólida sobre la que son depositados y fijados secuencias de genes o fragmentos de genes siguiendo un orden espacial determinado. Se utilizan para estudios de alta densidad, es decir, para analizar muchos genes/miRNAs/SNPs, etc al mismo tiempo en el tejido de interés.
Microdeleción - Microdeletion
Deleción que no puede ser detectada por técnicas citogenéticas clásicas.
Microsatélite - Microsatellite (sinónimos: ADN satélite, repeticiones cortas en tándem)
Segmentos repetitivos de ADN que comprenden de dos a cinco nucleótidos (repeticiones de dinucleótidos/trinucleótidos/ tetranucleótidos/ pentanucleótidos) dispersos por todo el genoma en las regiones no codificadoras que hay entre o dentro de los genes (intrones), se utilizan frecuentemente como marcadores en el análisis de ligamiento debido a la alta variabilidad natural existente en el número de repeticiones entre los individuos. Estas regiones son propiamente inestables y susceptibles a mutaciones.
Mitosis - Mitosis
Proceso por el cual una célula se divide y da lugar a dos células hijas.
Modificación del riesgo - Risk modification
Variación del riesgo genético de un individuo basándose en la información previamente desconocida sobre los antecedentes familiares o en los resultados de los estudios genéticos; dependiendo de la información nueva, el riesgo puede aumentar o disminuir.
Modo de herencia - Mode of inheritance (sinónimos: patrón de herencia)
Manera en la que un determinado rasgo o trastorno genético se transmite de una generación a la siguiente. Ejemplos son el modo de herencia autosómico, dominante y recesivo, el modelo ligado al cromosoma X, dominante y recesivo, y la herencia mitocondrial.
Monosomía - Monosomy
Presencia de sólo uno de la pareja de cromosomas homólogos. La monosomía parcial se refiere a la presencia de una sola copia de un segmento de un cromosoma.
Mosaicismo - Mosaicism
Mosaicismo de línea germinal - Germline mosaicism
Presencia simultánea de dos o más líneas celulares genética o citogenética confinadas a la línea germinal (óvulos o espermatozoides); anteriormente se denominaba mosaicismo gonadal.
Mosaicismo gonadal - Gonadal mosaicism
Véase Mosaicismo de línea germinal.
Mosaicismo somático - Somatic mosaicism
Dos o más líneas celulares que varían en su constitución genética o citogenética en las células del organismo (puede incluir o no las células germinales).
Mutación - Mutation (sinónimos: alteración de la secuencia, mutación del splicing)
Cualquier alteración producida en un gen con respecto a su estado natural; puede ser patológica o una variante no patológica.
1. Mutaciones patológicas: aquellas que dan lugar a una función anormal del gen y a un fenotipo de enfermedad. Los tipos de mutaciones patológicas son los siguientes:


Adaptado de Thompson & Thompson Genetics in Medicine, 6th Edition; RL Nussbaum, RR McInnes, HF Willard, Genetic Variation in Individuals: Mutation and Polymorphism,pág 81, Copyright 2001, con permiso de Elsevier
2. Polimorfismos (variantes normales): Mutaciones que no tienen efectos adversos sobre la función del gen y no dan lugar a un fenotipo alterado (enfermedad).
Mutación de línea germinal - Germline mutation
Presencia de un gen alterado en el óvulo o espermatozoide (célula germinal) que puede transmitirse a las generaciones siguientes.
En un individuo, la mutación puede darse en una de cuatro situaciones posibles:
1.- Exclusivamente en una sola célula germinal (óvulo o espermatozoide).
2.- Exclusivamente en las células somáticas (células del organismo, pero NO en la línea germinal); son las normalmente asociadas al desarollo de un tumor.
3.- En algunas células germinales y en otras somáticas.
4.- En todas las células del organismo.


Mutación de novo - De novo mutation (sinónimos: mutación génica de novo, mutación génica nueva, mutación nueva)
Mutación de sentido erróneo - Missense mutation
Sustitución de un solo par de bases que da lugar a la traducción de un aminoácido diferente en esa posición.
Mutación dominante negativa - Dominant negative mutation
Mutación cuyo producto génico afecta de forma adversa al producto génico normal de tipo salvaje de la misma célula, normalmente al dimerizarse (combinarse) con él. En las moléculas poliméricas, tales como el colágeno, las mutaciones dominantes negativas suelen ser más deletéreas que las que no originan ningún producto génico (mutaciones nulas o alelos nulos).
Mutación en el sitio de corte y empalme (splicing) - Splicing
Mutación que altera o suprime la secuencia específica que indica el sitio en el que tiene lugar el splicing de un intrón. A consecuencia de estas mutaciones, uno o más intrones permanecen en el ARN mensajero maduro y pueden alterar la generación del producto proteico.
Mutación familiar específica - Familial mutation
En una familia, la alteración observada en una secuencia que causa una determinada enfermedad o predispone a ella; la mutación puede ser rara o común.
Mutación intrónica - Intronic mutation
Mutación (normalmente una sustitución de bases) localizada en un intrón que genera un sitio de corte y empalme (splicing) alternativo que compite con los sitios de splicing normales durante el procesamiento del ARN. Esta mutación da lugar a la formación de ARN mensajero maduro con secuencias intrónicas.
Mutación no descrita - Novel mutation
Alteración nueva de un gen, que no ha sido previamente descrita. No debe confundirse con mutación “nueva” o “de novo”.
Mutación particular - Private mutation (sinónimo: mutación única)
Tipo de mutación patológica rara que se observa sólo en unas pocas familias.
Mutación patológica - Disease
Alteración en un gen que causa o predispone a un individuo a desarrollar una enfermedad específica.
Mutación por cambio de marco - Frameshift mutation (sinónimos: deleción fuera de marco, mutación fuera de marco)
Inserción o deleción de una secuencia de pares de bases que no es múltiplo de tres y por lo tanto, altera el marco de lectura del triplete, lo que da lugar normalmente a la creación de un codón de terminación (parada) prematuro y cuyo resultado es un producto proteico truncado.
Mutación predisponente - Predisposing mutation (sinónimo: gen de susceptibilidad)
Mutación génica que incrementa la susceptibilidad o predisposición de un individuo a desarrollar una determinada enfermedad. Cuando se heredan mutaciones patogénicas en estos genes de susceptibilidad, es probable, pero no seguro, que el individuo desarrolle síntomas de la enfermedad.
Mutación puntual - Point mutation
Alteración en la secuencia del ADN causada por el cambio, inserción o deleción de un único nucleótido.
Mutación sin cambio de pauta de lectura - In-frame mutation
Mutación que no produce un cambio en el marco de lectura del triplete cuando se produce la transcripción de ADN a ARN mensajero. Estas mutaciones, sin embargo, pueden dar lugar a la síntesis de un producto proteico anormal si, al traducirse el ARN a proteína, el cambio de triplete supone un cambio de aminoácido.
Mutación sin sentido - Nonsense mutation
Mutación que altera un codón convirtiéndolo en uno de terminación (stop codon) prematura del proceso de traducción.
Mutagénico - Mutagenic
Capaz de inducir cambios en la información genética de una célula.
N
Nivel de bandas - Band level
Número total de bandas estimadas presentes en los cromosomas del cariotipo, tratados con una técnica específica de bandeo (G,R, etc...). Cuando se detiene el proceso de división celular y se realiza la tinción en una fase más prematura de la mitosis (prometafase), los cromosomas son más largos y aparecen aproximadamente 700-1200 bandas. En una fase posterior de la mitosis (metafase), los cromosomas están más condensados y aparecen aproximadamente 300-600 bandas. Cuanto mayor es el nivel de bandas, mejor es la resolución, lo que permite identificar anomalías cromosómicas más sutiles, así como sus puntos de rotura.
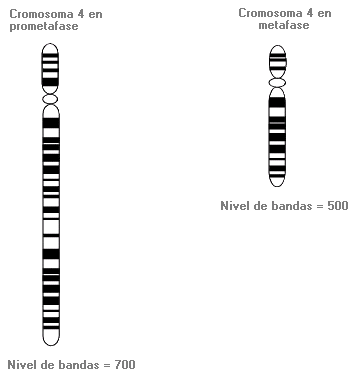
No afectado - Unaffected
Individuo que no manifiesta síntomas de un trastorno determinado.
Nombre del locus - Locus name
Abreviatura asignada de manera informal que se utiliza en el proceso de mapeado para denominar un supuesto gen antes de que sea identificado. Una vez que el gen es identificado, el nombre del locus se sustituye generalmente por el símbolo genético asignado formalmente (que suele diferir del nombre del locus).
Northern blot - Northern blot (sinónimo: análisis northern blotting)
Separación de secuencias o fragmentos de ARN, digeridos parcialmente por endonucleaseas, en un gel electroforético.
Núcleo - Nucleus
Organelo celular en eucariotes que contiene el material genético.
Nucleótido - Nucleotide
Molécula compuesta por una base nitrogenada (adenina, guanina, timina o citosina en ADN; adenina, guanina, uracilo o citosina en ARN), un grupo fosfato y un azúcar (desoxirribosa en ADN; ribosa en ARN). El ADN y el ARN son polímeros de muchos nucleótidos.
Ñ
Nivel de bandas - Band level
Número total de bandas estimadas presentes en los cromosomas del cariotipo, tratados con una técnica específica de bandeo (G,R, etc...). Cuando se detiene el proceso de división celular y se realiza la tinción en una fase más prematura de la mitosis (prometafase), los cromosomas son más largos y aparecen aproximadamente 700-1200 bandas. En una fase posterior de la mitosis (metafase), los cromosomas están más condensados y aparecen aproximadamente 300-600 bandas. Cuanto mayor es el nivel de bandas, mejor es la resolución, lo que permite identificar anomalías cromosómicas más sutiles, así como sus puntos de rotura.

No afectado - Unaffected
Individuo que no manifiesta síntomas de un trastorno determinado.
Nombre del locus - Locus name
Abreviatura asignada de manera informal que se utiliza en el proceso de mapeado para denominar un supuesto gen antes de que sea identificado. Una vez que el gen es identificado, el nombre del locus se sustituye generalmente por el símbolo genético asignado formalmente (que suele diferir del nombre del locus).
Northern blot - Northern blot (sinónimo: análisis northern blotting)
Separación de secuencias o fragmentos de ARN, digeridos parcialmente por endonucleaseas, en un gel electroforético.
Núcleo - Nucleus
Organelo celular en eucariotes que contiene el material genético.
Nucleótido - Nucleotide
Molécula compuesta por una base nitrogenada (adenina, guanina, timina o citosina en ADN; adenina, guanina, uracilo o citosina en ARN), un grupo fosfato y un azúcar (desoxirribosa en ADN; ribosa en ARN). El ADN y el ARN son polímeros de muchos nucleótidos.
O
Oligonucleótidos específicos del alelo - Allele-specific oligonucleotide testing (ASO)
Prueba de genética molecular empleada para la detección de una mutación específica por medio de un segmento sintético de ADN de una longitud aproximada de 20 pares de bases (oligonucleótido). El investigador diseña la secuencia específica que se une a la secuencia complementaria en una muestra de ADN problema, lo que permite su identificación.
Oncogen - Oncogene
Gen activado por una mutación y que potencialmente induce el desarrollo de cáncer. Muchos de los oncogenes están implicados, directa o indirectamente, en el control del ciclo celular.
P
Panel de genes - Multigene panel
Prueba genética en la que se analiza simultáneamente el estado de un grupo de genes asociados con una determinada patología. Son muy usados los que analizan el conjunto de genes asociados con la susceptibilidad a desarrollar cáncer colorrectal, de mama, etc., o los genes relacionados con la discapacidad intelectual. Los avances en las técnicas de secuenciación permiten analizar en un único test un número prácticamente ilimitado de genes. Frente al habitual análisis de un único gen, el uso de estos paneles se ha extendido en los años más recientes tanto en el terreno de la investigación, como en la práctica clínica ya que permiten una valoración potencialmente más completa de los riesgos derivados de la alteración de genes de susceptibilidad.
Par de bases - Base pair: (sinónimo: bp)
Dos bases nitrogenadas unidas mediante enlaces débiles en el ADN de cadena doble; el emparejamiento específico de estas bases (adenina con timina y guanina con citosina) facilita la replicación exacta del ADN; la cuantificación de los pares de bases (p. ej. 8 bp) se refiere a la longitud física de una secuencia de nucleótidos.
ADN de cadena doble que muestra el emparejamiento específico de los pares de bases G y C, A y T .
Parentesco - Kindred
Relación entre los miembros de una familia; término que se utiliza frecuentemente en los estudios de ligamiento para referirse a las familias numerosas.
Pariente de primer grado - First-degree relative
Cualquier individuo que esté separado por una meiosis de uno de los miembros de su familia (es decir, padre/madre, hermano/a, hijo/a).
Los parientes de primer grado de la persona señalada con la flecha son los padres, hermanos e hijos:
Algunas implicaciones clínicas:
Un individuo comparte la mitad de sus genes con cada uno de sus padres y cada uno de sus hijos.
Un individuo comparte, por término medio, la mitad de sus genes con cada uno de sus hermanos.
Pariente de segundo grado - Second-degree relative
Cualquier individuo que esté separado por dos meiosis de uno de los miembros de su familia; familiar con el que un individuo comparte la cuarta parte de sus genes (es decir, abuelos, nietos, tío, tía, sobrino, sobrina, hermanastros).
Paternidad alternativa - Alternate paternity (sinónimos: falsa paternidad, no paternidad):
Situación en la que el supuesto padre de un determinado individuo no es el padre biológico.
Patrón de herencia mendeliano - Mendelian inheritance pattern
Característicos de organismos que se reproducen sexualmente. Gregor Mendel describió dos tipos de “factores” (genes) conforme a su expresión sobre el fenotipo: los dominantes y los recesivos. Si además tenemos en cuenta que los individuos de sexo femenino tienen dos cromosomas X, y los del sexo masculino una X y una Y, los modos de transmisión de los caracteres de una mutación, son un total de cuatro.
PCR - PCR (Polymerase chain reaction) (sinónimo: reacción en cadena de la polimerasa)
Procedimiento que genera millones de copias de un segmento corto de ADN mediante ciclos repetidos de: 1) desnaturalización del ADN, 2) acoplamiento de los oligonucleótidos y 3) extensión mediante la acción de la ADN polimerasa. La PCR es un procedimiento muy común en los estudios de genética molecular y se puede utilizar para: 1) generar una cantidad suficiente de ADN para realizar determinadas pruebas (p. ej. análisis de la secuencia, rastreo de mutaciones); o 2) puede ser una prueba diagnóstica en sí misma (por ejemplo amplificación específica del alelo, cuantificación del número de repeticiones de trinucleótidos).
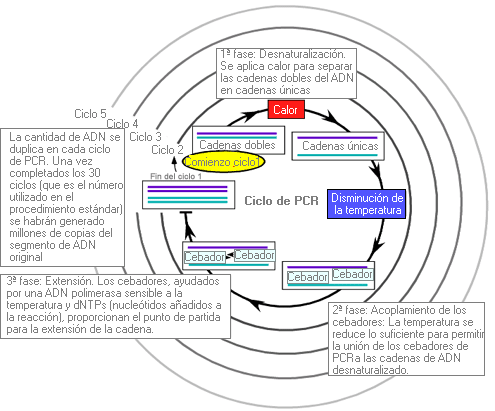
PCR cuantitativa - Quantitative PCR (sinónimos: PCR cuantitativa en tiempo real)
Uso de PCR para determinar la cantidad de ADN o ARN en una muestra; se utiliza frecuentemente para detectar mutaciones por deleción y mutaciones por duplicación.
Anteriormente, la PCR cuantitativa determinaba la cantidad de producto (ADN o ARN) tras completar un número específico de ciclos; sin embargo, este procedimiento no es del todo fiable o preciso y se ha sustituido en gran parte por la PCR cuantitativa “en tiempo real” (denominada también “cinética”), en la que se determina la cantidad del producto (ADN o ARN) tras completar cada ciclo de PCR.
PCR cuantitativa en tiempo real
I. Se amplifica la región de ADN o ARN de interés en presencia de una sonda fluorescente; la intensidad de la señal fluorescente generada aumenta en proporción a la cantidad de producto resultante en PCR.
II. La intensidad de la señal fluorescente, que se mide al completar cada ciclo de PCR, se representa en una “gráfica de amplificación” para determinar el CT (del inglés cycle threshold), que se define como el punto en el que se detecta por primera vez la señal fluorescente, es decir, el producto de la PCR.
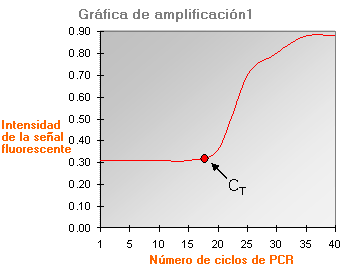
III. Se compara el valor CT frente a una curva estándar (basándose en los valores CT de muestras control y sus respectivas cantidades iniciales de ADN o ARN conocidas) para determinar la cantidad de ADN o ARN en la muestra del paciente. La presencia de una mutación por deleción o duplicación se puede deducir comparando la cantidad de ADN o ARN en la muestra del paciente con la cantidad en una muestra control. Por ejemplo, si la cantidad de ADN o ARN en la muestra del paciente fuese ~50% respecto a la del control normal, esto indicaría una mutación por deleción; si la cantidad de ADN o ARN en la muestra fuese ~150% respecto a la del control normal, indicaría una mutación por duplicación.
1Adaptado de PennState College of Medicine, Quantitative Real-Time PCR
Algunas implicaciones clínicas
Al igual que la PCR, la PCR cuantitativa se ha convertido en una técnica estándar en todos los laboratorios clínicos de genética molecular.
Los análisis para detectar mutaciones por deleción hemicigótica (p. ej. deleciones en DMD en varones afectados por distrofia muscular de Duchenne) se pueden realizar con técnicas de PCR estándar. Sin embargo, se debe realizar PCR cuantitativa, en lugar de estándar, para detectar mutaciones por deleción heterocigótica (p. ej. deleciones heterocigóticas en (1) individuos afectados por un trastorno causado por una deleción en un gen contiguo autosómico, como, por ejemplo, el síndrome de Williams, (2) portadores de un trastorno autosómico recesivo, como la atrofia muscular espinal y (3) mujeres portadoras de un trastorno ligado al cromosoma X, por ejemplo la distrofia muscular de Duchenne).
Pedigrí - Pedigree
Véase Árbol genealógico.
Penetrancia - Penetrance
Proporción de individuos que presentan una mutación causante de una patología determinada y muestran síntomas clínicos de esa patología. La mayoría de las veces este término se refiere a las patologías con herencia autosómica dominante.

¿Cuál es la diferencia entre penetrancia y expresividad variable?
La penetrancia es la proporción de individuos con una mutación patológica que presentan manifestaciones clínicas de la patología asociada a esa mutación. La expresividad variable se refiere al rango o espectro de manifestaciones clínicas observadas en individuos que presentan una patología determinada. El término expresividad variable se aplica a enfermedades con cualquier patrón de herencia.
¿De qué forma influye la expresividad variable en la penetrancia?
La penetrancia depende de las manifestaciones de enfermedad que se utilizan para determinar si un individuo está afectado. Por ejemplo, se puede suponer que los individuos con mínimas manifestaciones de una patología autosómica dominante no están afectados, por lo que la penetrancia reducida en este caso sería tan solo aparente. Consideremos lo siguiente:
Se sabe que una paciente presenta una mutación en FBN1, causante del síndrome de Marfan, porque tanto su padre como su hijo están afectados. Los resultados de la exploración física de esta paciente son normales, por lo que se deduce que la paciente no presenta manifestaciones del síndrome de Marfan, esto es una falta de penetrancia. Sin embargo, el ecocardiograma revela dilatación aórtica, que es una característica del síndrome de Marfan. La paciente realmente tiene manifestaciones del síndrome. Los resultados basados en la exploración física y en el ecocardiograma revelarán una penetrancia más alta del síndrome de Marfan que los estudios basados sólo en la exploración física.
Péptido - Peptide
Molécula formada por aminoácidos unidos por enlaces peptídicos (enlaces covalentes entre el grupo amino [–NH2] de un aminoácido y el grupo carboxilo [–COOH] de otro aminoácido). Una serie de aminoácidos unidos también se denomina polipéptido. Las proteínas están formadas por uno o varios polipéptidos.
Pérdida de heterocigosidad - Loss of heterozygosity (LOH, de sus siglas en inglés)
Cuando en un locus determinado, que es heterocigoto para un alelo mutante deletéreo y un alelo normal, se produce una deleción, u otro evento mutacional, en el alelo normal que convierte a la célula bien en hemicigótica (es decir, con un único alelo deletéreo), bien en homocigótica para el alelo deletéreo.
La LOH es el mecanismo en el que se basa el desarrollo de algunos tipos de cáncer en los que una sola mutación en uno de los homólogos de un gen supresor tumoral no es suficiente para iniciar el crecimiento del tumor. Sin embargo, la deleción o inactivación del segundo alelo en el cromosoma homólogo, da lugar a un crecimiento celular incontrolado.
La LOH puede ser el fundamento del desarrollo del cáncer en tumores esporádicos y hereditarios. En los tumores esporádicos, los dos alelos son normales en el momento de la concepción, mientras que en los tumores hereditarios hay un alelo mutante en el momento de la concepción.
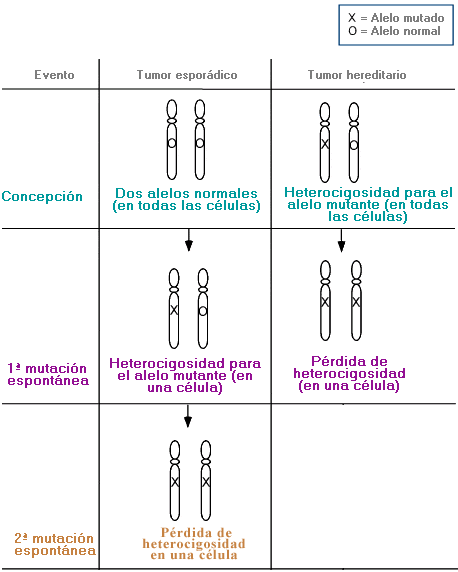
PGD - Preimplantation Genetic Diagnosis
Véase Diagnóstico preimplantacional.
Pirimidina - Pyrimidine
Es un compuesto orgánico, similar al benceno, y a la piridina pero con dos átomos de nitrógeno que sustituyen al carbono en las posiciones 1 y 3. La pirimidina tiene tres derivados que forman parte de los ácidos nucleicos: la timina, la citosina y el uracilo; las tres tienen un grupo carbonilo (C=O) en el carbono 2; las dos primeras forman parte del ADN donde se aparean con sus purinas complementarias, mientras que la última está presente solo en el ARN. En el ADN, adenina (A) y guanina (G) se emparejan con timina (T) y citosina (C), respectivamente, y en el ARN adenina se une a uracilo (U) y la guanina a citosina.
Pirrolisina - Pyrrolysine
La pirrolisina (abreviaturas Pyl o O) es un aminoácido natural, derivado de la lisina, codificado en el genoma de algunas arqueas metanógenas y una especie de bacteria (Desulfitobacterium hafniense). Forma parte de un código genético inusual, propio de estos organismos, y se considera el aminoácido proteinogénico número 22. Se encuentra codificada en el ARNm por el codón UAG.
Plásmido - Plasmid
Molécula pequeña, a menudo circular, de ADN extracromosómico, cuya replicación es independiente de la del ADN cromosómico. Son comunes en bacterias y levaduras. Por lo general contienen un reducido número de genes que suele conferir ciertas ventajas al hospedador, por ejemplo, resistencia a un determinado antibiótico.
Los plásmidos se usan en ingeniería genética aprovechando su capacidad para reproducirse de manera independiente del ADN cromosómico.
Pleiotropia - Pleiotropy
Múltiples efectos fenotípicos o caracteres distintos causados por la acción de un solo gen o par de genes. El término se utiliza de manera particular cuando no se sospecha una relación entre los rasgos o caracteres fenotípicos.
Ploidía - Ploidy
Número de grupos o juegos completos de cromosomas que contiene una determinada célula. Las células somáticas humanas son diploides, dos grupos completos de cromosomas, cada uno, con 23 cromosomas, procedente de un progenitor. En cambio, los gametos, óvulo y espermatozoide, son haploides, cada uno con 23 cromosomas.
Poligénico - Polygenic
Rasgo o patología causado por las contribuciones aditivas de mutaciones producidas en múltiples genes en diferentes loci.
Polimorfismo - Polymorphism
Polimorfismo de Conformación de Cadena Simple o SSCP -
Polimorfismo de longitud de fragmentos de restricción - Restriction fragment length polymorphism (sinónimo: RFPL)
Variación natural (polimórfica) de una secuencia de ADN entre los individuos. Se visualiza porque se suprimen o se crean nuevos sitios de restricción (corte) en el ADN, que cuando es digerido por una endonucleasa da lugar a fragmentos de ADN de diferente longitud.
Polimorfismo de nucleótido único - Single Nucleotide Polymorphism o SNP
Variación en la secuencia de ADN que implica una sola base: adenina (A), timina (T), citosina (C) o guanina (G). Son los polimorfismos genéticos más frecuentes y se considera que determinan gran parte de la variabilidad genética entre individuos. Se conocen varios millones de SNP repartidos por todos los cromosomas humanos, aproximadamente 1 cada 300-100 pares de bases. La frecuencia de un SNP en población general se sitúa por encima del 1%.
Diversas técnicas de análisis masivo del genoma se fundamentan en el estudio de miles o millones de SNP que cubren la práctica totalidad del genoma (Genome Wide Association Studies, o GWAS), para buscar la asociación de algunos de ellos con enfermedades concretas, utilizando para ello grandes números tanto de individuos enfermos, como de individuos sanos.
Polimorfismos de conformación de cadenas sencillas - Single-stranded conformational polymorphism (SSCP)
Técnica para el rastreo de mutaciones que identifica segmentos de ADN de cadena sencilla que muestran un patrón de migración anormal en la electroforesis en gel.
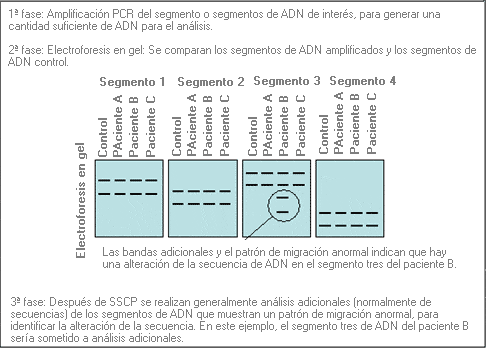
Algunas implicaciones clínicas
La técnica SSCP se puede utilizar cuando las mutaciones están distribuidas por todo el gen.
El SSCP puede comprender el análisis de todo el gen o sólo de regiones específicas.
Poliploidía - Polyploidy
Incremento del número de cromosomas de una célula que es múltiplo del número haploide (23). La triploidía se refiere a tres grupos o juegos completos de cromosomas en una sola célula (en los seres humanos, la triplodía se corresponde con la presencia de un total de 69, 3x23, cromosomas en cada célula); la tetraploidía se refiere a cuatro grupos de cromosomas en una sola célula (en los seres humanos serían 92, 4x23, cromosomas por célula).
Portador - Carrier
Portador obligado - Obligate carrier (sinónimo: heterocigoto obligado)
Portador sintomático - Manifesting carrier
Individuo que en un locus determinado presenta un alelo recesivo patológico en un cromosoma y un alelo normal en el otro cromosoma y que manifiesta algunos síntomas de la enfermedad. Este término se usa con frecuencia para portadoras de una mutación recesiva ligada al cromosoma X que manifiestan signos de la enfermedad, aunque su fenotipo suele ser menos severo que el de los varones con la misma mutación.
Aunque con algunas excepciones, los portadores de una mutación patológica recesiva por regla general no manifiestan síntomas clínicos de la enfermedad. Sin embargo, los portadores de mutaciones recesivas ligadas al cromosoma X presentan ocasionalmente algunas manifestaciones clínicas de la enfermedad, por lo que se les denomina portadores sintomáticos. Se supone que el mecanismo subyacente en los portadores sintomáticos es una inactivación sesgada o no aleatoria del cromosoma X, de tal manera que en la mayoría de las células el cromosoma X activo es el que tiene la mutación.
Ejemplos de enfermedades ligadas al cromosoma X en las que son frecuentes las portadoras sintomáticas:
Hemofilia A: Tiempo de coagulación prolongado, hemorragia excesiva durante intervenciones quirúrgicas menores y menstruaciones abundantes.
Distrofia muscular de Duchenne: Debilidad muscular, hipertrofia de pantorrillas leve y/o miocardiopatía dilatada.
Síndrome X frágil (mutación con penetrancia completa): Retraso mental.
Potenciador - Enhancer
Pequeña región del ADN eucariota que puede aumentar los niveles de transcripción de un gen o un grupo de genes mediante la unión a proteínas (factores de transcripción). Las regiones enhancer no tienen por qué estar localizadas cerca de los genes sobre los que actúa, ni siquiera en el mismo cromosoma.
Predisposición genética - Genetic predisposition (sinónimo: susceptibilidad genética)
Mayor propensión de un individuo a padecer una enfermedad determinada debido a la presencia de una o más mutaciones génicas asociadas con un aumento del riesgo de que se manifieste la enfermedad y/o debido a la existencia de antecedentes familiares que también indican una mayor probabilidad de que se desarrolle la enfermedad.
Prevalencia - Prevalence
Frecuencia de una enfermedad en la población, contabilizando tanto los casos antiguos como los de nueva aparición.
Primer - Primer
Probando - Proband (sinónimos: caso índice, propósito)
Producto génico - Gene product
Profase - Prophase
Es la primera fase de la mitosis y de la meiosis. En ella se produce la condensación de todo el material genético (ADN)-que normalmente existe en forma de cromatina condensada dentro de una estructura altamente ordenada llamada cromosoma- y el desarrollo bipolar del huso acromático. Los dos pares de centríolos migran hacia extremos opuestos de la célula, la membrana nuclear se disuelve al final de la profase y los cromosomas se anclan en los microtúbulos del huso gracias al cinetocoro que se encuentra en el centrómero.
Prolina - Proline
La prolina (Pro, P) o prolalina es uno de los aminoácidos que forman las proteínas de los seres vivos. En el ARN mensajero está codificada como CCU, CCC, CCA o CCG. Se puede formar directamente a partir de la cadena pentacarbonada del ácido glutámico, y por tanto no es un aminoácido esencial.
Prometafase - Prometaphase
Es el proceso o fase posterior a la profase y anterior a la metafase en la mitosis celular. Según cómo interactúen entre sí durante esta fase la membrana nuclear y el huso mitótico de microtúbulos, la mitosis puede ser abierta (la membrana nuclear se separa, y los microtúbulos atraviesan el espacio nuclear. Ocurre en una pequeña parte de los organismos multicelulares) o cerrada (el huso mitótico se forma dentro del núcleo o sus microtúbulos pueden penetrar hasta los cromosomas a través de la membrana nuclear sin que esta se rompa). Ocurre en los hongos y algunos protistas.
Promotor - Promoter
Secuencia de ADN necesaria para activar un gen. La transcripción del gen se inicia en el promotor. Generalmente se encuentran cerca del comienzo de un gen, el promotor tiene un sitio de unión para la enzima ARN polimerasa encargada de la síntesis del ARNm.
Proteómica - Proteomics
Estudio a escala masiva de la distribución y número de proteínas codificadas por los genes, y sus interacciones.
Protooncogen - Protooncogen
Gen normal generalmente implicado en funciones celulares básicas de crecimiento y diferenciación celular, que cuando muta se convierte en oncogen, es decir promotor del proceso tumoral.
Proyecto Genoma Humano - Human Genome Project
El Proyecto Genoma Humano fue una investigación internacional que descifró la totalidad de la secuencia del genoma humano, mapeando y secuenciando todos sus genes. Oficialmente se inició en 1990 como un programa a quince años vista, con el que se pretendía registrar los 80.000-100.000 genes humanos (finalmente se comprobó que eran alrededor de 20.000-25.000). Los rápidos avances tecnológicos aceleraron los tiempos de modo que los resultados iniciales se publicaron en febrero de 2001, y la secuenciación se completó en el 2003, aunque no se conocen en detalle las funciones de todos los elementos del genoma.
Los objetivos concretos del Proyecto fueron:
• Identificar los aproximadamente 100.000 genes humanos en el ADN.
• Determinar la secuencia de 3 billones de bases químicas que conforman el ADN.
• Guardar la información en bases de datos.
• Desarrollar de modo rápido y eficiente tecnologías de secuenciación.
• Desarrollar herramientas para análisis de datos.
• Dirimir las cuestiones éticas, legales y sociales que se derivan del proyecto.
Prueba de parentesco - Parentage testing (sinónimos: prueba de maternidad, prueba de paternidad)
Proceso mediante el cual se comparan secuencias de ADN de un niño y un adulto determinados para calcular la probabilidad de que los dos individuos estén emparentados. Mediante los análisis de ADN se puede descartar con seguridad, pero no confirmar absolutamente que un individuo es el padre o la madre biológicos.
Pseudoautosómico, región pseudoautosómica - Pseudoautosomal, Pseudoautosomal región
Regiones distales de los brazos cortos de los cromosomas sexuales X e Y que recombinan entre sí durante la meiosis del varón.
Pseudogen - Pseudogene
Copia de un gen que carece normalmente de intrones y de otras secuencias de ADN esenciales para su función. Los pseudogenes, aunque son genéticamente similares al gen funcional original, no se expresan y frecuentemente contienen numerosas mutaciones.
Pseudogenes:
Consisten en secuencias de ADN muy similares a los genes conocidos, pero no son funcionales.
Se encuentran dispersos en el genoma.
Se cree que son subproductos de la evolución, que representan genes rudimentarios que una vez fueron funcionales.
Se supone que han perdido su función original por la introducción de mutaciones en las secuencias codificadoras o reguladoras.
 Supuestos mecanismos por los que se forman los pseudogenes:
Supuestos mecanismos por los que se forman los pseudogenes:
Duplicación de un gen funcional seguida de la introducción de numerosas mutaciones que finalmente dan lugar a su inactivación.
Retrotransposición – proceso mediante el cual se generan una o más copias de ADN a partir del transcripto de ARNm del gen y se vuelven a insertar en el genoma en cualquier localización, no necesariamente cerca del sitio de su gen progenitor. Puesto que se generan a partir de ARNm procesado, estos pseudogenes carecen de intrones.
Puntos calientes de mutación - Hotspot mutation region
Secuencias de adn muy susceptibles a mutaciones debido a una inestabilidad inherente, tendencia hacia un entrecruzamiento desigual o predisposición química a sustituciones de nucleótidos simples; región en la que se observan mutaciones con más frecuencia de la habitual.
Purina - Purine
Es una base nitrogenada, un compuesto orgánico heterocíclico aromático. La estructura de la purina está compuesta por dos anillos fusionados, uno de seis átomos y el otro de cinco. Dos de las bases de los ácidos nucleicos, adenina y guanina, son derivados de una purina. En el ADN, estas bases se unen con sus pirimidinas complementarias, la timina y la citosina, a través de enlaces de hidrógeno.
Q
R
Rasgo - Trait
Reacción en cadena de la polimerasa - Polymerase chain reaction (PCR)
Véase PCR.
Recesivo - Recessive
Véase Autosómico recesivo.
Recesivo ligado al cromosoma X - X-linked recessive
Modo de herencia en el que una mutación en un gen del cromosoma X hace que el fenotipo esté expresado en varones que son hemicigóticos para la mutación del gen (es decir, sólo tienen un cromosoma X) y en mujeres que son homocigóticas para la mutación (es decir, presentan una copia de la mutación del gen en cada uno de los dos cromosomas X). Los Portadores femeninos que tienen una sola copia de la mutación no suelen expresar el fenotipo, aunque las diferencias en la inactivación del cromosoma X pueden dar lugar a distintos grados de expresión clínica en estos portadores.
Árbol genealógico que representa el patrón de herencia recesiva ligada al cromosoma X.
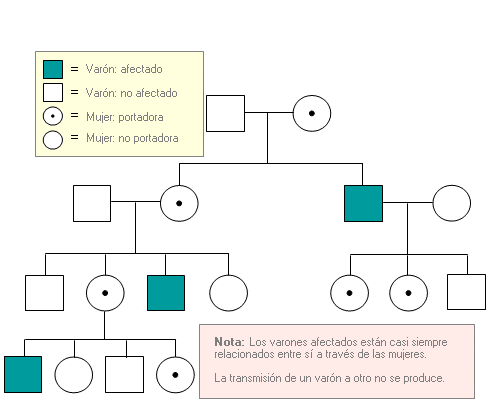
Recombinación - Recombination (sinónimo: sobrecruzamiento)
Intercambio de un segmento de ADN entre dos cromosomas homólogos durante la meiosis (aunque en algunas ocasiones se puede dar en mitosis), cuyo resultado es una combinación nueva de material genético en el gameto.
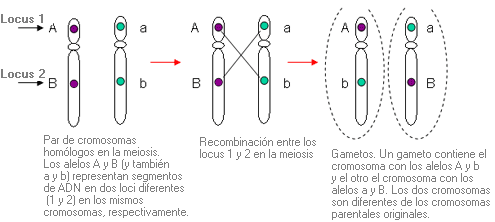 Dos genes que están localizados en el mismo cromosoma pueden segregarse juntos o no. La recombinación entre los cromosomas homólogos durante la meiosis da lugar a combinaciones nuevas de genes en el gameto, y por lo tanto, a descendientes.
Dos genes que están localizados en el mismo cromosoma pueden segregarse juntos o no. La recombinación entre los cromosomas homólogos durante la meiosis da lugar a combinaciones nuevas de genes en el gameto, y por lo tanto, a descendientes.
A través del proceso de recombinación, cada cromosoma transmitido de un padre a su hijo contiene algunos segmentos heredados del abuelo y otros de la abuela de ese niño.
Fuente: Elsevier.
La recombinación desempeña un papel importante para asegurar la variabilidad genética entre los individuos.
El comportamiento durante la meiosis de los alelos en dos loci (1 y 2) del mismo cromosoma depende de su proximidad.
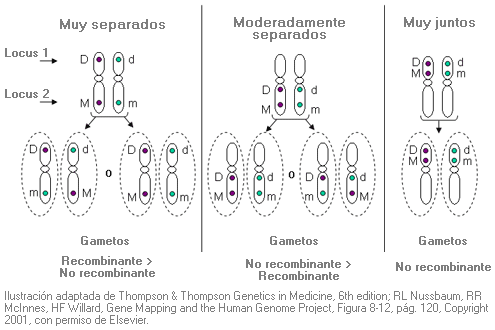
Región codificante - Coding region (sinónimo: marco de lectura abierta)
Todos los exones de un gen que contribuyen a su producto o productos proteicos.

Figura 1. El ADN transcrito inicialmente a ARN mensajero inmaduro (ARNm) consta de secuencias codificantes (exones) y no codificantes (intrones).

Figura 2. Los intrones se han escindido del ARNm inmaduro para formar ARNm maduro, con lo cual quedan sólo los exones que codificarán finalmente el producto aminoacídico.
Ilustraciones adaptadas de Thompson & Thompson Genetics in Medicine, 6th edition; RL Nussbaum, RR McInnes, HF Willard, The Human Genome: Structure and Function of Genes and Chromosomes, Figura 3-6, pág. 20, Copyright 2001, con permiso de Elsevier.
Región crítica - Critical region
Parte específica de un cromosoma o de un gen que cuando experimenta alguna alteración (deleción, duplicación u otras mutaciones) produce el conjunto característico de anomalías fenotípicas asociadas con un síndrome o enfermedad determinada.
Región de puntos calientes de mutación - Hotspot mutation region
Secuencias de ADN muy susceptibles de ser mutadas debido a una inestabilidad inherente, tendencia hacia un entrecruzamiento desigual o predisposición química a sustituciones de nucleótidos simples; región en la que se observan mutaciones con más frecuencia de la habitual.
Región promotora - Promoter region
Véase Promotor.
Región subtelomérica - Subtelomeric region
Región cromosómica más cercana al telómero (cada uno de los extremos del romosoma) que está compuesta por secuencias repetitivas de ADN muy polimórficas, situadas típicamente en las proximidades de las áreas ricas en genes. Las microdeleciones y las reordenamientos sutiles que alteran los genes en las regiones subteloméricas pueden causar retraso mental; para detectar estas anomalías normalmente es necesario utilizar hibridación fluorescente in situ (FISH) o arrays de CGH, que permiten una evaluación más detallada de las regiones subteloméricas.
Regulación génica - Gene regulation
Es el proceso de activación y desactivación de los genes. En las primeras etapas del desarrollo, las células asumen funciones específicas y la regulación génica asegura que los genes apropiados se expresen en el lugar y momentos adecuados. La regulación génica también contribuye a la respuesta de un organismo a los estímulos procedentes de su entorno. La regulación génica se lleva a cabo por una variedad de mecanismos como el estado de condensación de la cromatina, la modificación química de los genes o la activación o desactivación de los mismos mediante su asociación con proteínas reguladoras.
Reordenamiento - Rearrangement
Alteración estructural de un cromosoma que implica normalmente la rotura y/o la reunión de un segmento de material cromosómico, que da lugar a una configuración anormal; la inversión y la translocación son ejemplos de ello.
Repetición de trinucleótidos - Trinucleotide repeat
Secuencias de tres nucleótidos que se repiten varias veces en tándem en un mismo gen. La variación polimórfica normal en el número de repeticiones, que carece de significación clínica, ocurre normalmente entre los individuos. Los alelos anormalmente grandes se clasifican de menor a mayor tamaño en alelos mutables normales, alelos de penetrancia reducida y alelos de penetrancia completa, respectivamente.
La secuencia de un alelo que contiene cuatro repeticiones CAG tiene la estructura siguiente:
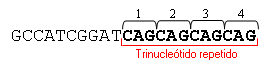
Un trinucleótido repetido con nueve repeticiones CCG tiene la estructura siguiente:
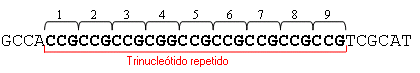
1. A diferencia de otros alelos que son estables de una generación a otra, los alelos que contienen repeticiones de trinucleótidos son inestables y dinámicos y pueden experimentar una expansión cuando son transmitidos de una generación a la siguiente. Las expansiones grandes del número de repeticiones pueden causar anomalías en la función y expresión de un gen. A continuación, se proporcionan algunos ejemplos de trastornos genéticos causados por expansiones de trinucleótidos repetidos:
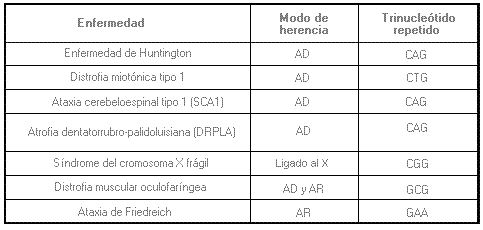
2. Las expansiones causantes de enfermedad, pueden ser pequeñas (A) o grandes (B).
A) Distrofia muscular oculofaríngea
Tamaño de los alelos normales: 6 repeticiones GCG
Tamaño de los alelos de penetrancia completa: 8-13 repeticiones GCG.
B) Distrofia miotónica1
Tamaño de los alelos normales: <30 repeticiones CTG
Tamaño de los alelos de penetrancia completa: 80-2000 repeticiones CTG
3. En algunos trastornos causados por repetición de trinucleótidos, tales como el síndrome del cromosoma X frágil, existe un rango de repeticiones de alelos mutables normales que son inestables y tienden a expandirse cuando son transmitidos de una generación a la siguiente. Los individuos con alelos mutables normales no suelen resultar afectados.
Síndrome del cromosoma X frágil
Tamaño de los alelos normales: ≤58 repeticiones CGG
Tamaño de los alelos mutables normales: 59- ~ 200 repeticiones CGG
Tamaño de los alelos de penetrancia completa: <200 repeticiones CGG
En algunos trastornos causados por repetición de trinucleótidos, tales como la enfermedad de Huntington, existe un rango de alelos de penetrancia reducida (36-39 alelos), por lo que uno de estos alelos podría dar lugar a una enfermedad de comienzo muy tardío y a ninguna manifestación clínica de la enfermedad durante la vida del individuo.
Enfermedad de Huntington
Tamaño de los alelos normales: ≤26 repeticiones CAG
Tamaño de los alelos mutables normales: 27-35 repeticiones CAG
Tamaño de los alelos de penetrancia reducida: 36-39 repeticiones CAG
Tamaño de los alelos de penetrancia completa: ≥40 repeticiones CAG
Repeticiones en tándem de número variable - Variable number tandem repeats (sinónimo: VNTR)
Ordenación lineal de múltiples copias de secuencias cortas repetidas de ADN, de longitud variable pero mayores de tres bases, y muy polimórficas, por lo que resultan útiles como marcadores en el análisis de ligamiento.
Replicación del ADN - DNA replication
La replicación del ADN es el proceso mediante el cual se duplica una molécula (sintetiza una copia idéntica) de ADN. Cuando una célula se divide, en primer lugar, debe duplicar su genoma para que cada célula hija contenga un juego completo de cromosomas. De esta manera se garantiza la transmisión de la información genética.
Resultado falso negativo - False negative result
Resultado de una prueba que indica que un individuo no está afectado y/o no presenta una mutación génica determinada, cuando en realidad está afectado y/o presenta tal mutación; es decir, cuando el resultado de una prueba realizada en un individuo afectado es negativo.
Resultado falso positivo - False positive result
Resultado de una prueba que indica que un individuo está afectado y/o presenta una mutación génica determinada, cuando en realidad no está afectado y/o no presenta tal mutación; es decir, cuando el resultado de una prueba realizada en un individuo que realmente no está afectado es positivo.
Riesgo - Risk
En el contexto de la Genética, el término riesgo se refiere a la probabilidad de que un individuo se vea afectado por una determinada enfermedad. Tanto los genes como el medio ambiente pueden jugar un papel en la aparición de la enfermedad. El riesgo de un individuo será mayor en la medida en que haya heredado un gen o genes que incrementan la susceptibilidad a determinada enfermedad. Por otro lado, la probabilidad de desarrollar la enfermedad puede ser mayor en individuos que viven o trabajan en un ambiente que promueve el desarrollo de esa enfermedad.
Riesgo de recurrencia - Recurrence risk
Probabilidad de que un rasgo o trastorno que está presente en un miembro de una familia, se manifieste de nuevo en otros miembros de la familia en la misma o en posteriores generaciones.
Riesgo hereditario - Background risk (sinónimo: riesgo poblacional)
Proporción de individuos en una población determinada que están afectados por una patología específica o que presentan mutaciones en un gen determinado; en el proceso de asesoramiento genético esto se determina generalmente mediante una comparación con el riesgo personal del paciente, dados sus antecedentes familiares u otras circunstancias.

Riesgo poblacional - Population risk (sinónimo: riesgo basal)
Proporción de individuos en la población general que están afectados por un trastorno específico o son portadores de un gen determinado. En el proceso de asesoramiento genético se utiliza la cifra de riesgo poblacional en comparación con el riesgo personal del paciente derivado de sus antecedentes familiares u otras circunstancias.
S
Secuenciación del ADN - DNA sequencing
Es el conjunto de métodos que tienen por objetivo determinar el orden exacto de los nucleótidos (A, C, G y T) en la molécula de ADN. La secuencia de bases de ADN lleva la información que una célula necesita para originar moléculas de ARN y proteínas. Los avances en las técnicas de secuenciación de ADN han sido muy notables en los años recientes gracias al Proyecto Genoma Humano. Es posible aplicar estas técnicas para conocer la secuencia de un único gen, de varios genes (paneles), al conjunto de genes de un individuo (exoma) o, finalmente a todo el genoma de un individuo.
El método de secuenciación del ADN desarrollado por Frederick Sanger y colaboradores en 1977 ha sido el de uso más extendido en los últimos 40 años. En los años más recientes ese método está siendo sustituido por la secuenciación de nueva generación (“next-generation sequencing”, NGS, o los términos sinónimos “massively parallel” o “deep sequencing”) que ha revolucionado el mundo de la investigación biomédica al ser capaz de analizar simultáneamente millones de pequeños fragmentos de ADN. La tecnología de NGS permite disponer en horas de la secuencia del exoma o del genoma humano completo.
Segregación - Segregation
Separación de los cromosomas homólogos y su distribución aleatoria en los diferentes gametos en la meiosis. Se aplica también a la separación de los alelos de un locus y su distribución en las células hijas que se produce durante la meiosis o en la mitosis.
Sensibilidad - Sensitivity
Frecuencia con la que una prueba proporciona un resultado positivo cuando el individuo sometido a dicha prueba está en realidad afectado y/o presenta la mutación génica en cuestión.
Serina - Serine
La serina (abreviada Ser o S) es uno de los veinte aminoácidos que pueden componer las proteínas. Es un aminoácido no esencial, lo que significa que se requiere para el cuerpo humano para funcionar correctamente, pero no tiene que provenir de una fuente externa (en la dieta) puesto que el cuerpo es capaz de sintetizarlo internamente. En su lugar, se produce típicamente en el cuerpo a partir de metabolitos como la glicina. Sus codones son UCU, UCC, UCA, UCG, AGU y AGC.
Silvestre - Wild type
El alelo que se encuentra con mayor frecuencia en la naturaleza, y que por tanto se designa arbitrariamente como "normal".
Símbolo genético - Gene symbol
Abreviatura exclusiva del nombre de un gen, compuesta por letras del alfabeto latino mayúsculas y en cursiva y por números arábigos, que es asignada formalmente por el HUGO Gene Nomenclature Committee una vez identificado un gen (Nota: los posibles genes pueden ser denominados por su nombre de locus antes de que sean identificados).
La definición siguiente está adaptada de Genew: The Human Gene Nomenclature Database (www.gene.ucl.ac.uk/nomenclature/):
El “símbolo genético” debe estar compuesto preferentemente por un máximo de seis caracteres. Se pretende que el “nombre genético o nombre completo del gen” que es descriptivo y más largo, sea conciso y exprese el carácter o la función del gen. La primera letra del símbolo genético debe ser la misma que la del nombre del gen, con objeto de facilitar la ordenación y clasificación alfabética, p. ej. el gen denominado “cáncer de mama, comienzo precoz 1” tiene el símbolo “BRCA1”. Cada gen humano conocido tiene un nombre y símbolo genético aprobado por el comité HUGO. Todos los símbolos aprobados están incluidos en la base de datos Human Gene Nomenclature de Genew. Cada símbolo es único y se asegura que se proporciona a cada gen sólo un símbolo aprobado. Es necesario proporcionar un símbolo único a cada gen de manera que todos podamos hablar de ellos sabiendo de lo que se trata, esto también facilita la recuperación de datos de publicaciones por medios electrónicos. Preferentemente cada símbolo mantiene una estructura paralela en los diferentes miembros de una familia de genes y pueden ser utilizados también en otras especies, en particular el ratón. El HUGO ha aprobado ya los símbolos de más de 13.000 genes, lo que representa la tercera parte del total de los genes humanos, que se estiman en 30.000. Los científicos y responsables de publicaciones científicas (p. ej. Genomics, Nature Genetics and Cytogenetics and Cell Genetics) y bases de datos (p. ej. RefSeq, OMIM, GDB, MGD y LocusLink) siguen solicitando símbolos nuevos individuales. También hay una demanda de grupos de símbolos nuevos por parte de los que investigan familias de genes, segmentos de cromosomas o cromosomas enteros. Puesto que el análisis de la secuencia del genoma humano está casi completo, hay una demanda creciente por la aprobación rápida de símbolos génicos. Sin embargo, en todos los casos se están realizando esfuerzos considerables por utilizar un símbolo aceptable por todos aquellos que trabajan en este campo.
Síndrome de genes contiguos - Contiguous gene syndrome
Síndrome de microdeleción - Microdeletion syndrome (sinónimo: síndrome de deleción de genes contiguos)
Síndrome causado por una deleción cromosómica que comprende varios genes y es demasiado pequeña para poder ser detectada al microscopio utilizando métodos citogenéticos convencionales. Dependiendo del tamaño de la deleción, se pueden utilizar ocasionalmente otras técnicas, tales como FISH u otros métodos de análisis de ADN, para su identificación.
Sitio de restricción - Restriction site
Secuencia de ADN que es reconocida por una endonucleasa (una proteína que corta el ADN) como el lugar en el que se cortará el ADN, o también llamado "diana de corte".
SNP - Single Nucleotide Polimorphism
Variación en la secuencia del adn (con respecto a una secuencia consenso) que afecta a una única base (A,T,G,C) en posiciones concretas del genoma (1 cada 1000 bases en promedio), y que se observa en la población con una frecuencia de al menos 1%. En la especie humana se estima que existen en torno a de 12 millones de SNPs, de los que a finales de 2007 se han identificado algo más de un tercio.
Sobrecruzamiento desigual - Unequal crossing over
Emparejamiento incorrecto e intercambio de ADN entre regiones de cromosomas homólogos o no homólogos, pero genéticamente similares, que da lugar a la duplicación o deleción del ADN en las células hijas.
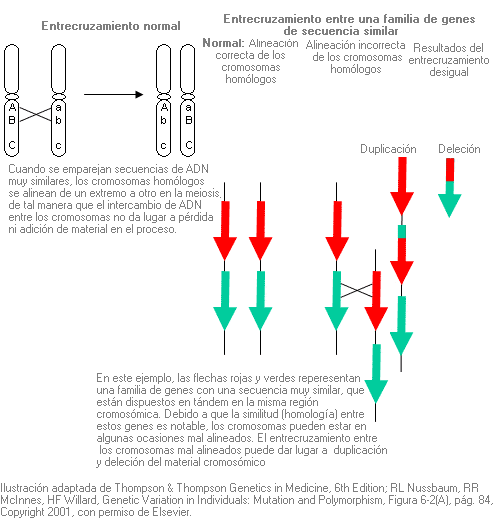 Situaciones más susceptibles a un entrecruzamiento desigual:
Situaciones más susceptibles a un entrecruzamiento desigual:
Miembros de “familias” multigénicas cuyos genes comparten una homología significativa entre sí (p. ej. grupo de genes de alfaglobina; grupo de genes de pigmentos visuales rojos y verdes).
Cuando un gen y un pseudogen están dispuestos en tándem en un cromosoma (p. ej. CYP21 y su pseudogen CYP21P; SMN1y SMN2).
Cuando hay secuencias similares a lo largo de la misma cadena de ADN que predisponen a inversiones o deleciones (p. ej. el gen F8; síndromes de genes contiguos, tales como deleción en 22q11.2).
Cuando hay secuencias no codificadoras homólogas (p. ej. familia Alu de secuencias repetidas de ADN dispersas en todo el genoma).
Sonda - Probe
Southern blot - Southern blot: (sinónimos: Southern blotting, análisis de Southern blotting)
Técnica de análisis de genética molecular que se utiliza para detectar diferencias en la longitud de los fragmentos de ADN tras ser digeridos por enzimas de restricción (lo que se conoce normalmente como RFLP: polimorfismos de longitud de fragmentos de restricción). La longitud de los fragmentos de restricción es variable en tamaño, desde pocas bases a kilobases.
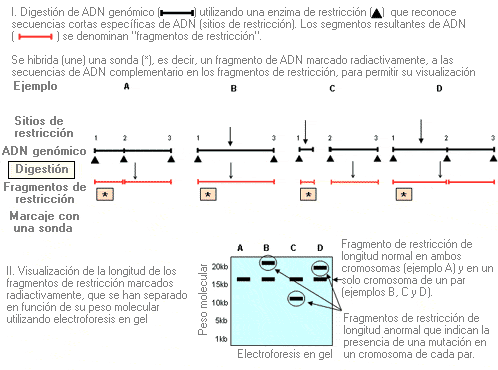
Splicing (corte y empalme) - Splicing (sinónimo: mutación por splicing)
Proceso mediante el cual los intrones, es decir, las regiones no codificadoras de los genes, son escindidos del transcripto de ARN mensajero primario y los exones (es decir, las regiones codificadoras) se unen para generar ARN mensajero maduro.
Sustitución - Substitution
Es el cambio o sustitución de una base por otra en la secuencia de ADN. Se conoce como “transición” cuando la base cambia por otra del mismo tipo: purina (A y G) por purina o pirimidina (C y T) por pirimidina. En la “transversión” el cambio es de una purina por una pirimidina o de una pirimidina por una purina.
T
TagSNP - TagSNP
Es un SNP (Single Nucleotide Polymorphim) o polimorfismo de una base en una región del genoma en la que existe un alto desequilibrio de ligamiento, y que representa al conjunto de SNPs dentro ese bloque haplotípico. Con el análisis de los tagSNPs es posible inferir la totalidad de la variación genética e identificar asociaciones fenotípicas sin genotipar el resto de los SNPs en esta región cromosómica.
Tasa de mutación - Mutation rate
La tasa de mutación de un gen o una secuencia de ADN es la frecuencia en la que se producen nuevas mutaciones en ese gen o la secuencia en cada generación.
TCGA -
Véase el Atlas del Genoma del Cáncer.
Técnica de bandas - Banding technique
Empleo de una de las diversas técnicas de coloración disponibles para teñir los cromosomas que se encuentran en una fase determinada del proceso de división celular. Los cromosomas adquieren de este modo un patrón específico de franjas (bandas) claras y oscuras que permiten identificarlos y evaluar su estructura.
Telofase - Telophase
Es la cuarta y última fase de la mitosis celular. Es la reversión de los procesos que tuvieron lugar durante la profase y prometafase. Los cromosomas ya están separados en dos grupos de cromátidas en los dos extremos de la célula. Las cromátidas se desenrollan y el ADN vuelve a tomar forma de hilos de cromatina difusa. Se crea alrededor de cada grupo una membrana nuclear. El huso mitótico desaparece.
Telomerasa - Telomerase
La telomerasa es una ribonucleoproteína cuya actividad transcriptasa inversa es la de añadir repeticiones de novo TTAGGG en los extremos de los telómeros para así poder mantener una longitud telomérica estable. El mantenimiento de la longitud telomérica, requiere un equilibrio entre erosión y alargamiento, para evitar un acortamiento excesivo o una mayor actividad que podría favorecer la inmortalidad de células malignas.
Telómero - Telomere
Los telómeros son los extremos de los cromosomas. Son regiones de ADN no codificante, altamente repetitivas (5'-TTAGGG-3') cuya función principal es la estabilidad estructural de los cromosomas en las células eucariotas, la división celular y el tiempo de vida de las estirpes celulares.
Temperatura de melting (Tm) - Melting Temperature
También llamada temperatura de fusión del ADN es la temperatura en la cual el 50% del ADN tiene sus hebras separadas (desnaturalización del ADN).
Templado (hebra) - Template (strand)
Hebra del ADN dúplex que actúa como molde para la transcripción.
Terapia génica - Gene Therapy
Método para la corrección de una enfermedad genética basado en la transferencia de material genético a las células del paciente con la finalidad de restablecer una función celular que estaba abolida o defectuosa, introducir una nueva función o bien interferir con una función existente.
Termociclador - Thermocycler
También conocido como máquina de PCR es un instrumento usado en biología molecular que permite realizar de forma automatizada y secuencial los ciclos de temperaturas necesarios para llevar a cabo la reacción en cadena de la polimerasa (PCR) de amplificación de ADN.
Tétrada - Tetrad
Conjunto de las cuatro cromátides (dos por cada cromosoma homólogo de un bivalente) que están en sinapsis durante la primera división meiótica.
Timina - Thymine
Es una de las cinco bases nitrogenadas constituyentes de los ácidos nucleicos (las otras cuatro son la adenina, la guanina, la citosina, y el uracilo, este último sólo presente en el ARN). Es un compuesto heterocíclico derivado de la pirimidina que parte del ADN y en el código genético se representa con la letra T.
Tirosina - Tyrosine
La tirosina es uno de los 20 aminoácidos que forman las proteínas. Se clasifica como un aminoácido no esencial en los mamíferos ya que su síntesis se produce a partir de la hidroxilación de otro aminoácido: la fenilalanina. Sus abreviaturas son Tyr e Y.
Traducción - Translation
Proceso de biosíntesis proteica donde se convierte una secuencia de ARN mensajero en una cadena de aminoácidos para formar una proteína. Previa a la traducción viene el proceso de la transcripción. Las fases de la traducción son tres: iniciación, elongación y terminación y este proceso se lleva a cabo tanto en citoplasma de la célula como en el retículo endoplasmático rugoso.
Transcripción - Transcription
Es la síntesis enzimática de ARN utilizando un templado de ADN. Es el primer proceso de la expresión génica donde se transfiere la información contenida en la secuencia del ADN hacia la secuencia de la proteína utilizando diversos ARN como intermediarios. Esta secuencia de ADN es copiada a ARN con una enzima llamada ARN polimerasa que sintetiza ARN mensajero que contiene la información de la secuencia del ADN. Este proceso también podría llamarse síntesis del ARN mensajero.
Transcriptasa Inversa - Reverse transcriptase
También llamada retrotranscriptasa es una enzima de tipo ADN-polimerasa, codificada por retrovirus, cuya función es sintetizar ADN de doble cadena utilizando como templado una molécula de ARN monocatenario, es decir, catalizar la retrotranscripción o transcripción inversa.
Transcriptoma - Transcriptome
Es el conjunto, distribución y número de tránscritos de ARN que se genera desde el núcleo al citoplasma como paso intermedio para generar proteínas.
Transcrito primario - Primary transcript
Es una molécula de ARN monocatenario que se obtiene inmediatamente después de la transcripción. El transcrito primario de eucariotas se caracteriza, básicamente, porque está formado por la unión de los intrones o fragmentos no codificantes y los exones o regiones codificantes. En eucariontes estos transcritos primarios requieren de modificación post transcripcional para la obtención de moléculas de ARN plenamente funcionales. En este proceso de maduración, se eliminan los intrones, transformando el transcrito primario en ARN mensajero maduro que será convertido en proteína en el proceso de la traducción.
Transfección - Transfection
Término general utilizado para referirse a la introducción de ADN exógeno al interior de una célula eucariota.
Transferencia genética - Gene transfer
Transferencia de material genético, que puede comprender desde un pequeño segmento de adn hasta un genoma entero, de una célula humana a otro tipo de célula en cultivo para estudiar la frecuencia con que los marcadores genéticos conocidos son transferidos conjuntamente al genoma receptor; se utiliza para determinar la proximidad física de los marcadores genéticos en el genoma humano; se utiliza asimismo para estudiar la expresión y regulación génica.
Transgénico - Transgenic
Célula u organismo que contiene en su línea germinal un ADN exógeno introducido experimentalmente.
Transición - Transition
Sustitución de una base púrica por otra púrica y una pirimidina por otra pirimidina.
Translocación - Translocation
Este término se emplea cuando cambia la ubicación de determinado material cromosómico. Existen dos tipos principales de translocaciones: recíprocas y robertsonianas.
Translocación críptica - Cryptic chromosome translocation
Translocación o reordenamiento cromosómico que requiere técnicas especiales (p. ej. hibridación fluorescente in situ [FISH], detección telomérica) para su detección, dado que es demasiado pequeña para ser identificada con técnicas citogenéticas convencionales.
Translocación recíproca - Reciprocal translocation
En una translocación recíproca, se produce el intercambio de segmentos entre dos cromosomas entre dos cromosomas no homólogos.
Una translocación recíproca puede ser:
Equilibrada (cuando no hay pérdida ni adición de material cromosómico). Las translocaciones equilibradas no suelen estar asociadas con anomalías fenotípicas; sin embargo, en casos raros, las alteraciones en los puntos de rotura pueden afectar a un gen y causar efectos adversos, incluyendo algunos trastornos genéticos conocidos.
No equilibrada (cuando hay pérdida o adición de material cromosómico). Las translocaciones no equilibradas casi siempre dan lugar a un fenotipo anormal.

Translocación robertsoniana - Robertsonian translocation
Unión de dos cromosomas acrocéntricos por los centrómeros, con pérdida de sus brazos cortos y formando un solo cromosoma y este nuevo cromosoma se denomina "cromosoma por translocación. En los cromosomas acrocéntricos, el centrómero está localizado cerca del extremo del cromosoma; y son el 13, 14, 15, 21 y 22.
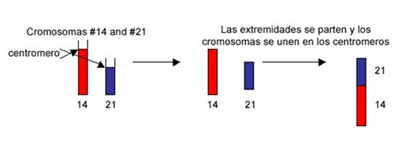
Transposon (Tn) -
Son regiones de ADN (200-2000 pares de bases) capaces de moverse de manera autosuficiente de una parte a otra parte del genoma, mediante un mecanismo de corta-pega y cuya consecuencia es la producción de mutaciones.
Transversión - Transversion
Sustitución de una base púrica por pirimidina o viceversa.
Treonina - Threonine
La treonina (abreviada Thr o T) es uno de los veinte aminoácidos que componen las proteínas; su cadena lateral es hidrófila. Está codificada en el ARN mensajero como ACU, ACC, ACA o ACG. La treonina no es sintetizada en suficiente cantidad en humanos, por lo que debe ingerirse en proteínas que consume.
Triploide - Triploid
Célula u organismo que posee tres complementos cromosómicos, de manera que posee un número total de cromosomas que es triple del haploide. La célula por lo tanto es 3N.
Triptófano - Tryptophan
El triptófano (abreviado como Trp o W) es un aminoácido esencial en la nutrición humana. Es uno de los 20 aminoácidos incluidos en el código genético (codón UGG). Se clasifica entre los aminoácidos apolares, también llamados hidrófobos.
Trisomía - Trisomy
Una trisomía es aquella situación en la que existen tres cromosomas de un mismo tipo en un organismo diploide: en vez de un par homólogo de cromosomas es un triplete (2n + 1). El ejemplo más frecuente es la trisomía 21, que produce el síndrome de Down.
U
Unidad de Mapa - map unit (m.u.)
Es la unidad utilizada para medir la distancia de mapa entre genes ligados (ubicados en el mismo cromosoma). Una unidad de mapa genético de ligamiento en eucariotas diploides equivale a una frecuencia de 1% de recombinación o lo que es lo mismo 1 centiMorgan (abreviado cM).
Unidad transcripcional - Transcriptional unit
Es un concepto muy amplio que engloba a cualquier segmento de ADN transcrito a un único ARN, independiente del número de genes de los que contenga información.
Uracilo - Uracil
Es una pirimidina, una de las cuatro bases nitrogenadas que forman parte del ARN y en el código genético se representa con la letra U. El uracilo reemplaza en el ARN a la timina que es una de las cuatro bases nitrogenadas que forman el ADN. Al igual que la timina, el uracilo siempre se empareja con la adenina mediante dos puentes de hidrógeno, pero le falta el grupo metilo.
V
Valina - Valine
La valina (abreviada Val o V) es uno de los veinte aminoácidos codificados por el ADN. En el ARN mensajero, está codificada por GUA, GUG, GUU o GUC. Nutricionalmente, en humanos, es uno de los aminoácidos esenciales.
Valor C - C Value
La cantidad de ADN por genoma haploide (un solo juego cromosómico), en el caso de los organismos diploides como el ser humano, el valor C es la cantidad correspondiente a un juego de 23 cromosomas.
Valor predictivo negativo - Negative predictive value
Probabilidad de que un individuo que presenta un resultado negativo en una prueba, sea realmente no afectado y/o no presente la mutación del gen en cuestión.
Valor predictivo positivo - Positive predictive value (sinónimo: PPV)
Probabilidad de que un individuo que presenta un resultado positivo en una prueba sea en realidad portador del gen en cuestión, o desarrolle la enfermedad.
Variabilidad genética - Genetic variability
Se denomina variabilidad genética a los cambios genéticos que aparecen en los genes de una especie o de una población. La variabilidad genética permite la evolución de las especies, ya que en cada generación solamente una fracción de la población sobrevive y se reproduce transmitiendo características particulares a su progenie. De esta manera, a mayor variabilidad genética, mayor velocidad en el desarrollo de los cambios evolutivos. La variabilidad genética se origina por mutaciones y recombinaciones genéticas. Los procesos que dirigen o eliminan variabilidad genética son la selección natural y la deriva genética.
Variabilidad interfamiliar - Interfamilial variability
Diversidad o variabilidad en la presentación clínica de una enfermedad determinada en los individuos afectados de diferentes familias.
Variabilidad intrafamiliar - Intrafamilial variability
Diversidad o variabilidad en la presentación clínica de una enfermedad determinada en los individuos afectados de la misma familia directa o lejana.
Variante alélica de significado desconocido - Allelic variant of unknown significance
Alteración de la secuencia normal de un gen, cuyo significado no está claro hasta realizar un estudio adicional del genotipo y de su correspondiente fenotipo en una población suficientemente amplia; la secuenciación completa de un gen permite identificar generalmente numerosas (a veces cientos) variantes alélicas de un gen determinado.
Ejemplos de variantes alélicas:
· Variante alélica normal: Cambio (mutación) que no interfiere en la función normal del gen y está asociado con un fenotipo normal; se conoce también como polimorfismo benigno. Nota: En GeneReviews, la composición del gen con función normal se describe generalmente bajo este epígrafe en el texto, lo que implica que los alelos salvajes y los polimorfismos benignos se incluyan en la misma categoría; sin embargo, el término “variante” implica técnicamente un cambio con respecto al tipo salvaje.
· Variante alélica patológica: Cambio (mutación) que altera la función normal del gen y da lugar a un fenotipo anormal; se conoce también como mutación patológica.
· Variante alélica de significado desconocido: Cambio (mutación) cuyo resultado es un fenotipo desconocido. Nota: Las variantes alélicas de significado desconocido deben ser estudiadas en poblaciones amplias, para determinar si se pueden clasificar definitivamente como variantes alélicas normales o patológicas.
Es probable que la secuenciación completa de cualquier gen revele numerosas variantes alélicas. Consideremos los ejemplos siguientes:
· Gen CFTR asociado a fibrosis quística: Se han documentado más de 2000 variantes alélicas con más de 900 mutaciones patológicas conocidas.
· Gen PAH asociado a fenilcetonuria: Se han documentado más de 3000 variantes alélicas con más de 400 mutaciones patológicas conocidas.
· Gen BRCA1 asociado a cáncer de mama y de ovario: Se han documentado más de 600 variantes alélicas de las cuales aproximadamente un tercio son de significado clínico desconocido.
Variante benigna - Benign variant (sinónimo: polimorfismo)
Ventaja selectiva - Selective advantage
La característica de un organismo que le permite sobrevivir y reproducirse mejor que otros organismos en un grupo poblacional bajo un ambiente determinado siendo la base de la evolución por selección natural. Básicamente existe un incremento de la eficacia biológica producido por un genotipo determinado, de manera que la frecuencia de ese genotipo tiende a aumentar en la población.
VIH - HIV (Human immunodeficiency virus)
Acrónimo de Virus de la Inmunodeficiencia Humana, miembro del grupo Lentivirus de los Retrovirus que causa la infección por VIH y puede llevar al desarrollo del síndrome de inmunodeficiencia adquirida (SIDA) aunque no todas las personas con VIH desarrollan SIDA. Este virus daña el sistema inmunitario mediante la destrucción de los glóbulos blancos que combaten las infecciones poniendo en riesgo al individuo a contraer infecciones importantes y ciertos tipos de cáncer.
Virus - Virus
Agente infeccioso que posee material genético en forma de ADN o ARN con una envuelta proteica y que es incapaz de llevar a cabo actividades metabólicas o reproducirse por sí solo, teniendo que invadir una célula huésped (procarionte, eucarionte vegetal, eucarionte animal) para obtener la energía para sobrevivir.
VNTR - Variable Number of Tandem Repeats
Acrónimo inglés de "Variable Number of Tandem Repeats". Es una región en el genoma hipervariable donde una secuencia de entre 6-25 nucleótidos aparece repetida en tándem. Estas regiones son polimórficas, por lo que cada individuo presenta diferente número de repeticiones. También se les conoce como minisatélites, y son utilizados como marcadores genéticos en estudios de ligamiento y en genética forense.
W
Western blot - Western blot
Separación de proteínas en un gel electroforético para su identificación mediante técnicas inmunológicas.
Wild-type -
Wild-type significa en inglés 'salvaje' o 'silvestre' y se refiere a la forma más común, aquella que predomina en la población. Los genetistas llaman los individuos con la versión 'normal' de una característica individuos wild-type y los individuos con una característica única los denominan mutantes. Si nos referimos a variantes genéticas el alelo wild-type es el alelo que se encuentra en mayor frecuencia en la población general.
X
XX -
Representación de los cromosomas sexuales de una mujer (el llamado par 23 de cromosomas), que tiene dos cromosomas X, un cromosoma X de cada progenitor.
XY -
Representación de los cromosomas sexuales de un hombre (el llamado par 23 de cromosomas), que hereda el cromosoma X de su madre y el cromosoma Y de su padre.
Y
YAC (Yeast Artificial Chromosome) -
Vector de clonamiento en la forma de un cromosoma artificial de levadura. Es un vector que pretende imitar las características de un cromosoma normal de una levadura (eucariota), portando un centrómero y telómeros terminales. Esto permite clonar en levaduras, largas secuencias de ADN eucarionte (200 kb - 3.000 kb).
Z
Zigoto - Zygote
Es la célula resultante de la fusión del gameto masculino y el gameto femenino en la reproducción sexual de los organismos y que luego se dividirá mitóticamente para dar lugar a un individuo diploide.
Zipper de Leucina - Leucine zipper
También llamado cremallera de leucina es un motivo supersecundario de proteínas que consta de dos segmentos peptídicos en hélice alfa. En un extremo, estas hélices interaccionan entre sí gracias a la interacción entre cadenas laterales de leucina y en el otro lo hacen con secuencias específicas de bases en el surco mayor del ADN. Es un dominio de dimerización de expresión, en particular en proteínas de la clase factores de transcripción.