- Todos
- A
- B
- C
- D
- E
- F
- G
- H
- I
- J
- K
- L
- M
- N
- Ñ
- O
- P
- Q
- R
- S
- T
- U
- V
- W
- X
- Y
- Z
S
Secuenciación del ADN - DNA sequencing
Es el conjunto de métodos que tienen por objetivo determinar el orden exacto de los nucleótidos (A, C, G y T) en la molécula de ADN. La secuencia de bases de ADN lleva la información que una célula necesita para originar moléculas de ARN y proteínas. Los avances en las técnicas de secuenciación de ADN han sido muy notables en los años recientes gracias al Proyecto Genoma Humano. Es posible aplicar estas técnicas para conocer la secuencia de un único gen, de varios genes (paneles), al conjunto de genes de un individuo (exoma) o, finalmente a todo el genoma de un individuo.
El método de secuenciación del ADN desarrollado por Frederick Sanger y colaboradores en 1977 ha sido el de uso más extendido en los últimos 40 años. En los años más recientes ese método está siendo sustituido por la secuenciación de nueva generación (“next-generation sequencing”, NGS, o los términos sinónimos “massively parallel” o “deep sequencing”) que ha revolucionado el mundo de la investigación biomédica al ser capaz de analizar simultáneamente millones de pequeños fragmentos de ADN. La tecnología de NGS permite disponer en horas de la secuencia del exoma o del genoma humano completo.
Segregación - Segregation
Separación de los cromosomas homólogos y su distribución aleatoria en los diferentes gametos en la meiosis. Se aplica también a la separación de los alelos de un locus y su distribución en las células hijas que se produce durante la meiosis o en la mitosis.
Sensibilidad - Sensitivity
Frecuencia con la que una prueba proporciona un resultado positivo cuando el individuo sometido a dicha prueba está en realidad afectado y/o presenta la mutación génica en cuestión.
Serina - Serine
La serina (abreviada Ser o S) es uno de los veinte aminoácidos que pueden componer las proteínas. Es un aminoácido no esencial, lo que significa que se requiere para el cuerpo humano para funcionar correctamente, pero no tiene que provenir de una fuente externa (en la dieta) puesto que el cuerpo es capaz de sintetizarlo internamente. En su lugar, se produce típicamente en el cuerpo a partir de metabolitos como la glicina. Sus codones son UCU, UCC, UCA, UCG, AGU y AGC.
Silvestre - Wild type
El alelo que se encuentra con mayor frecuencia en la naturaleza, y que por tanto se designa arbitrariamente como "normal".
Símbolo genético - Gene symbol
Abreviatura exclusiva del nombre de un gen, compuesta por letras del alfabeto latino mayúsculas y en cursiva y por números arábigos, que es asignada formalmente por el HUGO Gene Nomenclature Committee una vez identificado un gen (Nota: los posibles genes pueden ser denominados por su nombre de locus antes de que sean identificados).
La definición siguiente está adaptada de Genew: The Human Gene Nomenclature Database (www.gene.ucl.ac.uk/nomenclature/):
El “símbolo genético” debe estar compuesto preferentemente por un máximo de seis caracteres. Se pretende que el “nombre genético o nombre completo del gen” que es descriptivo y más largo, sea conciso y exprese el carácter o la función del gen. La primera letra del símbolo genético debe ser la misma que la del nombre del gen, con objeto de facilitar la ordenación y clasificación alfabética, p. ej. el gen denominado “cáncer de mama, comienzo precoz 1” tiene el símbolo “BRCA1”. Cada gen humano conocido tiene un nombre y símbolo genético aprobado por el comité HUGO. Todos los símbolos aprobados están incluidos en la base de datos Human Gene Nomenclature de Genew. Cada símbolo es único y se asegura que se proporciona a cada gen sólo un símbolo aprobado. Es necesario proporcionar un símbolo único a cada gen de manera que todos podamos hablar de ellos sabiendo de lo que se trata, esto también facilita la recuperación de datos de publicaciones por medios electrónicos. Preferentemente cada símbolo mantiene una estructura paralela en los diferentes miembros de una familia de genes y pueden ser utilizados también en otras especies, en particular el ratón. El HUGO ha aprobado ya los símbolos de más de 13.000 genes, lo que representa la tercera parte del total de los genes humanos, que se estiman en 30.000. Los científicos y responsables de publicaciones científicas (p. ej. Genomics, Nature Genetics and Cytogenetics and Cell Genetics) y bases de datos (p. ej. RefSeq, OMIM, GDB, MGD y LocusLink) siguen solicitando símbolos nuevos individuales. También hay una demanda de grupos de símbolos nuevos por parte de los que investigan familias de genes, segmentos de cromosomas o cromosomas enteros. Puesto que el análisis de la secuencia del genoma humano está casi completo, hay una demanda creciente por la aprobación rápida de símbolos génicos. Sin embargo, en todos los casos se están realizando esfuerzos considerables por utilizar un símbolo aceptable por todos aquellos que trabajan en este campo.
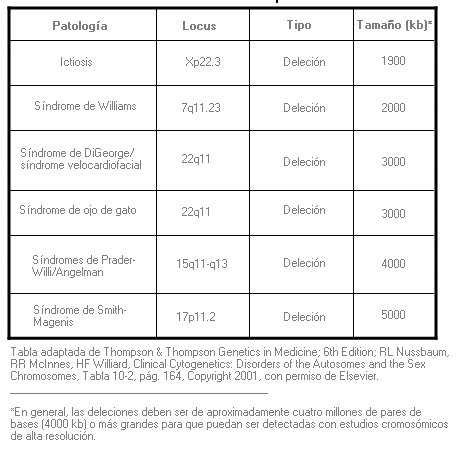
Síndrome de genes contiguos - Contiguous gene syndrome
Síndrome de microdeleción - Microdeletion syndrome (sinónimo: síndrome de deleción de genes contiguos)
Síndrome causado por una deleción cromosómica que comprende varios genes y es demasiado pequeña para poder ser detectada al microscopio utilizando métodos citogenéticos convencionales. Dependiendo del tamaño de la deleción, se pueden utilizar ocasionalmente otras técnicas, tales como FISH u otros métodos de análisis de ADN, para su identificación.
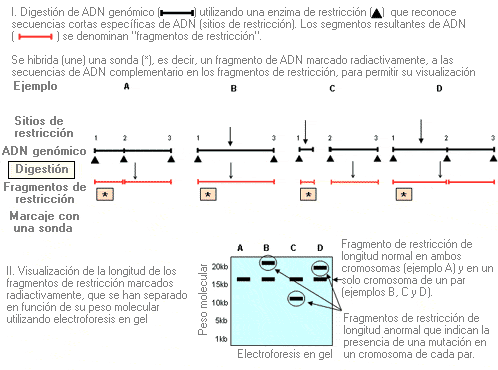
Sitio de restricción - Restriction site
Secuencia de ADN que es reconocida por una endonucleasa (una proteína que corta el ADN) como el lugar en el que se cortará el ADN, o también llamado "diana de corte".
SNP - Single Nucleotide Polimorphism
Variación en la secuencia del adn (con respecto a una secuencia consenso) que afecta a una única base (A,T,G,C) en posiciones concretas del genoma (1 cada 1000 bases en promedio), y que se observa en la población con una frecuencia de al menos 1%. En la especie humana se estima que existen en torno a de 12 millones de SNPs, de los que a finales de 2007 se han identificado algo más de un tercio.
Sobrecruzamiento desigual - Unequal crossing over
Emparejamiento incorrecto e intercambio de ADN entre regiones de cromosomas homólogos o no homólogos, pero genéticamente similares, que da lugar a la duplicación o deleción del ADN en las células hijas.
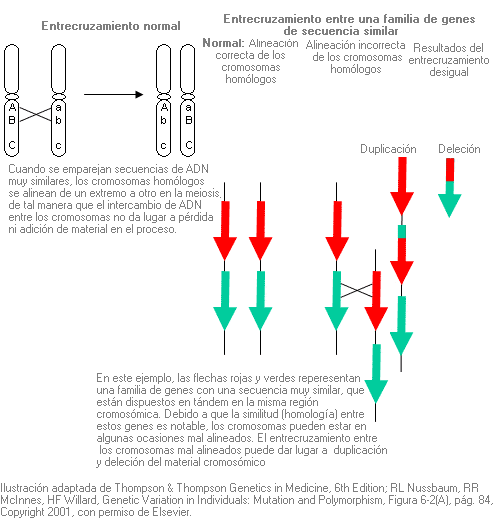 Situaciones más susceptibles a un entrecruzamiento desigual:
Situaciones más susceptibles a un entrecruzamiento desigual:
Miembros de “familias” multigénicas cuyos genes comparten una homología significativa entre sí (p. ej. grupo de genes de alfaglobina; grupo de genes de pigmentos visuales rojos y verdes).
Cuando un gen y un pseudogen están dispuestos en tándem en un cromosoma (p. ej. CYP21 y su pseudogen CYP21P; SMN1y SMN2).
Cuando hay secuencias similares a lo largo de la misma cadena de ADN que predisponen a inversiones o deleciones (p. ej. el gen F8; síndromes de genes contiguos, tales como deleción en 22q11.2).
Cuando hay secuencias no codificadoras homólogas (p. ej. familia Alu de secuencias repetidas de ADN dispersas en todo el genoma).
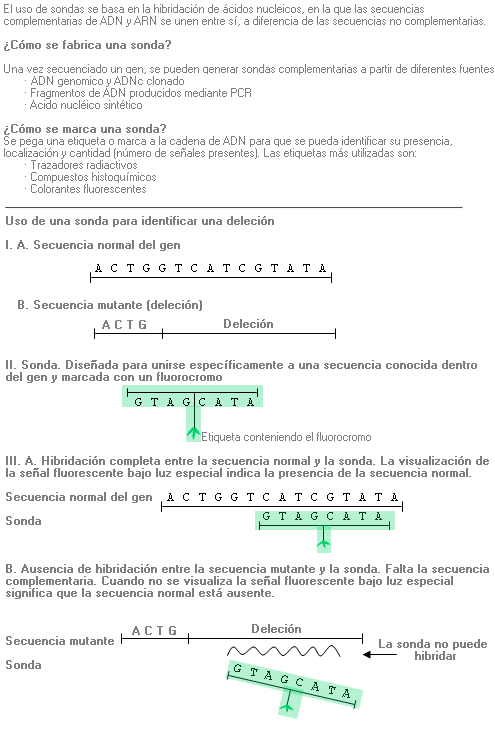
Sonda - Probe
Southern blot - Southern blot: (sinónimos: Southern blotting, análisis de Southern blotting)
Técnica de análisis de genética molecular que se utiliza para detectar diferencias en la longitud de los fragmentos de ADN tras ser digeridos por enzimas de restricción (lo que se conoce normalmente como RFLP: polimorfismos de longitud de fragmentos de restricción). La longitud de los fragmentos de restricción es variable en tamaño, desde pocas bases a kilobases.
Splicing (corte y empalme) - Splicing (sinónimo: mutación por splicing)
Proceso mediante el cual los intrones, es decir, las regiones no codificadoras de los genes, son escindidos del transcripto de ARN mensajero primario y los exones (es decir, las regiones codificadoras) se unen para generar ARN mensajero maduro.
Sustitución - Substitution
Es el cambio o sustitución de una base por otra en la secuencia de ADN. Se conoce como “transición” cuando la base cambia por otra del mismo tipo: purina (A y G) por purina o pirimidina (C y T) por pirimidina. En la “transversión” el cambio es de una purina por una pirimidina o de una pirimidina por una purina.