- Todos
- A
- B
- C
- D
- E
- F
- G
- H
- I
- J
- K
- L
- M
- N
- Ñ
- O
- P
- Q
- R
- S
- T
- U
- V
- W
- X
- Y
- Z
P
Panel de genes - Multigene panel
Prueba genética en la que se analiza simultáneamente el estado de un grupo de genes asociados con una determinada patología. Son muy usados los que analizan el conjunto de genes asociados con la susceptibilidad a desarrollar cáncer colorrectal, de mama, etc., o los genes relacionados con la discapacidad intelectual. Los avances en las técnicas de secuenciación permiten analizar en un único test un número prácticamente ilimitado de genes. Frente al habitual análisis de un único gen, el uso de estos paneles se ha extendido en los años más recientes tanto en el terreno de la investigación, como en la práctica clínica ya que permiten una valoración potencialmente más completa de los riesgos derivados de la alteración de genes de susceptibilidad.
Par de bases - Base pair: (sinónimo: bp)
Dos bases nitrogenadas unidas mediante enlaces débiles en el ADN de cadena doble; el emparejamiento específico de estas bases (adenina con timina y guanina con citosina) facilita la replicación exacta del ADN; la cuantificación de los pares de bases (p. ej. 8 bp) se refiere a la longitud física de una secuencia de nucleótidos.
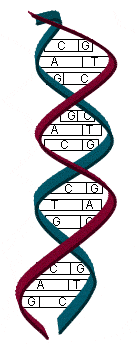
ADN de cadena doble que muestra el emparejamiento específico de los pares de bases G y C, A y T .
Parentesco - Kindred
Relación entre los miembros de una familia; término que se utiliza frecuentemente en los estudios de ligamiento para referirse a las familias numerosas.
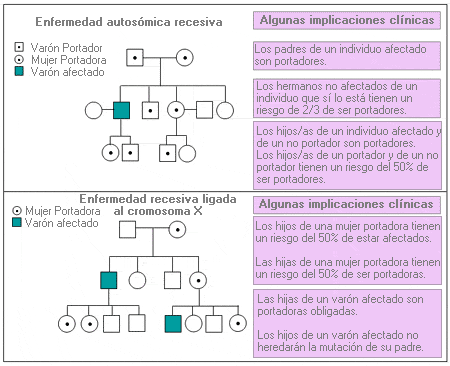
Pariente de primer grado - First-degree relative
Cualquier individuo que esté separado por una meiosis de uno de los miembros de su familia (es decir, padre/madre, hermano/a, hijo/a).
Los parientes de primer grado de la persona señalada con la flecha son los padres, hermanos e hijos:
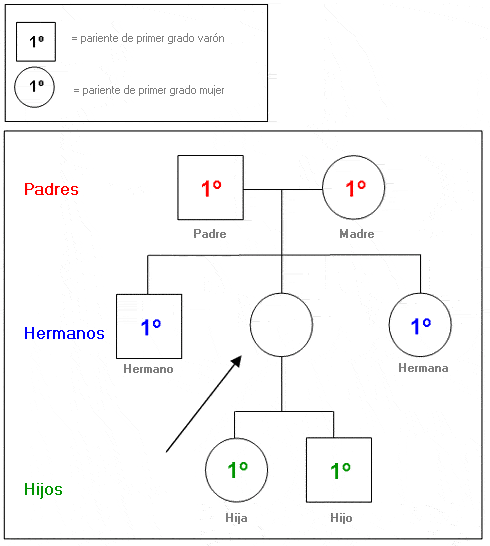
Algunas implicaciones clínicas:
Un individuo comparte la mitad de sus genes con cada uno de sus padres y cada uno de sus hijos.
Un individuo comparte, por término medio, la mitad de sus genes con cada uno de sus hermanos.
Pariente de segundo grado - Second-degree relative
Cualquier individuo que esté separado por dos meiosis de uno de los miembros de su familia; familiar con el que un individuo comparte la cuarta parte de sus genes (es decir, abuelos, nietos, tío, tía, sobrino, sobrina, hermanastros).
Paternidad alternativa - Alternate paternity (sinónimos: falsa paternidad, no paternidad):
Situación en la que el supuesto padre de un determinado individuo no es el padre biológico.
Patrón de herencia mendeliano - Mendelian inheritance pattern
Característicos de organismos que se reproducen sexualmente. Gregor Mendel describió dos tipos de “factores” (genes) conforme a su expresión sobre el fenotipo: los dominantes y los recesivos. Si además tenemos en cuenta que los individuos de sexo femenino tienen dos cromosomas X, y los del sexo masculino una X y una Y, los modos de transmisión de los caracteres de una mutación, son un total de cuatro.
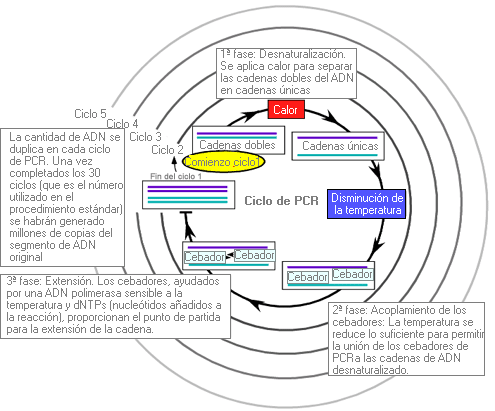
PCR - PCR (Polymerase chain reaction) (sinónimo: reacción en cadena de la polimerasa)
Procedimiento que genera millones de copias de un segmento corto de ADN mediante ciclos repetidos de: 1) desnaturalización del ADN, 2) acoplamiento de los oligonucleótidos y 3) extensión mediante la acción de la ADN polimerasa. La PCR es un procedimiento muy común en los estudios de genética molecular y se puede utilizar para: 1) generar una cantidad suficiente de ADN para realizar determinadas pruebas (p. ej. análisis de la secuencia, rastreo de mutaciones); o 2) puede ser una prueba diagnóstica en sí misma (por ejemplo amplificación específica del alelo, cuantificación del número de repeticiones de trinucleótidos).
PCR cuantitativa - Quantitative PCR (sinónimos: PCR cuantitativa en tiempo real)
Uso de PCR para determinar la cantidad de ADN o ARN en una muestra; se utiliza frecuentemente para detectar mutaciones por deleción y mutaciones por duplicación.
Anteriormente, la PCR cuantitativa determinaba la cantidad de producto (ADN o ARN) tras completar un número específico de ciclos; sin embargo, este procedimiento no es del todo fiable o preciso y se ha sustituido en gran parte por la PCR cuantitativa “en tiempo real” (denominada también “cinética”), en la que se determina la cantidad del producto (ADN o ARN) tras completar cada ciclo de PCR.
PCR cuantitativa en tiempo real
I. Se amplifica la región de ADN o ARN de interés en presencia de una sonda fluorescente; la intensidad de la señal fluorescente generada aumenta en proporción a la cantidad de producto resultante en PCR.
II. La intensidad de la señal fluorescente, que se mide al completar cada ciclo de PCR, se representa en una “gráfica de amplificación” para determinar el CT (del inglés cycle threshold), que se define como el punto en el que se detecta por primera vez la señal fluorescente, es decir, el producto de la PCR.
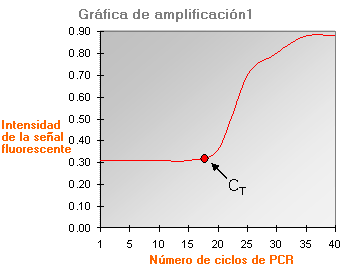
III. Se compara el valor CT frente a una curva estándar (basándose en los valores CT de muestras control y sus respectivas cantidades iniciales de ADN o ARN conocidas) para determinar la cantidad de ADN o ARN en la muestra del paciente. La presencia de una mutación por deleción o duplicación se puede deducir comparando la cantidad de ADN o ARN en la muestra del paciente con la cantidad en una muestra control. Por ejemplo, si la cantidad de ADN o ARN en la muestra del paciente fuese ~50% respecto a la del control normal, esto indicaría una mutación por deleción; si la cantidad de ADN o ARN en la muestra fuese ~150% respecto a la del control normal, indicaría una mutación por duplicación.
1Adaptado de PennState College of Medicine, Quantitative Real-Time PCR
Algunas implicaciones clínicas
Al igual que la PCR, la PCR cuantitativa se ha convertido en una técnica estándar en todos los laboratorios clínicos de genética molecular.
Los análisis para detectar mutaciones por deleción hemicigótica (p. ej. deleciones en DMD en varones afectados por distrofia muscular de Duchenne) se pueden realizar con técnicas de PCR estándar. Sin embargo, se debe realizar PCR cuantitativa, en lugar de estándar, para detectar mutaciones por deleción heterocigótica (p. ej. deleciones heterocigóticas en (1) individuos afectados por un trastorno causado por una deleción en un gen contiguo autosómico, como, por ejemplo, el síndrome de Williams, (2) portadores de un trastorno autosómico recesivo, como la atrofia muscular espinal y (3) mujeres portadoras de un trastorno ligado al cromosoma X, por ejemplo la distrofia muscular de Duchenne).
Pedigrí - Pedigree
Véase Árbol genealógico.
Penetrancia - Penetrance
Proporción de individuos que presentan una mutación causante de una patología determinada y muestran síntomas clínicos de esa patología. La mayoría de las veces este término se refiere a las patologías con herencia autosómica dominante.

¿Cuál es la diferencia entre penetrancia y expresividad variable?
La penetrancia es la proporción de individuos con una mutación patológica que presentan manifestaciones clínicas de la patología asociada a esa mutación. La expresividad variable se refiere al rango o espectro de manifestaciones clínicas observadas en individuos que presentan una patología determinada. El término expresividad variable se aplica a enfermedades con cualquier patrón de herencia.
¿De qué forma influye la expresividad variable en la penetrancia?
La penetrancia depende de las manifestaciones de enfermedad que se utilizan para determinar si un individuo está afectado. Por ejemplo, se puede suponer que los individuos con mínimas manifestaciones de una patología autosómica dominante no están afectados, por lo que la penetrancia reducida en este caso sería tan solo aparente. Consideremos lo siguiente:
Se sabe que una paciente presenta una mutación en FBN1, causante del síndrome de Marfan, porque tanto su padre como su hijo están afectados. Los resultados de la exploración física de esta paciente son normales, por lo que se deduce que la paciente no presenta manifestaciones del síndrome de Marfan, esto es una falta de penetrancia. Sin embargo, el ecocardiograma revela dilatación aórtica, que es una característica del síndrome de Marfan. La paciente realmente tiene manifestaciones del síndrome. Los resultados basados en la exploración física y en el ecocardiograma revelarán una penetrancia más alta del síndrome de Marfan que los estudios basados sólo en la exploración física.
Péptido - Peptide
Molécula formada por aminoácidos unidos por enlaces peptídicos (enlaces covalentes entre el grupo amino [–NH2] de un aminoácido y el grupo carboxilo [–COOH] de otro aminoácido). Una serie de aminoácidos unidos también se denomina polipéptido. Las proteínas están formadas por uno o varios polipéptidos.
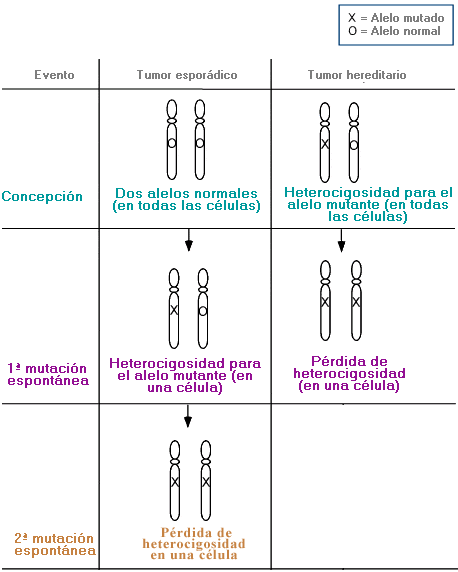
Pérdida de heterocigosidad - Loss of heterozygosity (LOH, de sus siglas en inglés)
Cuando en un locus determinado, que es heterocigoto para un alelo mutante deletéreo y un alelo normal, se produce una deleción, u otro evento mutacional, en el alelo normal que convierte a la célula bien en hemicigótica (es decir, con un único alelo deletéreo), bien en homocigótica para el alelo deletéreo.
La LOH es el mecanismo en el que se basa el desarrollo de algunos tipos de cáncer en los que una sola mutación en uno de los homólogos de un gen supresor tumoral no es suficiente para iniciar el crecimiento del tumor. Sin embargo, la deleción o inactivación del segundo alelo en el cromosoma homólogo, da lugar a un crecimiento celular incontrolado.
La LOH puede ser el fundamento del desarrollo del cáncer en tumores esporádicos y hereditarios. En los tumores esporádicos, los dos alelos son normales en el momento de la concepción, mientras que en los tumores hereditarios hay un alelo mutante en el momento de la concepción.
PGD - Preimplantation Genetic Diagnosis
Véase Diagnóstico preimplantacional.
Pirimidina - Pyrimidine
Es un compuesto orgánico, similar al benceno, y a la piridina pero con dos átomos de nitrógeno que sustituyen al carbono en las posiciones 1 y 3. La pirimidina tiene tres derivados que forman parte de los ácidos nucleicos: la timina, la citosina y el uracilo; las tres tienen un grupo carbonilo (C=O) en el carbono 2; las dos primeras forman parte del ADN donde se aparean con sus purinas complementarias, mientras que la última está presente solo en el ARN. En el ADN, adenina (A) y guanina (G) se emparejan con timina (T) y citosina (C), respectivamente, y en el ARN adenina se une a uracilo (U) y la guanina a citosina.
Pirrolisina - Pyrrolysine
La pirrolisina (abreviaturas Pyl o O) es un aminoácido natural, derivado de la lisina, codificado en el genoma de algunas arqueas metanógenas y una especie de bacteria (Desulfitobacterium hafniense). Forma parte de un código genético inusual, propio de estos organismos, y se considera el aminoácido proteinogénico número 22. Se encuentra codificada en el ARNm por el codón UAG.
Plásmido - Plasmid
Molécula pequeña, a menudo circular, de ADN extracromosómico, cuya replicación es independiente de la del ADN cromosómico. Son comunes en bacterias y levaduras. Por lo general contienen un reducido número de genes que suele conferir ciertas ventajas al hospedador, por ejemplo, resistencia a un determinado antibiótico.
Los plásmidos se usan en ingeniería genética aprovechando su capacidad para reproducirse de manera independiente del ADN cromosómico.
Pleiotropia - Pleiotropy
Múltiples efectos fenotípicos o caracteres distintos causados por la acción de un solo gen o par de genes. El término se utiliza de manera particular cuando no se sospecha una relación entre los rasgos o caracteres fenotípicos.
Ploidía - Ploidy
Número de grupos o juegos completos de cromosomas que contiene una determinada célula. Las células somáticas humanas son diploides, dos grupos completos de cromosomas, cada uno, con 23 cromosomas, procedente de un progenitor. En cambio, los gametos, óvulo y espermatozoide, son haploides, cada uno con 23 cromosomas.
Poligénico - Polygenic
Rasgo o patología causado por las contribuciones aditivas de mutaciones producidas en múltiples genes en diferentes loci.
Polimorfismo - Polymorphism
Polimorfismo de Conformación de Cadena Simple o SSCP -
Polimorfismo de longitud de fragmentos de restricción - Restriction fragment length polymorphism (sinónimo: RFPL)
Variación natural (polimórfica) de una secuencia de ADN entre los individuos. Se visualiza porque se suprimen o se crean nuevos sitios de restricción (corte) en el ADN, que cuando es digerido por una endonucleasa da lugar a fragmentos de ADN de diferente longitud.
Polimorfismo de nucleótido único - Single Nucleotide Polymorphism o SNP
Variación en la secuencia de ADN que implica una sola base: adenina (A), timina (T), citosina (C) o guanina (G). Son los polimorfismos genéticos más frecuentes y se considera que determinan gran parte de la variabilidad genética entre individuos. Se conocen varios millones de SNP repartidos por todos los cromosomas humanos, aproximadamente 1 cada 300-100 pares de bases. La frecuencia de un SNP en población general se sitúa por encima del 1%.
Diversas técnicas de análisis masivo del genoma se fundamentan en el estudio de miles o millones de SNP que cubren la práctica totalidad del genoma (Genome Wide Association Studies, o GWAS), para buscar la asociación de algunos de ellos con enfermedades concretas, utilizando para ello grandes números tanto de individuos enfermos, como de individuos sanos.
Polimorfismos de conformación de cadenas sencillas - Single-stranded conformational polymorphism (SSCP)
Técnica para el rastreo de mutaciones que identifica segmentos de ADN de cadena sencilla que muestran un patrón de migración anormal en la electroforesis en gel.
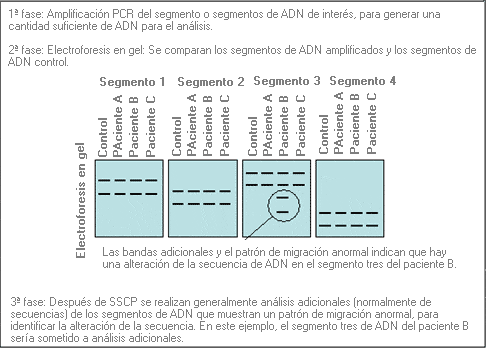
Algunas implicaciones clínicas
La técnica SSCP se puede utilizar cuando las mutaciones están distribuidas por todo el gen.
El SSCP puede comprender el análisis de todo el gen o sólo de regiones específicas.
Poliploidía - Polyploidy
Incremento del número de cromosomas de una célula que es múltiplo del número haploide (23). La triploidía se refiere a tres grupos o juegos completos de cromosomas en una sola célula (en los seres humanos, la triplodía se corresponde con la presencia de un total de 69, 3x23, cromosomas en cada célula); la tetraploidía se refiere a cuatro grupos de cromosomas en una sola célula (en los seres humanos serían 92, 4x23, cromosomas por célula).
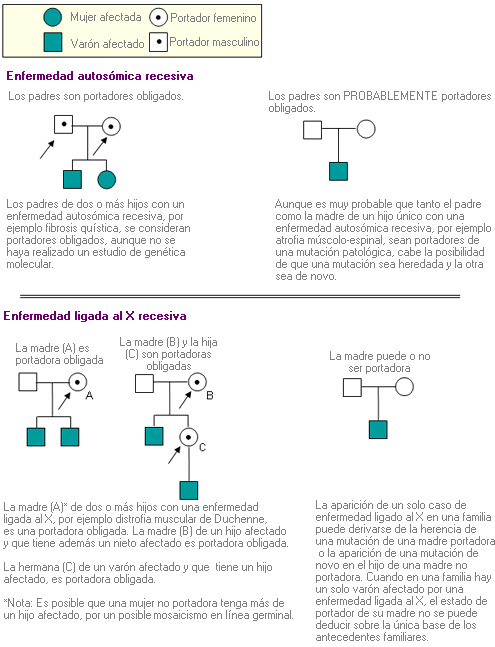
Portador - Carrier
Portador obligado - Obligate carrier (sinónimo: heterocigoto obligado)
Portador sintomático - Manifesting carrier
Individuo que en un locus determinado presenta un alelo recesivo patológico en un cromosoma y un alelo normal en el otro cromosoma y que manifiesta algunos síntomas de la enfermedad. Este término se usa con frecuencia para portadoras de una mutación recesiva ligada al cromosoma X que manifiestan signos de la enfermedad, aunque su fenotipo suele ser menos severo que el de los varones con la misma mutación.
Aunque con algunas excepciones, los portadores de una mutación patológica recesiva por regla general no manifiestan síntomas clínicos de la enfermedad. Sin embargo, los portadores de mutaciones recesivas ligadas al cromosoma X presentan ocasionalmente algunas manifestaciones clínicas de la enfermedad, por lo que se les denomina portadores sintomáticos. Se supone que el mecanismo subyacente en los portadores sintomáticos es una inactivación sesgada o no aleatoria del cromosoma X, de tal manera que en la mayoría de las células el cromosoma X activo es el que tiene la mutación.
Ejemplos de enfermedades ligadas al cromosoma X en las que son frecuentes las portadoras sintomáticas:
Hemofilia A: Tiempo de coagulación prolongado, hemorragia excesiva durante intervenciones quirúrgicas menores y menstruaciones abundantes.
Distrofia muscular de Duchenne: Debilidad muscular, hipertrofia de pantorrillas leve y/o miocardiopatía dilatada.
Síndrome X frágil (mutación con penetrancia completa): Retraso mental.
Potenciador - Enhancer
Pequeña región del ADN eucariota que puede aumentar los niveles de transcripción de un gen o un grupo de genes mediante la unión a proteínas (factores de transcripción). Las regiones enhancer no tienen por qué estar localizadas cerca de los genes sobre los que actúa, ni siquiera en el mismo cromosoma.
Predisposición genética - Genetic predisposition (sinónimo: susceptibilidad genética)
Mayor propensión de un individuo a padecer una enfermedad determinada debido a la presencia de una o más mutaciones génicas asociadas con un aumento del riesgo de que se manifieste la enfermedad y/o debido a la existencia de antecedentes familiares que también indican una mayor probabilidad de que se desarrolle la enfermedad.
Prevalencia - Prevalence
Frecuencia de una enfermedad en la población, contabilizando tanto los casos antiguos como los de nueva aparición.
Primer - Primer
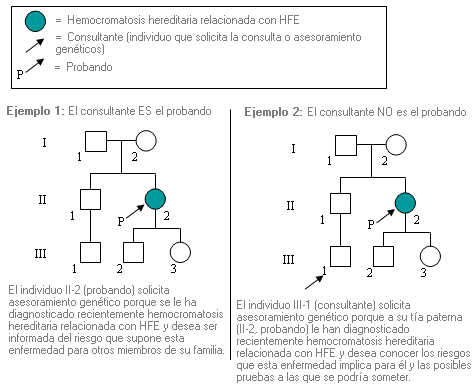
Probando - Proband (sinónimos: caso índice, propósito)
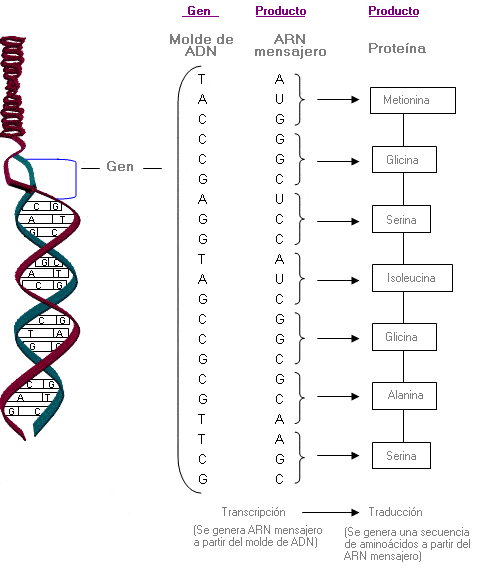
Producto génico - Gene product
Profase - Prophase
Es la primera fase de la mitosis y de la meiosis. En ella se produce la condensación de todo el material genético (ADN)-que normalmente existe en forma de cromatina condensada dentro de una estructura altamente ordenada llamada cromosoma- y el desarrollo bipolar del huso acromático. Los dos pares de centríolos migran hacia extremos opuestos de la célula, la membrana nuclear se disuelve al final de la profase y los cromosomas se anclan en los microtúbulos del huso gracias al cinetocoro que se encuentra en el centrómero.
Prolina - Proline
La prolina (Pro, P) o prolalina es uno de los aminoácidos que forman las proteínas de los seres vivos. En el ARN mensajero está codificada como CCU, CCC, CCA o CCG. Se puede formar directamente a partir de la cadena pentacarbonada del ácido glutámico, y por tanto no es un aminoácido esencial.
Prometafase - Prometaphase
Es el proceso o fase posterior a la profase y anterior a la metafase en la mitosis celular. Según cómo interactúen entre sí durante esta fase la membrana nuclear y el huso mitótico de microtúbulos, la mitosis puede ser abierta (la membrana nuclear se separa, y los microtúbulos atraviesan el espacio nuclear. Ocurre en una pequeña parte de los organismos multicelulares) o cerrada (el huso mitótico se forma dentro del núcleo o sus microtúbulos pueden penetrar hasta los cromosomas a través de la membrana nuclear sin que esta se rompa). Ocurre en los hongos y algunos protistas.
Promotor - Promoter
Secuencia de ADN necesaria para activar un gen. La transcripción del gen se inicia en el promotor. Generalmente se encuentran cerca del comienzo de un gen, el promotor tiene un sitio de unión para la enzima ARN polimerasa encargada de la síntesis del ARNm.
Proteómica - Proteomics
Estudio a escala masiva de la distribución y número de proteínas codificadas por los genes, y sus interacciones.
Protooncogen - Protooncogen
Gen normal generalmente implicado en funciones celulares básicas de crecimiento y diferenciación celular, que cuando muta se convierte en oncogen, es decir promotor del proceso tumoral.
Proyecto Genoma Humano - Human Genome Project
El Proyecto Genoma Humano fue una investigación internacional que descifró la totalidad de la secuencia del genoma humano, mapeando y secuenciando todos sus genes. Oficialmente se inició en 1990 como un programa a quince años vista, con el que se pretendía registrar los 80.000-100.000 genes humanos (finalmente se comprobó que eran alrededor de 20.000-25.000). Los rápidos avances tecnológicos aceleraron los tiempos de modo que los resultados iniciales se publicaron en febrero de 2001, y la secuenciación se completó en el 2003, aunque no se conocen en detalle las funciones de todos los elementos del genoma.
Los objetivos concretos del Proyecto fueron:
• Identificar los aproximadamente 100.000 genes humanos en el ADN.
• Determinar la secuencia de 3 billones de bases químicas que conforman el ADN.
• Guardar la información en bases de datos.
• Desarrollar de modo rápido y eficiente tecnologías de secuenciación.
• Desarrollar herramientas para análisis de datos.
• Dirimir las cuestiones éticas, legales y sociales que se derivan del proyecto.
Prueba de parentesco - Parentage testing (sinónimos: prueba de maternidad, prueba de paternidad)
Proceso mediante el cual se comparan secuencias de ADN de un niño y un adulto determinados para calcular la probabilidad de que los dos individuos estén emparentados. Mediante los análisis de ADN se puede descartar con seguridad, pero no confirmar absolutamente que un individuo es el padre o la madre biológicos.
Pseudoautosómico, región pseudoautosómica - Pseudoautosomal, Pseudoautosomal región
Regiones distales de los brazos cortos de los cromosomas sexuales X e Y que recombinan entre sí durante la meiosis del varón.
Pseudogen - Pseudogene
Copia de un gen que carece normalmente de intrones y de otras secuencias de ADN esenciales para su función. Los pseudogenes, aunque son genéticamente similares al gen funcional original, no se expresan y frecuentemente contienen numerosas mutaciones.
Pseudogenes:
Consisten en secuencias de ADN muy similares a los genes conocidos, pero no son funcionales.
Se encuentran dispersos en el genoma.
Se cree que son subproductos de la evolución, que representan genes rudimentarios que una vez fueron funcionales.
Se supone que han perdido su función original por la introducción de mutaciones en las secuencias codificadoras o reguladoras.
 Supuestos mecanismos por los que se forman los pseudogenes:
Supuestos mecanismos por los que se forman los pseudogenes:
Duplicación de un gen funcional seguida de la introducción de numerosas mutaciones que finalmente dan lugar a su inactivación.
Retrotransposición – proceso mediante el cual se generan una o más copias de ADN a partir del transcripto de ARNm del gen y se vuelven a insertar en el genoma en cualquier localización, no necesariamente cerca del sitio de su gen progenitor. Puesto que se generan a partir de ARNm procesado, estos pseudogenes carecen de intrones.
Puntos calientes de mutación - Hotspot mutation region
Secuencias de adn muy susceptibles a mutaciones debido a una inestabilidad inherente, tendencia hacia un entrecruzamiento desigual o predisposición química a sustituciones de nucleótidos simples; región en la que se observan mutaciones con más frecuencia de la habitual.
Purina - Purine
Es una base nitrogenada, un compuesto orgánico heterocíclico aromático. La estructura de la purina está compuesta por dos anillos fusionados, uno de seis átomos y el otro de cinco. Dos de las bases de los ácidos nucleicos, adenina y guanina, son derivados de una purina. En el ADN, estas bases se unen con sus pirimidinas complementarias, la timina y la citosina, a través de enlaces de hidrógeno.