- Todos
- A
- B
- C
- D
- E
- F
- G
- H
- I
- J
- K
- L
- M
- N
- Ñ
- O
- P
- Q
- R
- S
- T
- U
- V
- W
- X
- Y
- Z
A
Análisis basados en ADN - DNA-based analysis
Pruebas que implican el estudio de ADN mediante un análisis de ligamiento, secuenciación o cualquiera de los diferentes métodos utilizados para la detección de mutaciones.
Análisis de dosis - Dosage analysis
Método para determinar la cantidad de diversos componentes, incluyendo ADN, ARN y proteínas, por comparación con un estándar conocido; se puede utilizar para determinar el número de copias de una secuencia de ADN (es decir, para investigar posibles mutaciones por duplicación y deleción), mediante la comparación visual de la intensidad de las bandas o la cuantificación numérica con densitometría. Si un gen presenta copias adicionales, la intensidad es de más del 100% en el gel o película; mientras que si se ha perdido una copia del gen, la intensidad es de aproximadamente un 50%.
Análisis de fragmentos de restricción de longitud polimórfica - Restriction fragment length polymorphism analysis (sinónimo: análisis RFLP, estudios RFLP)
Estudio de los fragmentos de ADN de tamaño previsible originados por la digestión (corte) de una cadena de ADN por una enzima de restricción específica. Las alteraciones (mutaciones o polimorfismos) producidas en la secuencia de ADN que destruyen o crean nuevos sitios de corte en el ADN, modifican el tamaño (y el número) de los fragmentos de ADN resultantes de la digestión por una enzima de restricción específica.

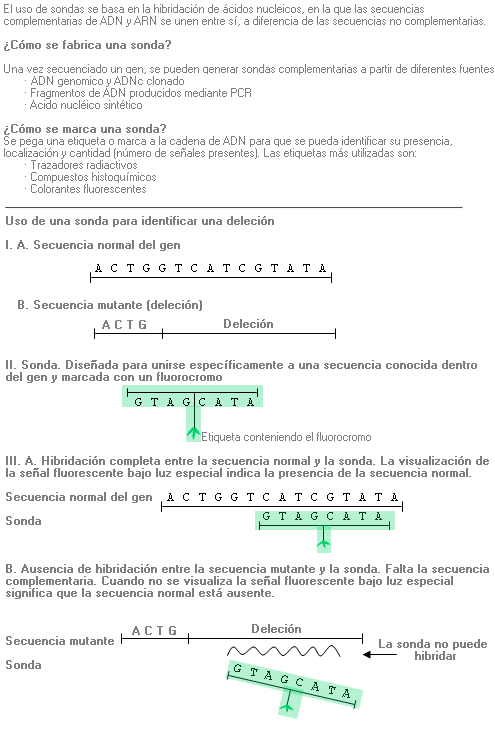
Análisis de secuencias - Sequence analysis (sinónimo: secuenciación génica, secuenciación)
Proceso mediante el cual se determina la secuencia de nucleótidos en un segmento de ADN.
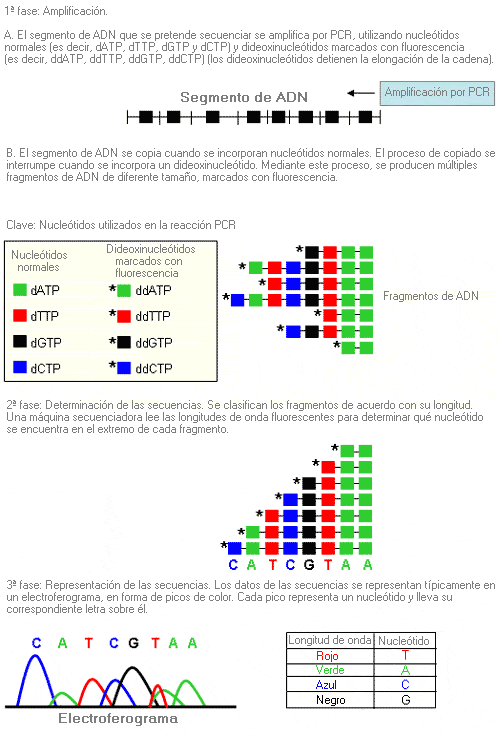
Se puede secuenciar un gen completo, pero la mayoría de las veces se secuencian sólo regiones específicas del gen que muy probablemente contendrán mutaciones (p. ej. los exones y los límites entre los intrones y los exones).
Al secuenciar un segmento de ADN, se identifican la mayoría de las variaciones producidas con respecto a la secuencia normal (por el contrario, el análisis mutacional identifica sólo mutaciones específicas en un segmento determinado de ADN).
Tipos de alteraciones de las secuencias que se pueden detectar:
Alteraciones de secuencia descritas en la literatura como patógenas
Alteraciones de secuencias previsiblemente patógenas, pero no descritas en la literatura
Alteraciones de secuencias de significación clínica imprevisible
Alteraciones de secuencias previsiblemente benignas, pero no descritas en la literatura
Alteraciones de secuencias benignas descritas en la literatura
Posibilidades en caso de que no se detecten alteraciones de las secuencias:
El paciente no presenta una mutación en el gen analizado (p. ej. hay una alteración de la secuencia en otro gen, en un locus diferente)
El paciente presenta una alteración de la secuencia que no se puede detectar mediante el análisis de secuencias (p. ej. deleción de tamaño grande)
El paciente presenta una alteración de la secuencia en una región del gen (p. ej. una región intrónica o reguladora) que no se ha detectado en el análisis de laboratorio
1Adaptado de ACGM Recommendations for Standards for Interpretation of Sequence Variations (2000)
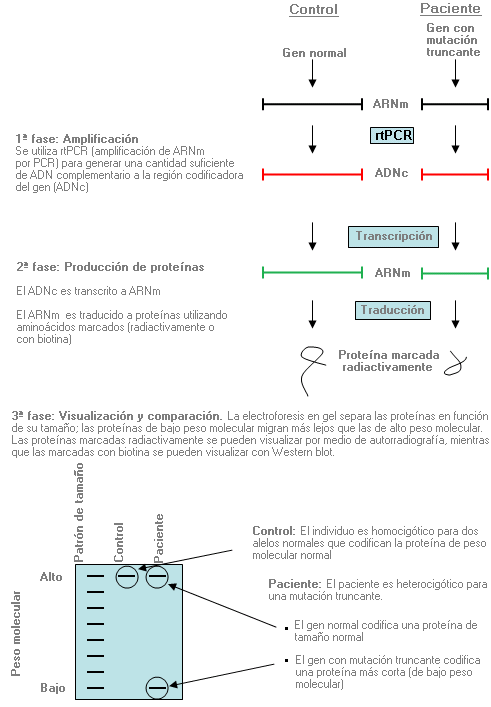
Análisis de truncación de proteínas - Protein truncation testing (sinónimo: PTT)
Método utilizado para identificar las proteínas que se acortan (truncan) a consecuencia de mutaciones que causan específicamente la terminación prematura del proceso de traducción del ARNm.
Análisis directo del ADN - Direct DNA analysis
La utilización de técnicas de secuenciación, análisis mutacional, rastreo de mutaciones o cualquier otro medio de estudio molecular genético, para detectar variantes en un gen que pueden estar asociadas a trastornos genéticos específicos. El análisis directo del adn es posible sólo cuando se conoce el gen o genes o la región genómica, asociados con la enfermedad.
Las técnicas utilizadas para el análisis de ADN directo pueden ser las siguientes:
Análisis de secuencias
Rastreo de mutaciones (p. ej. CSGE, DHPLC, SSCP)
Análisis mutacional (p. ej. PCR, Southern blot, análisis de repeticiones de trinucleótidos, FISH, análisis de truncación de proteínas (PTT), estudios de heterocigosidad, análisis RFLP, ASO)
El análisis de ADN directo contrasta con el análisis de ligamiento (que anteriormente se denominaba análisis de ADN indirecto)
Algunas implicaciones clínicas
El análisis de ADN directo sólo es posible cuando se conoce el gen (genes) o la región genómica asociados con una enfermedad
El análisis de ligamiento se puede realizar cuando no se conoce el gen o los genes asociados con una enfermedad
Análisis mutacional específico - Targeted mutation analysis (sinónimo: análisis mutacional dirigido)
Pruebas para detectar la presencia de una mutación específica (p. ej. Glu6Val en la Anemia de Células Falciformes), un tipo de mutación específica (p. ej. la expansión de trinucleótidos repetidos asociada a la Ataxia Espinocerebelosa tipo 1, deleciones asociadas a la Distrofia Muscular de Duchenne) o un conjunto de mutaciones (p. ej. un grupo de mutaciones en la Fibrosis Quística). Difiere de la secuenciación génica completa o el rastreo de mutaciones, en que éstos detectan la mayoría de las mutaciones en la región investigada.
El análisis mutacional se utiliza en las dos situaciones siguientes:
I. Estudio de enfermedades en las que:
La mayoría o todos los individuos afectados presentan la misma mutación (p. ej. Anemia de Células Falciformes).
La mayoría o todos los individuos afectados presentan el mismo tipo de mutación (p. ej. expansión de repeticiones de trinucleótidos).
Un grupo de mutaciones son las más comunes y explican un elevado porcentaje de los casos (véase Tabla).

II. Estudio de los miembros a riesgo de una familia tras haber identificado la mutación patológica específica de la familia en un pariente afectado. Por ejemplo, si en el análisis de la secuencia se identifica una mutación patológica en un miembro afectado de la familia, todos los familiares a riesgo deben ser sometidos sólo a un análisis mutacional dirigido a detectar esa mutación específica de la familia.
Algunas implicaciones clínicas
El análisis mutacional no es generalmente el primer estudio de genética molecular que se utiliza para la identificación de mutaciones en enfermedades que se sabe están asociadas con una proporción alta de mutaciones raras (por ejemplo, específicas de una familia).
Si en un individuo afectado no se puede detectar una mutación en el análisis mutacional, puede que sea necesario realizar un análisis de la secuencia o un rastreo de otras mutaciones para identificarla.
Análisis mutacional individualizado - Custom mutation analysis
Estudios de Genética molecular desarrollados especialmente en un laboratorio clínico en función de las necesidades, para detectar una mutación específica que predispone a una enfermedad que no se puede identificar por otros medios clínicos. Las situaciones en las que puede utilizarse un análisis mutacional individualizado son las siguientes: a) estudios de miembros de una familia que tienen riesgo de desarrollar una enfermedad específica, al haberse identificado una mutación patológica en un miembro afectado de esa misma familia; b) estudios prenatales en familias en las que se ha identificado una mutación patológica en un miembro afectado.
B
C
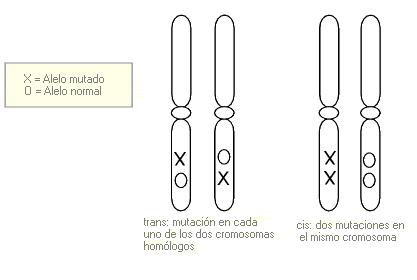
Configuración en cis - Cis configuration (sinónimos: cis, acoplamiento)
Término que indica que un individuo que es heterocigoto en dos loci adyacentes presenta esas dos mutaciones en el mismo cromosoma, en vez de una en cada cromosoma homólogo (configuración en trans).
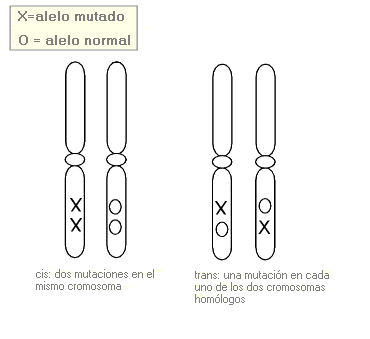
Configuración en trans - Trans configuration
Conversión génica - Gene conversion
Transferencia de secuencias de ADN entre dos genes muy similares, que se produce la mayoría de las veces por un entrecruzamiento desigual durante la meiosis. Puede ser un mecanismo que da lugar a una mutación si la transferencia de material altera la secuencia codificadora del gen o si el mismo material transferido contiene una o más mutaciones.
Puesto que los pseudogenes guardan una gran similitud con el gen activo, puede producirse un emparejamiento erróneo. La recombinación entre el pseudogen y el gen activo podría transferir una mutación desde el pseudogen al gen activo.
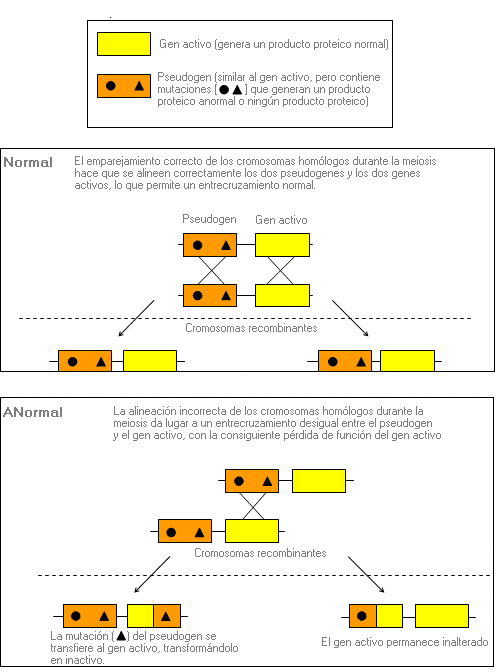
Correlación genotipo - fenotipo - Genotype - Phenotype correlation
Asociación entre determinada mutación o mutaciones (genotipo) y el fenotipo resultante.
Las posibles conclusiones sobre las correlaciones genotipo-fenotipo son las siguientes:
La estrecha correlación entre el genotipo y el fenotipo permite predecir el fenotipo de forma fiable, basándose en el genotipo.
La severidad y las manifestaciones clínicas específicas del fenotipo no se pueden predecir con seguridad debido a la amplia variabilidad de presentaciones fenotípicas entre los individuos que tienen el mismo genotipo.
No se han realizado estudios de correlación genotipo-fenotipo o bien éstos no son viables en la práctica debido al elevado número de mutaciones diferentes posibles.
A la inversa, la pregunta para la correlación fenotipo-genotipo es la siguiente: dado un fenotipo determinado ¿cuál es el genotipo más probable? En este caso, la evaluación del fenotipo sirve de orientación al médico/laboratorio respecto a las técnicas más apropiadas para la detección de mutaciones.
Ejemplo: Síndrome de deleción 22q11.2
Manifestaciones clínicas: Defectos cardíacos (en particular conotruncales), ausencia o función anormal del timo y/o las glándulas paratiroideas, paladar fisurado, rasgos faciales característicos, trastornos del habla y retraso del desarrollo.
Correlación genotipo-fenotipo: BAJA Un feto en el que se ha identificado la deleción 22q11.2 podría presentar todas las manifestaciones clínicas asociadas, sólo algunas de ellas o sólo trastornos del habla. No se puede predecir el fenotipo basándose en el genotipo.
Correlación fenotipo-genotipo: ALTA Niño nacido con defecto cardíaco conotruncal y ausencia de timo. La probabilidad de detectar una deleción en 22q11.2 es aproximadamente del 95%. El genotipo se puede predecir con seguridad basándose en el fenotipo.
Cribado - Screening
Pruebas destinadas a identificar a los individuos en una población determinada que presentan un riesgo más alto de tener o desarrollar un trastorno específico o de presentar una mutación génica por la cual desarrollarían un trastorno específico.
En el diagrama siguiente se representan las pruebas de detección prenatal del síndrome de Down, defectos del tubo neural abiertos y la trisomía 18 en la población embarazada, basadas en el análisis de marcadores séricos maternos.
Dependiendo de la prueba realizada, es posible que no se detecten alteraciones en algunos individuos afectados (resultado falso negativo), o que se identifiquen en individuos que no están afectados (resultado falso positivo).
1000 mujeres embarazadas (en total) sometidas al análisis de marcadores séricos maternos para la detección de posibles trastornos.
*Tasa de detección positiva, la sensibilidad y la especificidad varían en función del punto de corte de riesgo positivo de la prueba de detección y del número de análisis evaluados y determinados por cada laboratorio. 
D
Densitometría - Densitometry
Método para identificar la dosis o la expresión génica mediante la medición de la absorción de la luz en una autorradiografía (película) de una banda (o mancha) que representa el ADN, el ARN o una muestra de proteínas. Es una técnica útil para detectar mutaciones por duplicación y mutaciones por deleción heterocigótica.
DGGE - (Denaturing gradient gel electrophoresis) (sinónimo: Electroforesis en gel con gradiente desnaturalizante)
Técnica de electroforesis para muestras de adn de cadena doble que utiliza un gel en el que se crea un gradiente desnaturalizante mediante una sustancia química, por ejemplo, urea. Se emplea para la identificación de mutaciones que afectan al patrón de migración del adn en esas condiciones de electroforesis. La presencia de la mutación se detecta mediante comparación del patrón electroforético (punto de desnaturalización) de la muestra problema con la secuencia salvaje u original.
DHPLC (Denaturing high-performance liquid chromatography) -
Cromatografía líquida desnaturalizante de alto rendimiento. En genética es un método utilizado para la detección de mutaciones de uno o pocos nucleótidos. La base de la técnica es la diferencia de migración que presentan fragmentos homodúplex (secuencia doble de ADN que presenta un 100% de apareamiento entre las bases) frente a los heterodúplex (apareamiento no total) durante su migración por un gradiente en una columna.
Dominante ligado al cromosoma X - X-linked dominant
Describe un rasgo o trastorno de manifestación dominante causado por una [GLOSARIO]mutación[FIN_GLOSARIO] en un gen del cromosoma X. El fenotipo está expresado en mujeres heterocigóticas, así como en varones hemicigóticos (que sólo tienen un cromosoma X); los varones afectados suelen presentar un fenotipo más severo que las mujeres afectadas.

Probabilidad de transmisión de una mutación de un gen dominante ligado al cromosoma X.

E
Electroforesis en gel con gradiente desnaturalizante - Denaturing gradient gel electrophoresis (sinónimo: DGGE)
Técnica de electroforesis para muestras de ADN de cadena doble que utiliza un gel en el que se crea un gradiente desnaturalizante mediante una sustancia química, por ejemplo, urea. Se emplea para la identificación de mutaciones que afectan al patrón de migración del ADN en esas condiciones de electroforesis. La presencia de la mutación se detecta mediante comparación del patrón electroforético (punto de desnaturalización) de la muestra problema con la secuencia salvaje u original.
Electroforesis en gel sensible a conformación - Conformation-sensitive gel electrophoresis (sinónimo: CSGE)
Técnica electroforética que consiste en el análisis de un segmento de ADN para detectar errores de emparejamiento entre pares de bases normales y mutadas (ver explicación esquemática). Se emplea para el rastreo de mutaciones.
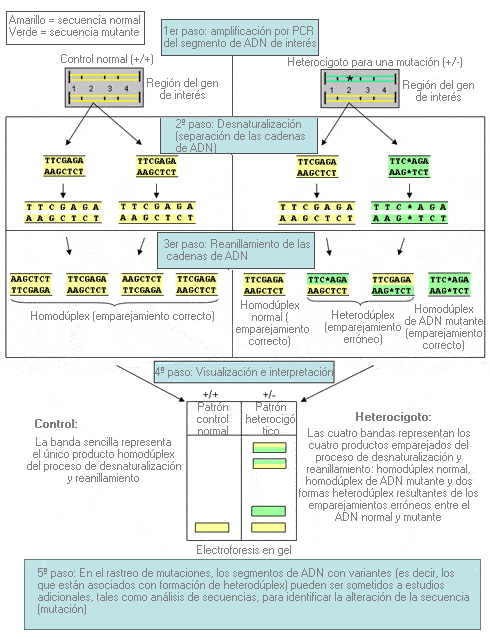
Enfoque isoeléctrico - Isoelectric focusing
Método de detección de mutaciones basado en la separación de proteínas en función del valor de pH en el que su carga neta es cero (punto isoeléctrico). Se suele utilizar conjuntamente con la técnica western blot para permitir la identificación de productos proteicos de tipo salvaje frente a mutante. La alteración de una secuencia de adn que dé lugar a una sustitución de aminoácidos puede variar el punto isoeléctrico de una proteína.
Especificidad - Specificity
Esporádico - Sporadic
La aparición casual de un trastorno o anomalía en un individuo cuyo riesgo de recurrencia no es probable en su familia.
Los trastornos esporádicos pueden ser de etiología genética o no genética.
Trastornos esporádicos de etiología genética
Disomía uniparental – El individuo afectado ha heredado los dos genes o cromosomas de un mismo par de cromosomas procedentes de uno de sus progenitores y ninguno del otro. No es probable que la aparición casual de una anomalía genética implique un riesgo de recurrencia en otros miembros de la familia.
Tumores esporádicos – Todos los tumores son de origen genético a nivel celular; sin embargo, NO todos son hereditarios. Es probable que un único tumor en un individuo de más edad esté causado por mutaciones adquiridas (no heredadas) de genes reguladores celulares, que dan lugar a una proliferación celular incontrolada.
Trastornos esporádicos de etiología no genética:
Síndrome de rubeola congénita en un niño cuya madre ha padecido rubeola durante el embarazo
Ataxia causada por alcoholismo
El término “esporádico” puede ser utilizado vagamente (e incorrectamente) para referirse a lo siguiente:
Trastornos autosómicos dominantes o ligados al cromosoma X que aparecen en un solo individuo de la familia, generalmente a consecuencia de una mutación nueva. Puesto que el riesgo de recurrencia aumenta en los hermanos del individuo afectado y es de hasta un 50% en los descendientes de este individuo, el uso del término “esporádico” no es apropiado. En este caso, el término correcto que debe utilizarse es “caso único”.
Estudios de genética molecular - Molecular genetic testing (sinónimos: análisis de ADN, análisis basados en ADN, estudios moleculares)
Pruebas que implican el estudio del ADN, mediante análisis de ligamiento, secuenciación o detección de mutaciones, utilizando uno o varios de los diferentes métodos disponibles.
Estudios de repeticiones de trinucleótidos - Trinucleotide repeat testing
Análisis genético cuyo objetivo es la cuantificación del número de repeticiones de grupos de tres nucleótidos en un segmento determinado de ADN.
Para cuantificar las repeticiones de trinucleótidos, se pueden utilizar dos tipos de análisis mutacional que se representan en el diagrama siguiente:
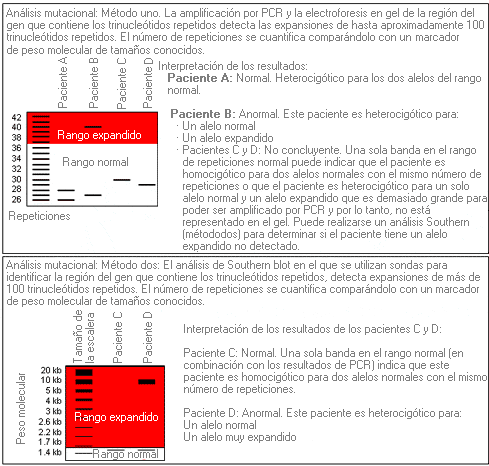
Algunas implicaciones clínicas:
El análisis Southern blot no se realiza de forma rutinaria en todos los laboratorios para clarificar la interpretación de los resultados de las bandas no concluyentes por PCR. Cuando se reciba un informe de un número de repeticiones “homocigóticas” normales, particularmente en un niño sintomático de corta edad que podría presentar una expansión grande, es recomendable que el médico confirme que se ha realizado el análisis Southern blot para evaluar la posibilidad de que haya una repetición de trinucleótidos muy expandida.
Si no está disponible el análisis Southern blot, se puede realizar un análisis mutacional basado en PCR/electroforesis en gel de los padres de un paciente con un número de repeticiones “homocigóticas” normales, para determinar si el paciente podría haber heredado de sus padres dos alelos con el mismo número de repeticiones.
Estudios prenatales individualizados - Custom prenatal testing
Pruebas de diagnóstico prenatal ofrecidas a familias en las que se han identificado mutaciones patológicas en un miembro afectado.
Evento post-cigótico - Post-zygotic event
Acontecimiento mutacional o alteración en la replicación/segregración de los cromosomas, que se produce una vez que un óvulo ha sido fertilizado por un espermatozoide y generalmente da lugar a mosaicismo (presencia de dos o más líneas celulares genéticamente distintas en el mismo organismo).
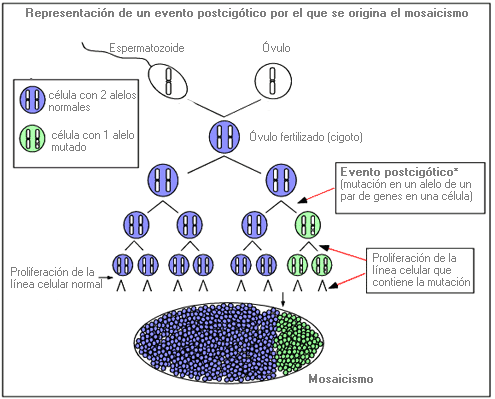
F
Fenocopia - Phenocopy
Variación en el fenotipo no hereditaria (generalmente referido a un único rasgo) que es causado por condiciones ambientales. Imita a un fenotipo producido por un genotipo específico. No es un tipo de mutación, ya que no es hereditaria.
G
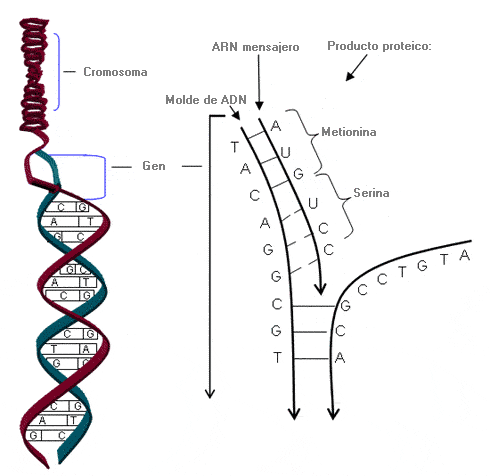
Gen - Gene
Gen supresor de tumores - Tumour suppressor gene
Gen implicado en procesos de crecimiento y/o diferenciación celular. Cuando este gen sufre una mutación que causa una pérdida o reducción en su función, la célula puede progresar a cáncer, generalmente en combinación con otros cambios genéticos.
H
Herencia mitocondrial - Mitochondrial inheritance
Las mitocondrias son organelas citoplasmáticas que producen ATP (la energía que origina la mayoría de las reacciones químicas en el organismo) y contienen su propio genoma. Las mutaciones producidas en los genes mitocondriales son responsables de diversos síndromes reconocidos y se heredan siempre de la madre, puesto que los óvulos contienen mitocondrias, mientras que los espermatozoides no.
Heterocigoto doble - Double heterozygote
Individuo que es heterocigoto para mutación en dos loci genéticos diferentes. Es decir, es portador de dos mutaciones en genes distintos.
Cuando el padre y la madre presentan una mutación en dos loci genéticos diferentes, sus hijos pueden ser heterocigotos dobles. Los loci pueden estar en el mismo cromosoma o en diferentes cromosomas, según se muestra a continuación.
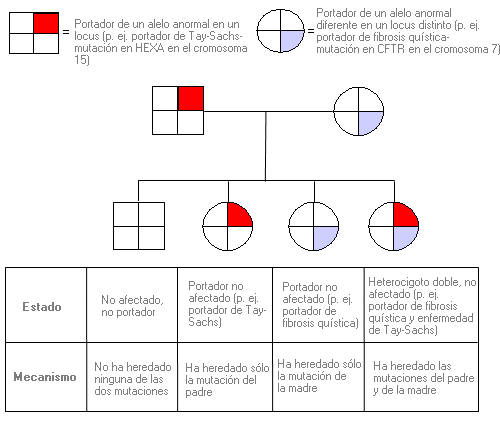
Nota: Puesto que en el ejemplo anterior se incluyen dos loci diferentes (dos alelos para cada uno), en cada hijo/a pueden darse 16 combinaciones posibles de los cuatro alelos de los padres.
Heterogeneidad alélica - Allelic heterogeneity (sinónimo: heterogeneidad molecular)
Heterogeneidad de locus - Locus heterogeneity
Situación en que las mutaciones producidas en Genes que se encuentran en diferentes loci cromosómicos dan lugar al mismo Fenotipo.
Ejemplo de heterogeneidad de locus: Retinitis pigmentosa (RP)
Fenotipo: Las anomalías de los fotorreceptores (bastoncillos y conos) de la retina dan lugar a una adaptación inadecuada a la oscuridad, causan pérdida de visión periférica y finalmente pérdida progresiva de visión.
Loci: Las mutaciones producidas en los genes en más de 35 loci diferentes dan lugar a retinitis pigmentosa no sindrómica que es clínicamnete indistinguible, por lo que no es posible deducir el gen alterado basándose sólo en el exámen ocular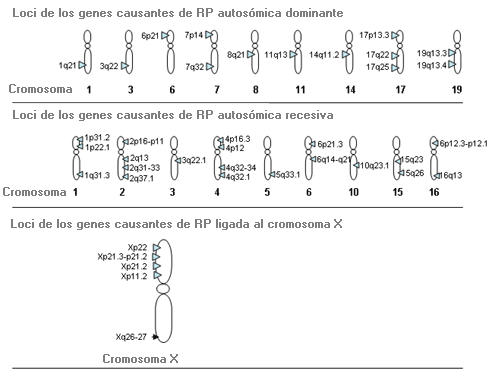
Comparar y contrastar el concepto de heterogeneidad de locus con lo siguiente:
Heterogeneidad alélica: Un solo fenotipo originado por diferentes mutaciones en el mismo gen y locus cromosómico (Nota: algunos trastornos muestran heterogeneidad de locus, otros heterogeneidad alélica y otros ambas características)
Expresividad: Variación de las manifestaciones clínicas (tipo y severidad) de una enfermedad genética entre individuos que presentan la misma alteración genética, incluso dentro de la misma familia.
(Nota: La heterogeneidad de locus implica que los individuos con diferentes genotipos tienen el mismo fenotipo; mientras que la expresividad variable implica que los individuos con el mismo genotipo presenten un fenotipo que muestra diferencias tanto en la severidad como en la edad de comienzo de sus manifestaciones).
Heterogeneidad genética - Locus heterogeneity
Situación en que las mutaciones producidas en genes distintos dan lugar al mismo fenotipo.
Fenotipo: Las anomalías de los fotorreceptores (bastoncillos y conos) de la retina dan lugar a una adaptación inadecuada a la oscuridad, causan pérdida de visión periférica y finalmente pérdida progresiva de visión.
Loci implicados: mutaciones producidas en más de 35 genes diferentes dan lugar a retinitis pigmentosa no sindrómica, que es clínicamente indistinguible. A esto se debe que no sea posible deducir qué gen es el alterado basándose sólo en el examen ocular.
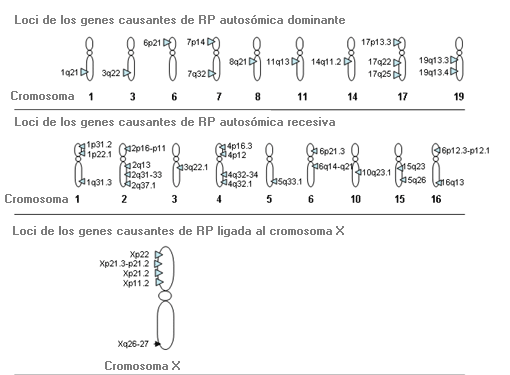
Heterogeneidad alélica: Un solo fenotipo originado por diferentes mutaciones en el mismo gen y locus cromosómico. (Nota: algunos trastornos muestran heterogeneidad genética, otros heterogeneidad alélica y otros ambas características).
Expresividad variable: variación de las manifestaciones clínicas (tipo y severidad) de una enfermedad genética entre individuos que presentan la misma alteración genética, incluso dentro de la misma familia.
(Nota: La heterogeneidad genética implica que los individuos con diferentes genotipos tienen el mismo fenotipo, mientras que la expresividad variable implica que los individuos con el mismo genotipo presenten un fenotipo que muestra diferencias tanto en la severidad como en la edad de comienzo de sus manifestaciones).
I
Inestabilidad de microsatélites - Microsatellite instability (sinónimos : MSI, fenotipo de error de replicación, RER)
Discrepancia entre el tamaño de los microsatélites en el adn de tejido tumoral comparado con el no tumoral de la misma persona, a consecuencia de las mutaciones producidas en un gen en la vía de reparación de los errores de emparejamiento de bases del adn (MMR). Estos gen es si no estuviesen mutados corregirían normalmente estos errores.
J
K
L
M
Mecanismo de reparación de bases mal emparejadas - Mismatch repair mechanism (sinónimo: reparación de emparejamientos erróneos)
Sistema de control de la integridad del ADN, en el que intervienen determinados genes, que identifica, escinde y corrige los errores de emparejamiento de las bases durante la replicación del ADN. Las mutaciones producidas en los genes responsables de este mecanismo pueden dar lugar a determinadas enfermedades genéticas y algunos tipos de cáncer.
Microsatélite - Microsatellite (sinónimos: ADN satélite, repeticiones cortas en tándem)
Segmentos repetitivos de ADN que comprenden de dos a cinco nucleótidos (repeticiones de dinucleótidos/trinucleótidos/ tetranucleótidos/ pentanucleótidos) dispersos por todo el genoma en las regiones no codificadoras que hay entre o dentro de los genes (intrones), se utilizan frecuentemente como marcadores en el análisis de ligamiento debido a la alta variabilidad natural existente en el número de repeticiones entre los individuos. Estas regiones son propiamente inestables y susceptibles a mutaciones.
Mutación - Mutation (sinónimos: alteración de la secuencia, mutación del splicing)
Cualquier alteración producida en un gen con respecto a su estado natural; puede ser patológica o una variante no patológica.
1. Mutaciones patológicas: aquellas que dan lugar a una función anormal del gen y a un fenotipo de enfermedad. Los tipos de mutaciones patológicas son los siguientes:


Adaptado de Thompson & Thompson Genetics in Medicine, 6th Edition; RL Nussbaum, RR McInnes, HF Willard, Genetic Variation in Individuals: Mutation and Polymorphism,pág 81, Copyright 2001, con permiso de Elsevier
2. Polimorfismos (variantes normales): Mutaciones que no tienen efectos adversos sobre la función del gen y no dan lugar a un fenotipo alterado (enfermedad).
Mutación de línea germinal - Germline mutation
Presencia de un gen alterado en el óvulo o espermatozoide (célula germinal) que puede transmitirse a las generaciones siguientes.
En un individuo, la mutación puede darse en una de cuatro situaciones posibles:
1.- Exclusivamente en una sola célula germinal (óvulo o espermatozoide).
2.- Exclusivamente en las células somáticas (células del organismo, pero NO en la línea germinal); son las normalmente asociadas al desarollo de un tumor.
3.- En algunas células germinales y en otras somáticas.
4.- En todas las células del organismo.


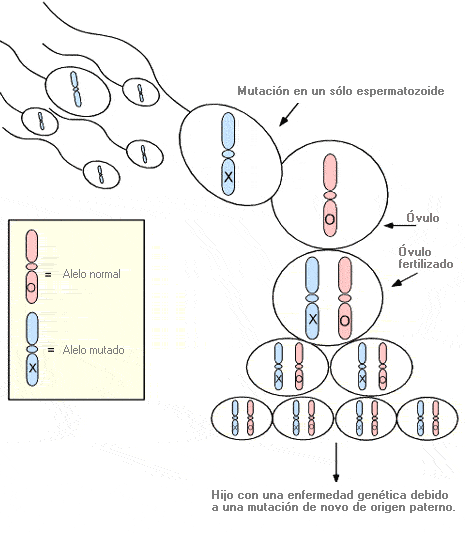
Mutación de novo - De novo mutation (sinónimos: mutación génica de novo, mutación génica nueva, mutación nueva)
Mutación de sentido erróneo - Missense mutation
Sustitución de un solo par de bases que da lugar a la traducción de un aminoácido diferente en esa posición.
Mutación dominante negativa - Dominant negative mutation
Mutación cuyo producto génico afecta de forma adversa al producto génico normal de tipo salvaje de la misma célula, normalmente al dimerizarse (combinarse) con él. En las moléculas poliméricas, tales como el colágeno, las mutaciones dominantes negativas suelen ser más deletéreas que las que no originan ningún producto génico (mutaciones nulas o alelos nulos).
Mutación en el sitio de corte y empalme (splicing) - Splicing
Mutación que altera o suprime la secuencia específica que indica el sitio en el que tiene lugar el splicing de un intrón. A consecuencia de estas mutaciones, uno o más intrones permanecen en el ARN mensajero maduro y pueden alterar la generación del producto proteico.
Mutación familiar específica - Familial mutation
En una familia, la alteración observada en una secuencia que causa una determinada enfermedad o predispone a ella; la mutación puede ser rara o común.
Mutación intrónica - Intronic mutation
Mutación (normalmente una sustitución de bases) localizada en un intrón que genera un sitio de corte y empalme (splicing) alternativo que compite con los sitios de splicing normales durante el procesamiento del ARN. Esta mutación da lugar a la formación de ARN mensajero maduro con secuencias intrónicas.
Mutación no descrita - Novel mutation
Alteración nueva de un gen, que no ha sido previamente descrita. No debe confundirse con mutación “nueva” o “de novo”.
Mutación particular - Private mutation (sinónimo: mutación única)
Tipo de mutación patológica rara que se observa sólo en unas pocas familias.
Mutación patológica - Disease
Alteración en un gen que causa o predispone a un individuo a desarrollar una enfermedad específica.
Mutación por cambio de marco - Frameshift mutation (sinónimos: deleción fuera de marco, mutación fuera de marco)
Inserción o deleción de una secuencia de pares de bases que no es múltiplo de tres y por lo tanto, altera el marco de lectura del triplete, lo que da lugar normalmente a la creación de un codón de terminación (parada) prematuro y cuyo resultado es un producto proteico truncado.
Mutación predisponente - Predisposing mutation (sinónimo: gen de susceptibilidad)
Mutación génica que incrementa la susceptibilidad o predisposición de un individuo a desarrollar una determinada enfermedad. Cuando se heredan mutaciones patogénicas en estos genes de susceptibilidad, es probable, pero no seguro, que el individuo desarrolle síntomas de la enfermedad.
Mutación puntual - Point mutation
Alteración en la secuencia del ADN causada por el cambio, inserción o deleción de un único nucleótido.
Mutación sin cambio de pauta de lectura - In-frame mutation
Mutación que no produce un cambio en el marco de lectura del triplete cuando se produce la transcripción de ADN a ARN mensajero. Estas mutaciones, sin embargo, pueden dar lugar a la síntesis de un producto proteico anormal si, al traducirse el ARN a proteína, el cambio de triplete supone un cambio de aminoácido.
Mutación sin sentido - Nonsense mutation
Mutación que altera un codón convirtiéndolo en uno de terminación (stop codon) prematura del proceso de traducción.
N
Ñ
O
P
PCR - PCR (Polymerase chain reaction) (sinónimo: reacción en cadena de la polimerasa)
Procedimiento que genera millones de copias de un segmento corto de ADN mediante ciclos repetidos de: 1) desnaturalización del ADN, 2) acoplamiento de los oligonucleótidos y 3) extensión mediante la acción de la ADN polimerasa. La PCR es un procedimiento muy común en los estudios de genética molecular y se puede utilizar para: 1) generar una cantidad suficiente de ADN para realizar determinadas pruebas (p. ej. análisis de la secuencia, rastreo de mutaciones); o 2) puede ser una prueba diagnóstica en sí misma (por ejemplo amplificación específica del alelo, cuantificación del número de repeticiones de trinucleótidos).
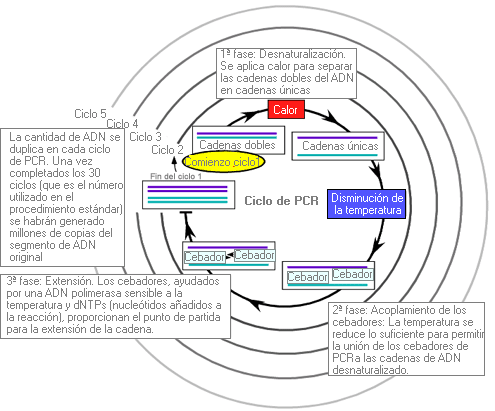
PCR cuantitativa - Quantitative PCR (sinónimos: PCR cuantitativa en tiempo real)
Uso de PCR para determinar la cantidad de ADN o ARN en una muestra; se utiliza frecuentemente para detectar mutaciones por deleción y mutaciones por duplicación.
Anteriormente, la PCR cuantitativa determinaba la cantidad de producto (ADN o ARN) tras completar un número específico de ciclos; sin embargo, este procedimiento no es del todo fiable o preciso y se ha sustituido en gran parte por la PCR cuantitativa “en tiempo real” (denominada también “cinética”), en la que se determina la cantidad del producto (ADN o ARN) tras completar cada ciclo de PCR.
PCR cuantitativa en tiempo real
I. Se amplifica la región de ADN o ARN de interés en presencia de una sonda fluorescente; la intensidad de la señal fluorescente generada aumenta en proporción a la cantidad de producto resultante en PCR.
II. La intensidad de la señal fluorescente, que se mide al completar cada ciclo de PCR, se representa en una “gráfica de amplificación” para determinar el CT (del inglés cycle threshold), que se define como el punto en el que se detecta por primera vez la señal fluorescente, es decir, el producto de la PCR.
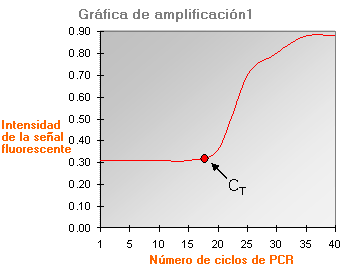
III. Se compara el valor CT frente a una curva estándar (basándose en los valores CT de muestras control y sus respectivas cantidades iniciales de ADN o ARN conocidas) para determinar la cantidad de ADN o ARN en la muestra del paciente. La presencia de una mutación por deleción o duplicación se puede deducir comparando la cantidad de ADN o ARN en la muestra del paciente con la cantidad en una muestra control. Por ejemplo, si la cantidad de ADN o ARN en la muestra del paciente fuese ~50% respecto a la del control normal, esto indicaría una mutación por deleción; si la cantidad de ADN o ARN en la muestra fuese ~150% respecto a la del control normal, indicaría una mutación por duplicación.
1Adaptado de PennState College of Medicine, Quantitative Real-Time PCR
Algunas implicaciones clínicas
Al igual que la PCR, la PCR cuantitativa se ha convertido en una técnica estándar en todos los laboratorios clínicos de genética molecular.
Los análisis para detectar mutaciones por deleción hemicigótica (p. ej. deleciones en DMD en varones afectados por distrofia muscular de Duchenne) se pueden realizar con técnicas de PCR estándar. Sin embargo, se debe realizar PCR cuantitativa, en lugar de estándar, para detectar mutaciones por deleción heterocigótica (p. ej. deleciones heterocigóticas en (1) individuos afectados por un trastorno causado por una deleción en un gen contiguo autosómico, como, por ejemplo, el síndrome de Williams, (2) portadores de un trastorno autosómico recesivo, como la atrofia muscular espinal y (3) mujeres portadoras de un trastorno ligado al cromosoma X, por ejemplo la distrofia muscular de Duchenne).
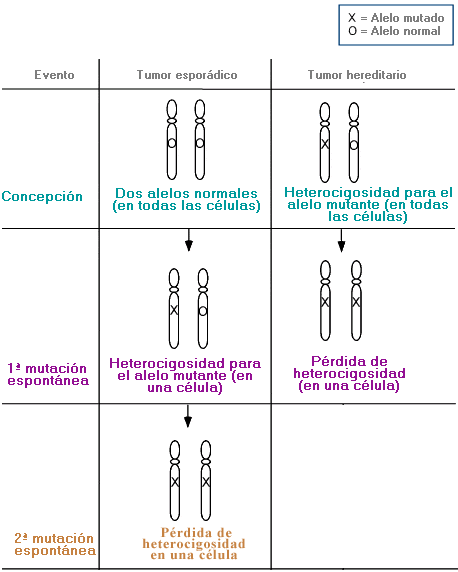
Pérdida de heterocigosidad - Loss of heterozygosity (LOH, de sus siglas en inglés)
Cuando en un locus determinado, que es heterocigoto para un alelo mutante deletéreo y un alelo normal, se produce una deleción, u otro evento mutacional, en el alelo normal que convierte a la célula bien en hemicigótica (es decir, con un único alelo deletéreo), bien en homocigótica para el alelo deletéreo.
La LOH es el mecanismo en el que se basa el desarrollo de algunos tipos de cáncer en los que una sola mutación en uno de los homólogos de un gen supresor tumoral no es suficiente para iniciar el crecimiento del tumor. Sin embargo, la deleción o inactivación del segundo alelo en el cromosoma homólogo, da lugar a un crecimiento celular incontrolado.
La LOH puede ser el fundamento del desarrollo del cáncer en tumores esporádicos y hereditarios. En los tumores esporádicos, los dos alelos son normales en el momento de la concepción, mientras que en los tumores hereditarios hay un alelo mutante en el momento de la concepción.
Poligénico - Polygenic
Rasgo o patología causado por las contribuciones aditivas de mutaciones producidas en múltiples genes en diferentes loci.
Polimorfismos de conformación de cadenas sencillas - Single-stranded conformational polymorphism (SSCP)
Técnica para el rastreo de mutaciones que identifica segmentos de ADN de cadena sencilla que muestran un patrón de migración anormal en la electroforesis en gel.
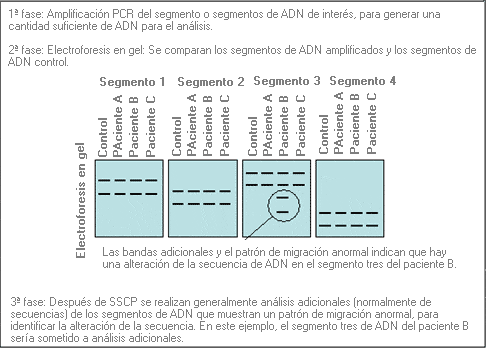
Algunas implicaciones clínicas
La técnica SSCP se puede utilizar cuando las mutaciones están distribuidas por todo el gen.
El SSCP puede comprender el análisis de todo el gen o sólo de regiones específicas.
Predisposición genética - Genetic predisposition (sinónimo: susceptibilidad genética)
Mayor propensión de un individuo a padecer una enfermedad determinada debido a la presencia de una o más mutaciones génicas asociadas con un aumento del riesgo de que se manifieste la enfermedad y/o debido a la existencia de antecedentes familiares que también indican una mayor probabilidad de que se desarrolle la enfermedad.
Pseudogen - Pseudogene
Copia de un gen que carece normalmente de intrones y de otras secuencias de ADN esenciales para su función. Los pseudogenes, aunque son genéticamente similares al gen funcional original, no se expresan y frecuentemente contienen numerosas mutaciones.
Pseudogenes:
Consisten en secuencias de ADN muy similares a los genes conocidos, pero no son funcionales.
Se encuentran dispersos en el genoma.
Se cree que son subproductos de la evolución, que representan genes rudimentarios que una vez fueron funcionales.
Se supone que han perdido su función original por la introducción de mutaciones en las secuencias codificadoras o reguladoras.
 Supuestos mecanismos por los que se forman los pseudogenes:
Supuestos mecanismos por los que se forman los pseudogenes:
Duplicación de un gen funcional seguida de la introducción de numerosas mutaciones que finalmente dan lugar a su inactivación.
Retrotransposición – proceso mediante el cual se generan una o más copias de ADN a partir del transcripto de ARNm del gen y se vuelven a insertar en el genoma en cualquier localización, no necesariamente cerca del sitio de su gen progenitor. Puesto que se generan a partir de ARNm procesado, estos pseudogenes carecen de intrones.
Puntos calientes de mutación - Hotspot mutation region
Secuencias de adn muy susceptibles a mutaciones debido a una inestabilidad inherente, tendencia hacia un entrecruzamiento desigual o predisposición química a sustituciones de nucleótidos simples; región en la que se observan mutaciones con más frecuencia de la habitual.
Q
R
Región crítica - Critical region
Parte específica de un cromosoma o de un gen que cuando experimenta alguna alteración (deleción, duplicación u otras mutaciones) produce el conjunto característico de anomalías fenotípicas asociadas con un síndrome o enfermedad determinada.
Región de puntos calientes de mutación - Hotspot mutation region
Secuencias de ADN muy susceptibles de ser mutadas debido a una inestabilidad inherente, tendencia hacia un entrecruzamiento desigual o predisposición química a sustituciones de nucleótidos simples; región en la que se observan mutaciones con más frecuencia de la habitual.
Riesgo hereditario - Background risk (sinónimo: riesgo poblacional)
Proporción de individuos en una población determinada que están afectados por una patología específica o que presentan mutaciones en un gen determinado; en el proceso de asesoramiento genético esto se determina generalmente mediante una comparación con el riesgo personal del paciente, dados sus antecedentes familiares u otras circunstancias.

S
T
Tasa de mutación - Mutation rate
La tasa de mutación de un gen o una secuencia de ADN es la frecuencia en la que se producen nuevas mutaciones en ese gen o la secuencia en cada generación.
Transposon (Tn) -
Son regiones de ADN (200-2000 pares de bases) capaces de moverse de manera autosuficiente de una parte a otra parte del genoma, mediante un mecanismo de corta-pega y cuya consecuencia es la producción de mutaciones.
U
V
Variabilidad genética - Genetic variability
Se denomina variabilidad genética a los cambios genéticos que aparecen en los genes de una especie o de una población. La variabilidad genética permite la evolución de las especies, ya que en cada generación solamente una fracción de la población sobrevive y se reproduce transmitiendo características particulares a su progenie. De esta manera, a mayor variabilidad genética, mayor velocidad en el desarrollo de los cambios evolutivos. La variabilidad genética se origina por mutaciones y recombinaciones genéticas. Los procesos que dirigen o eliminan variabilidad genética son la selección natural y la deriva genética.